Analysis of changes in the genome of Vibrio cholerae O1 El Tor genovariants during the current period of the cholera pandemic
- Authors: Smirnova N.I.1, Rybalchenko D.A.1, Lozovskiy Y.V.1, Krasnov Y.M.1, Kutyrev V.V.1
-
Affiliations:
- Research Anti-Plague Institute “Microbe”
- Issue: Vol 100, No 5 (2023)
- Pages: 346-357
- Section: ORIGINAL RESEARCHES
- URL: https://microbiol.crie.ru/jour/article/view/18482
- DOI: https://doi.org/10.36233/0372-9311-389
- EDN: https://elibrary.ru/tuekup
- ID: 18482
Cite item
Full Text
Abstract
Introduction. The genome variability of genetic variants of El Tor cholera agent has led to the emergence of strains carrying mutations in various genes associated with epidemically important pathogen properties. This situation requires an assessment of the trends in these changes in order to predict the pathogenic potential of previously unknown variants and promptly develop new tools for their diagnostics and prevention.
The purpose of this work was to analyze the dynamic changes in pathogenicity and drug resistance genes of V. cholerae El Tor genetic variants from endemic countries and Russia.
Materials and methods. We analyzed complete genome nucleotide sequences of 104 V. cholerae El Tor strains from the NCBI Gen Bank and European Nucleotide Archive databases, as well as those obtained by us. The nucleotide sequences were analyzed using the UGEN v. 45.1 software. The dendrogram was constructed using maximum parsimony algorithm in BioNumerics v.7.6 software package based on the multiple alignment generated using the Snippy 4.6.0 program.
Results. Genomic sequences of 103 strain genovariants isolated on the territory of nine endemic countries of Asia and Africa, as well as in Russia in 1991-2022, have been compared. It is shown that the process of genovariant genome changing was multistage and occurred due to the continuous accumulation of point mutations in key (ctxB and tcpA) and additional (rtxA) genes of pathogenicity and core genes of antibiotic resistance (gyrA, parC and carR), as well as a deletion in SXT element. The most important was the change in the ctxB gene and the emergence of new genovariants with the ctxB7 allele, which replaced the previously prevalent strains. Analysis of altered genome regions of 83 strains from endemic regions has revealed eight genotypes, while the strains (21 isolates) imported to Russia belonged to only five of them including highly virulent strains with the ctxB7 allele and lost PolR biovar-specific feature due to carR gene mutation. The established close phylogenetic relatedness of genovariants from Russia with strains from endemic Asian countries confirms their importation from this region.
Conclusion. The sequential occurrence and accumulation of mutations in the pathogenicity and drug resistance genes in the genome of genovariants in endemic regions have been shown, which leads to a change in their epidemically important features. The importation of new highly virulent genovariants into Russia has been established, which indicates the need for an ongoing assessment of changes in the genome of this pathogen for the timely development of adequate means of gene diagnostics and prevention.
Full Text
Введение
Семь известных пандемий холеры были вызваны токсигенными штаммами Vibrio cholerae, относящимися к серогруппе О1 и двум биоварам — классическому и El Tor [1]. Среди них особый интерес представляют штаммы V. cholerae El Tor, вызвавшие текущую, 7-ю пандемию холеры, продолжающуюся более 60 лет (с 1961 г.). Постоянные эпидемии и вспышки холеры El Tor в эндемичных странах Азии, Африки и Америки обусловливают риск завоза этой инфекции на территорию России, не являющейся эндемичной по холере [2].
Установлено, что глобальное распространение холеры из первичного очага (Бенгальского залива) в другие страны мира происходило тремя независимыми, но перекрывающимися волнами [3]. Молекулярно-генетический анализ различных штаммов показал, что на протяжении 3 волн в геноме возбудителя происходили значительные изменения. Эпидемии холеры в 1961–1991 гг. (1-я волна) вызывали токсигенные штаммы с типичными для биовара El Tor свойствами, содержащие в составе профага СТХEltφ гены ctxAB3, кодирующие, соответственно, A- и B-субьединицы холерного токсина — ключевого фактора патогенности [4]. Однако в 1991–1994 гг. (начало 2-й волны) в Бангладеш впервые были обнаружены генетически изменённые штаммы, несущие ген ctxB1 V. cholerae классического биовара — возбудителя предыдущих 6 пандемий азиатской холеры. Эти геноварианты возникли, видимо, в результате горизонтального переноса профага СТХφ классического типа (СТХclassφ) с генами ctxAB1 от V. cholerae классического биовара типичным штаммам V. cholerae El Tor [5]. Вместе с тем геном геновариантов оказался нестабильным, и в последние два десятилетия в эндемичных регионах появились штаммы с новыми мутациями в мобильных генетических элементах, связанных с патогенностью (остров патогенности VPI-1, профаг CTXφ) или способностью к эпидемическому распространению (остров пандемичности VSP-II) [6, 7]. Возникла мутация в гене tcpA из острова патогенности VPI-1, кодирующем основной белок токсин-корегулируемых пилей или TCP — ключевого фактора колонизации. Новый аллель гена tcpA обозначили как tcpACIRS101 [8] или tcpACIRS. Затем появились штаммы с новым аллелем гена ctxB — ctxB7 [6]. Важным следствием таких мутаций стало усиление вирулентности возбудителя за счёт повышенной продукции холерного токсина [9, 10]. Кроме того, обнаружили вариабельность устойчивости к лекарственным препаратам, связанную с присутствием в геноме разных типов интегративного конъюгативного элемента (integrative conjugating element, ICE) SXT, несущих различный набор генов резистентности к антибиотикам [11]. Более того, у геновариантов появились мутации в коровой области хромосомы. Так, штаммы из 3-й волны в гене rtxA, кодирующем многофункциональный цитотоксин MARTX (multifunctional autoprocessing repeats-in toxin), имели null-мутацию (аллель rtxA4), которая привела к образованию стоп-кодона и утрате биосинтеза этого токсина [12]. Выявлены также 2 точечные мутации в генах gyrA(G248T) и parC(C254T), кодирующих топоизомеразы, которые обусловили формирование у патогена резистентности к налидиксовой кислоте [13]. Более того, недавно стало известно о возникновении геновавариантов с мутацией в регуляторном гене carR(G265A), ставшей причиной утраты их устойчивости к полимиксину B — одному из фенотипических маркеров вибрионов El Tor [14].
Появление и широкое распространение различных генетических вариантов возбудителя в эндемичных очагах холеры привело к их завозу на территорию России в период 2-й и 3-й волн пандемии. Изучением геновариантов занимались многие исследователи [15–18]. Особое внимание было уделено установлению филогенетических связей штаммов из разных эндемичных и неэндемичных регионов [19–23]. Однако многие вопросы о динамике изменений генома геновариантов в течение длительного периода изучены недостаточно. Так, не проведён сравнительный анализ изменчивости генотипов штаммов геновариантов, выделенных в эндемичных регионах и на территории России в течение длительного периода. Между тем поиск генетических особенностей штаммов, выделенных на различных территориях и в разные временны΄е периоды, представляет значительный интерес для понимания направления изменения вирулентных свойств возбудителя. Более того, сведения о вариабельности генов патогенности и лекарственной устойчивости новых геновариантов в случае их завоза в Россию необходимы для своевременной разработки адекватных генодиагностических и профилактических средств. Всё это определяет актуальность работы.
Цель работы — анализ динамики изменения генов патогенности и лекарственной устойчивости генетических вариантов V. choleraе El Tor из эндемичных стран и России.
Решение поставленных вопросов было основано на анализе сиквенсов полных геномов различных штаммов геновариантов, циркулирующих в последние годы в разных эндемичных регионах и завезённых в Россию. Выбранный подход позволяет получить более полную и достоверную информацию по сравнению с другими методами об изменении указанных генов, расположенных в разных участках хромосомы.
Материалы и методы
В работе использовали секвенированные нуклеотидные последовательности полных геномов 86 штаммов геновариантов V. cholerae El Tor (1991–2022 гг.), взятых из баз данных NCBI GenBank и European Nucleotide Archive, а также полученные нами сиквенсы 14 штаммов геновариантов, завезённых в Россию в 1993–2014 гг., и 4 штаммов, выделенных во время вспышки холеры в 2011 г. на Украине [24] (таблица).
Штаммы V. cholerae биовара El Tor, полногеномные нуклеотидные последовательности которых использованы в работе
Strains of Vibrio cholerae biovar El Tor, the whole genome nucleotide sequences of which were used in the study
№ | No. | Место и год выделения The site and year of isolation | Аллель генов Allele of genes | Структура генов Genes structure | Номер доступа в Gen Bank/ENA Gen Bank/ENA access number | ||||
ctxB | tcpA | rtxA | gyrA | parC | carR | |||
Штаммы из эндемичных стран Азии и Африки | Strains from endemic Asian and African countries | ||||||||
N16961 | Бангладеш, 1975 | Bangladesh, 1975 | ctxB3 | tcpAElt | rtxA1 | int. | int. | int. | AE003852, AE003853 |
V212-1 | Индия, 1991 | India, 1992 | ctxB1 | tcpAElt | rtxA1 | int. | int. | int. | ERR018125 |
VC51 | Индия, 1992 | India, 1992 | ctxB1 | tcpAElt | rtxA1 | int. | int. | int. | ERR018124 |
MJ-1236 | Бангладеш, 1994 | Bangladesh, 1994 | ctxB1 | tcpAElt | rtxA1 | int. | int. | int. | CP001485 |
CIRS101 | Бангладеш, 2002 | Bangladesh, 2002 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | int. | ACVW01000000 |
4519 | Индия, 2005 | India, 2005 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | ERR025374 |
4656 | Индия, 2006 | India, 2006 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | int. | ERS013258 |
4488 | Индия, 2006 | India, 2006 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | int. | ERS013234 |
4551 | Индия, 2007 | India, 2007 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | ERR025357 |
4623 | Индия, 2007 | India, 2007 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | ERS013267 |
4646 | Индия, 2007 | India, 2007 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | ERS013237 |
VN243P07 | Вьетнам, 2007 | Vietnam, 2007 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | |
IND031 | Индия, 2009 | India, 2009 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX062980 |
IND033 | Индия, 2009 | India, 2009 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX062982 |
IND041 | Индия, 2009 | India, 2009 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX062990 |
IND048 | Индия, 2009 | India, 2009 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX062997 |
VC-6 | Непал, 2010 | Nepal, 2010 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | SRR308703 |
VC-14 | Непал, 2010 | Nepal, 2010 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | SRR308715 |
VC-19 | Непал, 2010 | Nepal, 2010 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | SRR308722 |
VC-16 | Непал, 2010 | Nepal, 2010 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | SRR308717 |
VC-8 | Непал, 2010 | Nepal, 2010 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | SRR308705 |
BGD043 | Бангладеш, 2010 | Bangladesh, 2010 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX179812 |
BGD113 | Бангладеш, 2010 | Bangladesh, 2010 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX179747 |
NHCM-053 | Бангладеш, 2011 | Bangladesh, 2011 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | GCA001187105 |
BGD117 | Бангладеш, 2011 | Bangladesh, 2011 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | DRX179750 |
BGD056 | Бангладеш, 2011 | Bangladesh, 2011 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX179776 |
IND051 | Индия, 2011 | India, 2011 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | DRX063000 |
IDH-04808 | Индия, 2012 | India, 2012 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX063018 |
BGD060 | Бангладеш, 2012 | Bangladesh, 2012 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX179773 |
BGD065 | Бангладеш, 2012 | Bangladesh, 2012 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX179774 |
BGD067 | Бангладеш, 2012 | Bangladesh, 2012 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX179777 |
BGD119 | Бангладеш, 2012 | Bangladesh, 2012 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | DRX179752 |
BGD120 | Бангладеш, 2012 | Bangladesh, 2012 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | DRX179753 |
BGD122 | Бангладеш, 2013 | Bangladesh, 2013 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | DRX179755 |
BGD082 | Бангладеш, 2013 | Bangladesh, 2013 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | int. | DRX179791 |
IND071 | Индия, 2013 | India, 2013 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | DRX063020 |
IDH-05298 | Индия, 2013 | India, 2013 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | DRX063023 |
IND079 | Индия, 2013 | India, 2013 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | DRX063028 |
IND082 | Индия, 2014 | India, 2014 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | DRX063031 |
IND090 | Индия, 2014 | India, 2014 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | DRX063039 |
BGD095 | Бангладеш, 2014 | Bangladesh, 2014 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | DRX179793 |
BGD089 | Бангладеш, 2014 | Bangladesh, 2014 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | DRX179789 |
BGD128 | Бангладеш, 2015 | Bangladesh, 2015 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | DRX179758 |
BGD132 | Бангладеш, 2015 | Bangladesh, 2015 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | DRX179760 |
Tanz 14 | Танзания, 2015 | Tanzania, 2015 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERS2318685 |
UG026 | Уганда, 2015 | Uganda, 2015 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SAMN08744332 |
4621STDY- 6714750 | Кения, 2015 | Kenya, 2015 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERS1572785 |
4621STDY- 6714749 | Кения, 2015 | Kenya, 2015 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERS1572784 |
UG020 | Уганда, 2016 | Uganda, 2016 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SAMN08744331 |
UG010 | Уганда, 2016 | Uganda, 2016 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SAMN08744330 |
4621STDY- 6714780 | Кения, 2016 | Kenya, 2016 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERS1572815 |
BGD143 | Бангладеш, 2016 | Bangladesh, 2016 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | DRX179768 |
BGD140 | Бангладеш, 2016 | Bangladesh, 2016 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | DRX179766 |
IND220 | Индия, 2016 | India, 2016 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | DRX179803 |
IND231 | Индия, 2016 | India, 2016 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | DRX179808 |
BGD137 | Бангладеш, 2016 | Bangladesh, 2016 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | DRX179763 |
CNRVC170168 | Йемен, 2016 | Yemen, 2016 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERR2265674 |
CNRVC170175 | Йемен, 2016 | Yemen, 2016 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERR2269613 |
NALMLE36 | Бангладеш, 2017 | Bangladesh, 2017 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | SIVL00000000 |
NALMLE34 | Бангладеш, 2017 | Bangladesh, 2017 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | SIVJ00000000 |
NALMLE37 | Бангладеш, 2017 | Bangladesh, 2017 | ctxB1 | tcpACIRS | rtxA1 | G248T | C254T | int. | SIVM00000000 |
THSTI_56695 | Индия, 2017 | India, 2017 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERR2270661 |
THSTI_56712 | Индия, 2017 | India, 2017 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERR2270662 |
CNRVC170179 | Йемен, 2017 | Yemen, 2017 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERR2269617 |
CNRVC170208 | Йемен, 2017 | Yemen, 2017 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERR2269811 |
CNRVC170197 | Йемен, 2017 | Yemen, 2017 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | ERR2269710 |
NALMLE05 | Бангладеш, 2018 | Bangladesh, 2018 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | int. | SIUG00000000 |
NALMLE01 | Бангладеш, 2018 | Bangladesh, 2018 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SIUC00000000 |
NALMLE03 | Бангладеш, 2018 | Bangladesh, 2018 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SIUE00000000 |
NALMLE31 | Бангладеш, 2018 | Bangladesh, 2018 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SIVG00000000 |
KDCP136 | Бангладеш, 2018 | Bangladesh, 2018 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SRR14297670 |
KDCP183 | Бангладеш, 2019 | Bangladesh, 2019 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | int. | SRR14297546 |
CMR50 | Камерун, 2019 | Cameroon, 2019 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SAMN16213389 |
CMR44 | Камерун, 2019 | Cameroon, 2019 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SAMN16213387 |
KDCP208 | Бангладеш, 2019 | Bangladesh, 2019 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SRR14297682 |
KDCP245 | Бангладеш, 2019 | Bangladesh, 2019 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SRR14297553 |
KDCP262 | Бангладеш, 2019 | Bangladesh, 2019 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SRR14297555 |
KMCP107 | Бангладеш, 2019 | Bangladesh, 2019 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SRR14297699 |
KMCP145 | Бангладеш, 2019 | Bangladesh, 2019 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SRR14297701 |
DMAVC-20 | Бангладеш, 2021 | Bangladesh, 2021 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SAMD00514837 |
DMAVC-1 | Бангладеш, 2022 | Bangladesh, 2022 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SAMD00514672 |
DMAVC-16 | Бангладеш, 2022 | Bangladesh, 2022 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SAMD00514833 |
DMAVC-19 | Бангладеш, 2022 | Bangladesh, 2022 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | SAMD00514836 |
Штаммы из Российской Федерации | Strains isolated in the Russian Federation | ||||||||
*М-1275 | Россия, Каспийск, 1993 Russia, Kaspiysk, 1993 | ctxB1 | tcpAElt | rtxA1 | int. | int. | int. | LRAF00000000 |
*М-1270 | Россия, Набережные Челны, 1993 Russia, Naberezhnye Chelny, 1993 | ctxB1 | tcpAElt | rtxA1 | int. | int. | int. | VXCC0000000 |
*М-1293 | Россия, Республика Дагестан, 1994 Russia, Dagestan Republic, 1994 | ctxB1 | tcpAElt | rtxA1 | int. | int. | int. | JFFW00000000 |
I-1181 | Россия, Новосибирская область, 1994 Russia, Novosibirsk region, 1994 | ctxB1 | tcpAElt | rtxA1 | int. | int. | int. | UCN00000000 |
I-1263 | Россия, Иркутск, 1997 Russia, Irkutsk, 1997 | ctxB1 | tcpAElt | rtxA1 | G248T | int. | int. | JPLT00000000 |
*Р-17644 | Россия, Ачинск, 1997 Russia, Achinsk, 1997 | ctxB1 | tcpAElt | rtxA1 | G248T | int. | int. | JRTW00000000 |
*М-1327 | Россия, Дагестан, 1998 Russia, Dagestan, 1998 | ctxB1 | tcpAElt | rtxA1 | int. | int. | int. | LRFE00000000 |
I-1300 | Россия, Южно-Сахалинск, 1999 Russia, Yuzhno-Sakhalinsk, 1999 | ctxB1 | tcpAElt | rtxA1 | int. | int. | int. | JZCC00000000 |
*М-1344 | Россия, Казань, 2001 Russia, Kazan, 2001 | ctxB1 | tcpAElt | rtxA1 | G248T | int. | int. | NEDY00000000 |
*М-1429 | Россия, Белорецк, 2004 Russia, Beloretsk, 2004 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | int. | LAEM00000000 |
*М1430 | Россия, Тверь, 2005 | Russia, Tver, 2005 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | int. | AYOM00000000 |
*Р-18899 | Россия, Мурманск, 2006 Russia, Murmansk, 2006 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | int. | LAKM00000000 |
*81 | Россия, Ростов-на-Дону, 2014 Russia, Rostov-on-Don, 2014 | ctxB1 | tcpACIRS | rtxA4 | G248T | C254T | int. | JRQM00000000 |
*L-3226 | Россия, Москва, 2010 | Russia, Moscow, 2010 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | JDVX00000000 |
*L-4150 | Россия, Москва, 2010 | Russia, Moscow, 2010 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | JNGT00000000 |
*76 | Украина, Мариуполь, 2011 Ukraine, Mariupol, 2011 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | MPVL00000000 |
*153 | Украина, Мариуполь, 2011 Ukraine, Mariupol, 2011 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | MWRE00000000 |
*39 | Украина, Мариуполь, 2011 Ukraine, Mariupol, 2011 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | MWRC00000000 |
*186 | Украина, Мариуполь, 2011 Ukraine, Mariupol, 2011 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | int. | PYBQ00000000 |
*M1509 | Россия, Москва, 2012 | Russia, Moscow, 2012 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | AYNL00000000 |
*3265/80 | Россия, Москва, 2014 | Russia, Moscow, 2014 | ctxB7 | tcpACIRS | rtxA4 | G248T | C254T | G265A | JRQL00000000 |
Примечание. *Штаммы, нуклеотидные последовательности геномов которых секвенированы нами; int. — интактный.
Note. *Nucleotide sequences of strains sequenced by us; int. — intact.
Для проведения исследований штаммы получали из Государственной коллекции патогенных бактерий РосНИПЧИ «Микроб», где они хранились в лиофилизированном состоянии. Бактерии культивировали в бульоне и агаре LB при 37ºС.
Чувствительность 18 штаммов к 2 антибиотикам определяли общепринятым диско-диффузионным методом с использованием полимиксина B (50 мкг) и налидиксовой кислоты (30 мкг) фирм «HiMedia Laboratories Pvt. Ltd» и «Oxoid».
Для проведения секенирования выполняли выделение и очистку геномной ДНК из бактериальной суспензии с использованием набора «Axy Prep Bacterial Genomic DNA Miniprep Kit» («Axygen»). Клетки предварительно обрабатывали мертиолятом натрия до конечной концентрации 1 : 10 000 (0,01%) и прогревали при 56ºC в течение 30 мин. Полные геномы холерных вибрионов секвенировали на генетическом анализаторе «Ion PGM» («Thermo Fisher Scientific») с использованием стандартных протоколов подготовки проб и программного обеспечения. Для картирования и поиска единичных нуклеотидных полиморфизмов (single nucleotide polymorphism, SNP) в полученных прочтениях использовали программный пакет «Snippy v. 4.6.0». Прочтения картировались на геном референсного штамма V. сholerae N16961, и затем формировалось множественное выравнивание, содержащее SNP в областях, присутствующих во всех исследуемых геномах. Для построения дендрограммы по алгоритму максимальной экономии применяли программный пакет «BioNumerics v. 7.6».
Результаты
Динамика изменения генов патогенности и лекарственной устойчивости геновариантов V. cholerae El Tor, циркулирующих в Азии и Африке
Для анализа измененений генов патогенности и лекарственной устойчивости мы провели биоинформационный анализ участков генома, содержащих ключевые (ctxB и tcpA) и дополнительные (rtxA) гены патогенности, а также гены резистентности к антибиотикам, локализованные в SXT-элементе и коровой области хромосомы (гены gyrA, parC и carR), 83 сиквенсов клинических штаммов V. cholerae El Tor из эндемичных регионов, взятых из международных баз данных. Проведённый анализ штаммов, изолированных на территории Индии, Бангладеш, Непала, Йемена, Вьетнама, Танзании, Кении, Уганды и Камеруна в 1991–2022 гг., позволил выявить заметные различия в их структуре у изолятов, выделенных в разные периоды. Сравнение структуры этих генов у различных вариантов говорит о том, что процесс их изменения происходил на протяжении относительно короткого периода времени и был многоступенчатым.
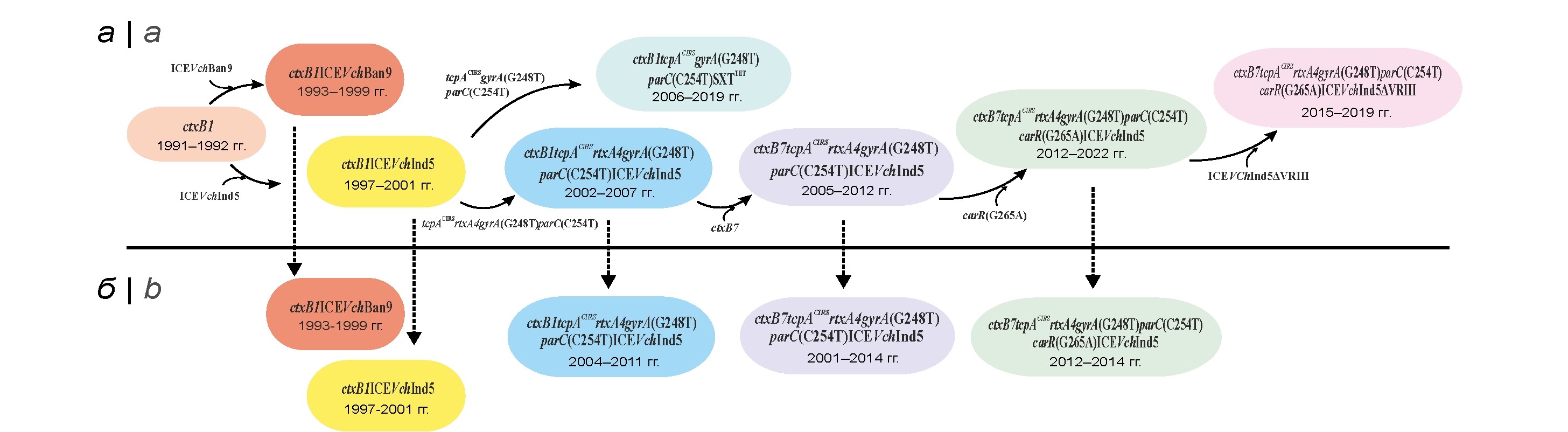
Из анализа полученных нами данных динамика изменения изучаемых свойств генетических вариантов возбудителя холеры El Tor представляется следующей (рис. 1, а). Первый этап — приобретение исходными геновариантами (ctxB1) различных типов SXT-элемента через горизонтальный перенос генов (1993–2001 гг.). Это событие привело к формированию штаммов 2 генотипов, различающихся набором генов резистентности к антибиотикам, — ctxB1ICEVchBan9, содержащих 7 генов (floR, strAB, sul2, dfrA1 и tetAR), кодирующих резистентность к хлорамфениколу, стрептомицину, сульфаниламиду, триметоприму и тетрациклину соответственно, и ctxB1ICEVcInd5, имеющих лишь 5 генов (floR, strAB, sul2 и dfrA1) при отсутствии генов tetAR. Все последующие этапы были связаны с мутациями генов патогенности и коровых генов резистентности к антибиотикам.
Одно из важнейших событий на следующем этапе микроэволюции — появление геновариантов с новыми аллелями ключевых (tcpACIRS) и дополнительных (rtxA4) генов патогенности, возникших за счёт несинонимичных однонуклеотидных замен в генах tcpA(A266G) и rtxA1(G13602A). Более того, одновременно возникли две точечные мутации в генах gyrA(G248T) и parC(C254T), кодирующих топоизомеразу II (ДНК-гиразу) и топоизомеразу IV, что обеспечило формирование у патогена резистентности к налидиксовой кислоте. Такие геноварианты c генотипом ctxB1tcpACIRSrtxA4gyrA(G248T)parC(С254T)ICEVchInd5, впервые обнаруженные в 2002 г. в Индии, были повсеместно распространены в эндемичных регионах Юго-Восточной Азии в 2002–2007 гг. (рис. 1, а). Тем не менее, наряду с ними, в эндемичных регионах Азии продолжали циркулировать штаммы с интактными генами ctxB1 и rtxA1, но с мутациями в генах tcpA, gyrA, parC. Среди них в Индии (2006–2010 гг.) и Бангладеш (2009–2019 гг.) преобладали штаммы с новым типом SXT — SXTTET, в составе которого были гены tetAR, strAB, sul2 и dfrA1, но, в отличие от других SXT, отсутствовал ген floR. Генотип таких штаммов был обозначен как ctxB1tcpACIRSrtxA1gyrA(G248T)parC(С254T)SXTTET. Вместе с тем распространения таких геновариантов на других территориях не отмечено. Самым существенным изменением генома возбудителя, повлекшим за собой стремительное распространение современных геновариантов, стала мутация в другом ключевом гене патогенности — ctxB1. Однонуклеотидная замена цитозина (С) на аденин (А) в позиции 58 привела к возникновению нового аллеля ctxB7 и появлению ранее неизвестных геновариантов с мутациями в указанных генах патогенности и устойчивости к антибиотикам (генотип ctxB7tcpACIRSrtxA4gyrA(G248T)parC(С254T)ICEVchInd5). Тем не менее их геном продолжал изменяться.
Значимое изменение, имеющее диагностическое значение, — возникновение дополнительной точечной мутации в регуляторном гене carR — замена гуанина на аденин в позиции 265(G265A). Такая мутация привела к утрате устойчивости к полимиксину у штаммов V. cholerae El Tor — специфического маркера вибрионов этого биовара [10] и появлению популяции с генотипом ctxB7tcpACIRSrtxA4gyrA(G248T)parC(С254T)carR(G265A)ICEVchInd5, повсеместно распространившейся в странах Азии и Африки. Последующая мутация в этих штаммах затронула геном ICEVchInd5. Возникла делеция размером около 10 т.п.н., при которой были утрачены 4 гена резистентности к антибиотикам (floR, strA, strB и sul2). Cохранился лишь ген dfrA1, определяющий устойчивость к триметоприму. Такие новые геноварианты с генотипом ctxB7tcpACIRSrtxA4gyrA(G248T)parC(С254T)carR(G265A)ICEVchInd5ΔVRIII стали широко распространёнными в 2009–2019 гг. в Кении, Танзании, Уганде, а также Бангладеш, Индии, Йемене (рис. 1, а).
Таким образом, cреди исследованных штаммов выявлены 8 типов генетических вариантов, различающихся набором изменённых генов патогенности и резистентности к антибиотикам.
Анализ изменения генов патогенности и лекарственной устойчивости геновариантов возбудителя холеры, выделенных в России
Для выявления изменений в геноме геновариантов, завезённых в Россию в 1993–2014 гг., получили полногеномные сиквенсы 18 токсигенных штаммов, изолированных от больных (15 изолятов) и из водной среды (1 штамм) на территории России и во время вспышки холеры в 2011 г. на Украине, в Мариуполе (таблица) [24]. Затем провели анализ нуклеотидных последовательностей указанных выше генов патогенности и лекарственной устойчивости этих штаммов и 3 клинических изолятов, сиквенсы которых были получены ранее.
Установлено, что изученные штаммы в зависимости от времени завоза несли в геноме разные аллели ключевых и дополнительных генов патогенности ctxB, tcpA и rtxA1, а также различные типы SXT-элемента. К тому же штаммы различались наличием мутации в генах gyrA, parC и carR, кодирующих резистентность к антибиотикам. На рис. 1, б представлены 5 выявленных генотипов завезённых вариантов возбудителя в Россию (1993–2014 гг.) и на Украину (2011 г.) и показано время их появления на этих территориях. На ранних этапах формирования геновариантов (1994–1999 гг.) в Республику Дагестан, Южно-Сахалинск и Новосибирск из Пакистана, Саудовской Аравии, Китая и Индии соответственно [25] были завезены штаммы c ctxB1, в геноме которых присутствовал ICEVchBan9 (таблица). Практически одновременно (1994–2001 гг.) в другие регионы России (Иркутск, Ачинск, Казань) из Казахстана [18] были занесены геноварианты ctxB1 c другим типом SXT — ICEVcInd5. Дальнейшие изменения генома привели к появлению в России других вариантов возбудителя с дополнительными мутациями в генах патогенности и коровых генах антибиотикоустойчивости. В 2004–2014 гг. в Белорецке, Твери, Мурманске и Ростове-на Дону от больных и из водной среды были выделены завезённые в основном из Индии штаммы [17] с новыми аллелями генов tcpA и rtxA1 — tcpACIRS и rtxA4 соответственно (таблица). Более того, эти варианты возбудителя содержали ещё мутации в генах gyrA и parС, которые привели к резистентности к налидиксовой кислоте. Такие мутации в геноме оказались весьма устойчивыми, поскольку они сохранялись у всех вариантов, выявленных впоследствии. Изолированные в более поздний период штаммы (Москва, Мариуполь; 2010–2014 гг.), отличаясь от предшествующих аллелем ctxB7, относились к новым генетическим вариантам с повышенным патогенным и эпидемическим потенциалом [6, 7]. Более того, при анализе сиквенсов среди новых вариантов возбудителя с геном ctxB7 были обнаружены два штамма (Москва, 2010, 2014 гг.) c дополнительной мутацией в гене carR(C254T), повлекшей за собой утрату резистентности к полимиксину B — диагностического маркера вибрионов El Tor (таблица).
Фенотипический анализ 18 штаммов показал, что 12 штаммов (М-1429, М1430, P-18899, 81, L-3226, L-44150, 76, 153, 39, 186, M1509, 3265/80), выделенных в 2004–2012 гг. и имеющих мутации в генах gyrA и parС, были устойчивы к налидиксовой кислоте, тогда как 6 штаммов (М-1275, М-1270, М-1293, P-17644, М-1327, М-1344), изолированных ранее (1993–2001 гг.) и сохранивших эти гены интактными, были чувствительны к данному антибиотику. Кроме того, 2 штамма (M1509 и 3265/80) c обнаруженной мутацией в гене carR действительно утратили резистентность к полимиксину B (таблица).
Филогенетические связи геновариантов возбудителя холеры из эндемичных стран и России
На рис. 2 представлены филогенетические связи 98 штаммов из Азии, Африки и России, установленные на основе их SNP-анализа. При сравнении полных геномов этих штаммов с референсной последовательностью штамма N16961 выявили 1606 SNP в коровых генах. Построенное филогенетическое дерево и его анализ показали, что исследуемые генетические варианты возбудителя образуют 7 кластеров, в каждый из которых входили штаммы с определённым генотипом (рис. 2). Так, кластеры I и II состояли из геновариантов, изолированных в начальный период их образования (1993–1998 и 1997–2001 гг. cоответственно) и различающихся присутствием в геноме разных типов SXT-элемента. Последующие поэтапные изменения генома возбудителя в процессе эволюции привели к формированию различных групп геноварианов, различающихся новыми мутациями генов патогенности и лекарственной устойчивости и вошедших в состав других 5 кластеров (III–VII), что отражает их геномное разнообразие. Наиболее удалённые от референсного штамма кластеры VI и VII образовали геноварианты, возникшие в последнее десятилетие и несущие сочетание последних наиболее существенных изменений в геноме (ctxB7, carR(C254T) и ctxB7, carR(C254T), ICEVchInd5ΔVRIII) с ранее возникшими мутациями в генах tcpA, rtxA1, gyrA и parС (рис. 2). Тем не менее, несмотря на геномное разнообразие, представленные варианты возбудителя, изолированные в разных эндемичных и неэндемичных регионах, филогенетически связаны друг с другом. Следует особо отметить тесную филогенетическую связь геновариантов из России c изолятами из эндемичных стран Юго-Восточной Азии. В частности, такая связь была выявлена между геновариантами, завезёнными в Россию в 2004–2014 гг., и штаммами, циркулирующими в Индии в тот же период.
Рис. 2. Филогенетическое дерево штаммов из кластеров I–VII генетических вариантов V. cholerae О1 биовара El Tor, построенное на основе полногеномного SNP-анализа. Использована программа «BioNumerics v. 7.6», метод — maximum parsimony tree. Красным обозначены гены патогенности, синим — гены лекарственной устойчивости.
Обсуждение
Одним из важнейших событий в эволюции возбудителя холеры El Tor является образование первых генетических вариантов (1991 г.) за счёт приобретения им генетического материала от V. cho- leraе классического биовара — гена ctxB1. Однако их геном оказался нестабильным, и возникли новые варианты возбудителя с неизвестными ранее свойствами. В нашей работе впервые проведён сравнительный анализ нуклеотидных последовательностей ключевых и дополнительных генов патогенности, а также генов лекарственной устойчивости 104 токсигенных штаммов, циркулирующих в 9 эндемичных регионах и занесённых на территории России на протяжении длительного периода (1991–2022 гг.). Cреди 83 исследованных штаммов, изолированных в эндемичных регионах Азии и Африки в 1991–2022 гг., нами выявлены 8 основных типов генетических вариантов, различающихся набором мутантных генов патогенности (точечные мутации в генах ctxB, tcpA и rtxA1) и резистентности к антибиотикам (разные типы SXT, точечные мутации в генах gyrA, parC и carR и делеция в ICEVchInd5).
Анализ динамики изменения их генома на протяжении почти 30 лет показал, что происходило последовательное накопление мутаций в генах, кодирующих эпидемически важные свойства. Следствием этих мутаций, по данным других исследователей, стало усиление вирулентности патогена за счёт повышенной продукции XT [9, 10], утрата PolR — диагностически значимого признака [14], и изменение спектра резистентности к антибиотикам. При этом каждые последующие мутации обеспечивали селективное преимущество новых геновариантов, выражающееся в большой скорости распространения и вытеснении ими ранее сформированных штаммов [1]. Наиболее существенной стала мутация в гене ctxB1, обусловившая появление штаммов с аллелем ctxB7 в дополнение к другим мутациям в генах патогенности и лекарственной устойчивости. Согласно показанной нами динамике изменения генома именно эти варианты, возникшие в современный период пандемии, широко распространены в странах Азии (Индии, Бангладеш, Йемен) и Африки (Танзания, Уганда, Кения), что полностью согласуется с данными других исследователей [1, 9]. Такая ситуация позволяет прогнозировать их появление на территории России в случае завоза возбудителя холеры в результате расширения экономических и туристических связей с указанными регионами. Однако вопрос о том, какой из известных вариантов станет доминирующим в ближайшие годы, остаётся открытым, поскольку продолжающиеся изменения генома являются, видимо, одним из основных механизмов реализации стратегии выживания патогена как в организме хозяина, так и в меняющейся окружающей среде.
Поскольку возможность таких событий подтверждена выявлением в России различных генетически изменённых штаммов, встал вопрос о динамике изменения их генотипа. Получены сиквенсы геномов 18 штаммов, изолированных в России и на Укриане (Мариуполь). Среднее значение покрытия полных последовательностей геномов составило 66,5. Проведённый нами анализ этих сиквенсов, а также 3 сиквенсов, полученных ранее, показал, что динамика изменения генов патогенности и лекарственной устойчивости этих изолятов на протяжении более 20 лет (1993–2014 гг.) полностью соответствовала таковой штаммов, циркулирующих в эндемичных регионах в разные периоды. Важен установленный нами факт, что в последнее десятилетие были завезены новые геноварианты с высокой вирулентностью и изменённым диагностическим свойством за счёт мутаций в генах ctxB1 и carR. Однако, cогласно нашим данным, уровень геномного разнообразия вариантов возбудителя из России был несколько ниже, чем в эндемичных регионах. Так, пока не обнаружены штаммы с утраченными генами резистентности к 4 антибиотикам (floR, strA, strB и sul2) за счёт протяжённой делеции SXT-элемента.
При проведении филогенетического анализа установлено не только геномное разнообразие геновариантов, но и доминирование в настоящее время в эндемичных регионах недавно возникших вариантов с дополнительными мутациями в генах вирулентности и лекарственной устойчивости, которые усилили их патогенный потенциал. Показанные изменения генома возбудителя в эндемичных регионах указывают на необходимость постоянной их оценки для своевременной разработки адекватных средств генодиагностики и профилактики. Обнаружение в России новых геновариантов с ранее неизвестными мутациями и возможные новые их завозы требует разработки быстрых способов их идентификации, что будет способствовать повышению эффективности эпидемиологического надзора за холерой. В частности, полученные данные были использованы нами для разработки быстрых способов идентификациии новых геновариантов с изменёнными генами патогенности и утраченным биовар-специфическим свойством методом мультилокусной полимеразной цепной реакции, что существенно ускорит их генодиагностику.
Выводы
- Среди изученных геновариантов из 9 эндемичных стран Азии и Африки выявлены 8 генотипов, различающихся мутациями в генах патогенности и лекарственной устойчивости. Среди них глобальное распространение имеют новые высоковирулентные геноварианты c аллелем ctxB7.
- Геноварианты, завезённые в Россию, относились к 5 генотипам, включая штаммы с аллелем ctxB7 и потерянным биовар-специфическим свойством PolR. Установленная их тесная филогенетическая связь со штаммами из эндемичных стран Азии подтверждает их завоз из этого региона.
- Геномное разнообразие вариантов возбудителя холеры в эндемичных регионах и их реальная возможность завоза в Россию указывают на необходимость постоянного молекулярно-генетического мониторинга возбудителя для выявления изменённых генов патогенности и лекарственной устойчивости с целью своевременной разработки адекватных средств диагностики и профилактики.
About the authors
Nina I. Smirnova
Research Anti-Plague Institute “Microbe”
Author for correspondence.
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-7115-6286
D. Sci. (Biol.), Prof., chief researcher, Laboratory of pathogenic vibrios, Russian Research Anti-Plague Institute “Microbe”
Russian Federation, SaratovDarya A. Rybalchenko
Research Anti-Plague Institute “Microbe”
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-3117-8229
junior researcher, Laboratory of pathogenic vibrios, Russian Research Anti-Plague Institute “Microbe”
Russian Federation, SaratovYulii V. Lozovskiy
Research Anti-Plague Institute “Microbe”
Email: rusrapi@microbe.ru
ORCID iD: 0000-0003-4382-7254
Cand. Sci. (Med.), junior researcher, Laboratory of pathogenic vibrios, Russian Research Anti-Plague Institute “Microbe”
Russian Federation, SaratovYaroslav M. Krasnov
Research Anti-Plague Institute “Microbe”
Email: rusrapi@microbe.ru
ORCID iD: 0000-0002-4909-2394
Cand. Sci. (Chem.), Head, Laboratory of genomic and proteomic analysis, Russian Research Anti-Plague Institute “Microbe”
Russian Federation, SaratovVladimir V. Kutyrev
Research Anti-Plague Institute “Microbe”
Email: rusrapi@microbe.ru
ORCID iD: 0000-0003-3788-3452
D. Sci. (Med.), Prof., Academician of RAS, Director, Russian Research Anti-Plague Institute “Microbe”
Russian Federation, SaratovReferences
Supplementary files