Determinants of resistance to levofloxacin and metronidazole in Russian clinical isolates of Helicobacter pylori based on whole-genome sequencing data
- Authors: Starkova D.A.1, Gladyshev N.S.2, Polev D.E.1, Saitova A.T.1, Glazunova K.A.3, Egorova S.A.1, Svarval A.V.1
-
Affiliations:
- St. Petersburg Pasteur institute
- Petrovsky National Research Centre of Surgery
- Saint Petersburg State University
- Issue: Vol 102, No 4 (2025)
- Pages: 465-473
- Section: ORIGINAL RESEARCHES
- URL: https://microbiol.crie.ru/jour/article/view/18926
- DOI: https://doi.org/10.36233/0372-9311-710
- EDN: https://elibrary.ru/YYLGLL
- ID: 18926
Cite item
Abstract
Introduction. Helicobacter pylori infection, however, data on the mechanisms of metronidazole (MTZ) and levofloxacin (LVX) resistance in Russia remain scarce. and levofloxacin (LVX) in Russia.
The aim of the study is to identify the determinants of resistance in clinical isolates of H. pylori to MTZ and LVX using whole-genome sequencing data.
Materials and methods. A retrospective analysis of 43 H. pylori isolates obtained from adult patients (2014–2022) was conducted. Susceptibility to antibiotics was determined using the bacteriological disk diffusion method. Whole-genome sequencing of 43 H. pylori strains was performed using a DNBSEQ-G50 sequencer.
Results. The evaluation of the phenotypic drug susceptibility test results showed that 11 isolates were susceptible to MTZ (MTZ-S), 31 were susceptible to LVX (LVX-S), while 32 isolates were resistant to MTZ (MTZ-R), and 12 were resistant to LVX (LVX-R). To identify the association between phenotypic and genotypic resistance, an analysis of nucleotide substitutions in the gyrA, gyrB, rdxA, frxA, fdxB and fur genes was conducted. Of all the mutations identified in the gyrA and gyrB genes, only D91/GNY in the gyrA gene was associated with phenotypic resistance to LVX and was found in 4/12 (33.3%) of the isolates (p < 0.05). The combined mutation D91G/N/Y+N87K in the gyrA gene was detected in 6/12 (50.0%) of LVX-R isolates (p < 0.001). Point mutations in the rdxA gene were detected in 21.9% (7/32) of MTZ-R isolates, leading to a frameshift or premature termination of protein synthesis. None of the mutations in the frxA, fur and fdxB genes were associated with H. pylori resistance to MTZ.
Conclusion. Based on the results of whole-genome sequencing of Russian clinical isolates of H. pylori, the detection of the combined mutation D91G/N/Y+N87K in the gyrA gene can serve as a predictor of the phenotypic resistance of H. pylori to levofloxacin.
Full Text
Introduction
Helicobacter pylori infection remains one of the most common chronic bacterial infections worldwide and is considered a major risk factor for the development of gastric cancer (approximately 90% of cases) [1]. According to the key principles of the Maastricht VI/Florence Consensus, which underpin national clinical guidelines, H. pylori infection (regardless of symptoms or complications) invariably causes gastritis, for which the only treatment is eradication therapy. Furthermore, the first principle of the consensus recommends conducting drug susceptibility testing prior to prescribing first-line therapy to ensure the rational use of antibiotics [2, 3].
H. pylori resistance to antibiotics is currently recognized as one of the most serious issues. Eradication therapy for H. pylori typically uses a combination of 2–3 antibiotics (such as amoxicillin, clarithromycin, metronidazole (MTZ), tetracycline, levofloxacin (LVX), or rifabutin), a proton pump inhibitor, and, in some regimens, a bismuth [2, 3]. However, the efficacy of H. pylori treatment has been steadily declining in recent years, in parallel with the growth of antibiotic resistance [4, 5]. Furthermore, the widespread use of antibiotics and low patient compliance not only foster resistance in H. pylori but also exert selective pressure on the broader gastrointestinal microbiome. This promotes the selection of resistance genes, enriches the resistome, and facilitates the spread of these genes among bacterial communities [6, 7].
The genetic mechanisms underlying the development of antibiotic resistance (AR) in H. pylori are not fully understood. It is generally believed that H. pylori resistance to antibiotics is due to de novo mutations in chromosomal DNA, which either alter the target of the antibiotic or prevent its activation within cells [6]. Nevertheless, a significant proportion of H. pylori strains resistant to antibiotics lack known genetic determinants of resistance. This indicates the complex and multifactorial nature of resistance mechanisms, which go beyond mutational activity and/or the activation of individual genes in response to antibiotic use. Potential mechanisms of H. pylori resistance include:
- increased expression of efflux system genes;
- synergistic interactions involving mutations, horizontal gene transfer, and activation of protective systems;
- cellular adaptation associated with the formation of biofilms and antibiotic-resistant coccoid forms;
Compensatory mutations that mitigate the fitness cost of resistance through epistatic interactions with resistance determinants;heteroresistance — a phenomenon where subpopulations of cells with different susceptibility to antibiotics are present simultaneously within a bacterial population.
All the mechanisms listed above not only contribute to the formation of H. pylori antibiotic resistance but also accelerate the development of multidrug resistance, which significantly complicates the eradication process and highlights the necessity for a thorough evaluation and improvement of therapeutic approaches [6–8].
The mechanism of action of MTZ, a first-line drug for H. pylori eradication therapy, involves the reduction of the nitro group under anaerobic conditions, leading to the formation of cytotoxic nitroanions and free radicals that damage DNA and disrupt the functioning of bacterial cells. H. pylori resistance to MTZ is due to a complex interplay of genetic and biochemical processes that are not yet fully understood. The main mechanism of resistance to MTZ is mediated by the inactivation of the reductase enzyme genes rdxA (which encodes an oxygen-insensitive nitroreductase) and frxA (which encodes flavin oxidoreductase), which reduces the ability of MTZ to be reduced to its active forms (NO2– and NO22–) and, consequently, diminishes the drug's antimicrobial effect [9, 10]. Mutations in the fur gene, which regulates iron uptake, and fdxB, which encodes ferredoxin, are also thought to contribute to the formation of AR [7, 8, 10]. Increased levels of antioxidant enzymes and the efflux system, as well as mutational activity in genes responsible for repairing damaged DNA, act as additional factors contributing to the development of H. pylori resistance to MTZ [6].
Another antibiotic used as a reserve treatment for Helicobacter pylori infection, LVX, exerts its antibacterial effect by inhibiting topoisomerase II (DNA gyrase) and topoisomerase IV — key enzymes involved in DNA replication and recombination processes. The most common mechanism of H. pylori resistance to fluoroquinolones is due to point mutations in the quinolone resistance-determining regions (QRDR ), particularly in codons 86, 87, 88, 91, 97 of the gyrA gene and codons 481, 484 and 463 of the gyrB gene [6, 7, 10]. However, the role of some of these mutations in the development of H. pylori resistance to LVX has not been proven.
The systematic and reliable data on the resistance rates of H. pylori to LVX and MTZ in Russia are scarce. Additionally, the genetic determinants of resistance to these antibiotics remain largely uncharacterized, impeding the development of effective molecular diagnostic tools for monitoring resistance in clinical practice. The aim of our study was to identify the determinants of resistance in H. pylori clinical isolates to MTZ and LVX using whole-genome sequencing data.
Materials and methods
A retrospective analysis was conducted on 43 clinical isolates of H. pylori obtained from adult patients with gastrointestinal diseases (2014–2022) at the Pasteur Research Institute of Epidemiology and Microbiology in St. Petersburg. The average age of the patients was 44.0 ± 4.5 years (range 22–70 years).
Biopsies from the gastric antrum and body, were placed in thioglycolate medium, homogenized and subsequently cultured on selective medium based on Columbia agar supplemented with 7% defibrinated horse blood and 1% IsoVitalex solution at 37°C under microaerophilic conditions (oxygen content ~5%) using GasPak 100 gas-generating pouches (BBL CampyPak Plus Microaerophilic System envelopes with Palladium Catalyst, BD Biosciences). H. pylori was identified using a set of biochemical tests (urease, catalase and oxidase) and a reagent kit for detecting H. pylori DNA by polymerase chain reaction (DNA-Technology).
To perform the antibiotic susceptibility test, the H. pylori bacterial culture was suspended to a density of 2 on the McFarland scale (~ 6 × 108 CFU/mL), 0.1 mL was applied to the surface of a Petri dish containing Müller–Hinton agar supplemented with 5% defibrinated horse blood, and evenly distributed across the surface with a spatula. The susceptibility of H. pylori isolates to MTZ and LVX was determined using the disk diffusion method: immediately after inoculation onto the agar surface, disks containing MTZ (5 µg/disk) and LVX (5 mcg/disk) were aseptically applied and incubated under microaerophilic conditions (5% O2, 10% CO2, 85% N2) at 37°C for 72 hours. After incubation was complete, the diameter of the zones of complete growth inhibition around the antibiotic disk was measured in millimeters. The interpretation of the disk diffusion method results was based on the threshold values presented in the publication by Z. Zhong et al.: H. pylori strains were considered resistant to MTZ (MTZ-R) when the inhibition zone diameter was ≤ 16 mm, and susceptible (MTZ-S) when the diameter was ≥ 17 mm; resistant to LVX (LVX-R) when the inhibition zone diameter was ≤ 17 mm, and susceptible (LVX-S) when the diameter was ≥ 18 mm [11].
Total DNA from pure cultures of H. pylori was extracted using the QIAamp DNA Mini Kit (QIAGEN GmbH) according to the manufacturer's instructions. The DNA concentration of each sample was quantified using a Qubit 4.0 fluorometer. Whole-genome sequencing was performed on a DNBSEQ-G50 sequencer (MGI Tech Co. Ltd.).
The quality assessment of paired-end libraries, adapter removal, and low-quality sequence trimming (Q-score < 20) were performed using the FastQC v. 0.12.1 and Trim Galore! v. 0.6.7 programs. Bacterial genomes were assembled de novo using the genomic assembler SPAdes v. 3.13.1, and the results were evaluated using QUAST v. 5.2.0 [12, 13]. The obtained genomic sequences were aligned to the H. pylori 26695 reference strain (GenBank acc. no. AE000511.1). The Snippy v.4.6.0 program was used to assess genetic variations between isolates and identify nucleotide substitutions1. Aligned nucleotide sequences were visually analyzed using UGENE v. 38.1 [14]. All genome assemblies of clinical isolates of H. pylori were deposited in the NCBI GenBank database under registration number PRJNA10110372.
Statistical analysis was performed using the R programming language v. 4.3.2. The agreement between phenotypic and genotypic resistance profiles was evaluated using the χ2 test and Fisher's exact test. Differences between groups were considered significant at p < 0.05.
Results
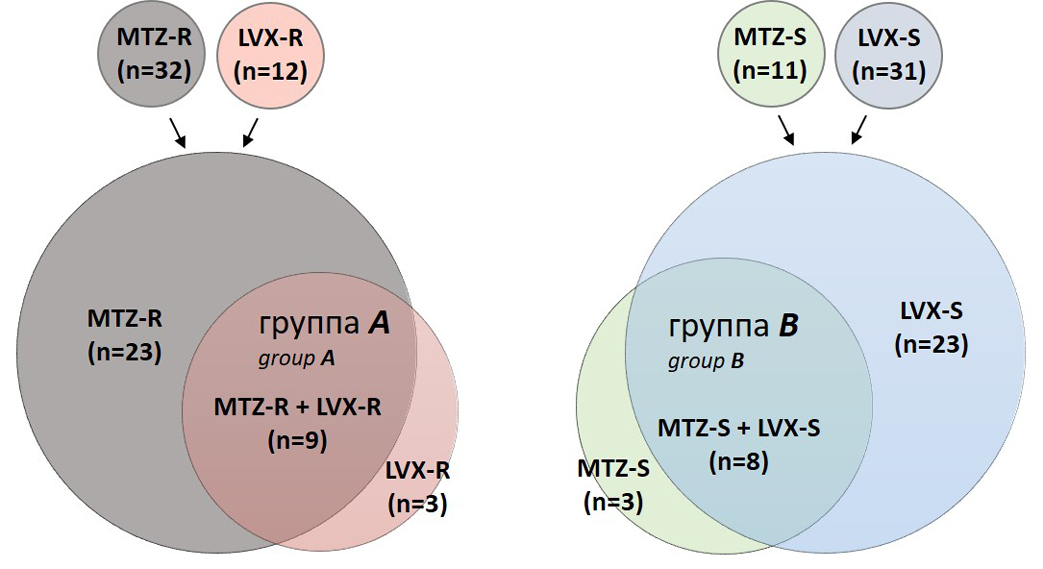
Phenotypic drug susceptibility testing of 43 clinical H. pylori isolates showed that 12 isolates were resistant to LVX (LVX-R), 32 to MTZ (MTZ-R); at the same time, 31 isolates were susceptible to LVX (LVX-S), and 11 to MTZ (MTZ-S). Among the resistant isolates, 9 were resistant to both antibiotics simultaneously (Group A), while among the susceptible isolates, 8 were susceptible to both LVX and MTZ (Group B: Fig. 1).
To identify the determinants of resistance to LVX and MTZ and their association with phenotypic resistance, all 43 isolates underwent whole-genome sequencing, followed by an analysis of nucleotide substitutions in the gyrA, gyrB, rdxA, frxA, fdxB and fur genes.
Of all the mutations in the gyrA gene, only D91N/Y/G was associated with phenotypic drug resistance and was detected in 33.3% (4/12) of LVX-R isolates (p < 0.05) (Table). The missense mutation N87K in the gyrA gene was detected in 16.7% (2/12) of LVX-R isolates and was not found in combination with D91N/Y/G and A88P mutations, while the A88P mutation was identified in only 1 (8.3%) resistant strain in combination with the D91N mutation. Given the absence of these mutations in LVX-S isolates, the combined D91N/Y/G+N87K mutation is significantly associated with resistance in clinical isolates and was detected in 50.0%
(6/12) of LVX-R isolates (p < 0.001). It should be noted that the D91N/Y/G, N87K and A88P mutations in the gyrA gene were found exclusively in group A isolates and were absent in monoresistant isolates. All mutations in the gyrB gene were present in H. pylori isolates regardless of their phenotypic resistance (p > 0.05). Mutations at positions 86 and 97 of the gyrA gene and 463 of the gyrB gene, which are presumably associated with H. pylori resistance to LVX, were not found in our sample.
Fig. 1. Venn diagram showing the combinations of phenotypic drug susceptibility statuses of clinical isolates of H. pylori to MTZ and LVX
Mutations in LVX and MTZ resistance genes in H. pylori clinical isolates of compared to the H. pylori 26695 reference genome, n (%)
Gene (locus) | Amino acid substitution | LVX-R (n = 12) | LVX-S (n = 31) | MTZ-R (n = 32) | MTZ-S (n = 11) | p |
gyrA (HP_0701) | D91N/Y/G | 4 (33.3) | 0 | 0.0040 | ||
N87K | 2 (16.7) | 0 | 0.0730 | |||
A88P | 1 (8.3) | 0 | 0.2790 | |||
gyrB (HP_0501) | D481E | 3 (25.0) | 8 (25.8) | ~ 1.0000 | ||
R484K | 3 (25.0) | 8 (25.8) | ~ 1.0000 | |||
rdxA (HP_0954) | S108A/P | 7 (21.9) | 0 | 0.1628 | ||
R16C | 7 (21.9) | 0 | 0.1628 | |||
L62V | 6 (18.7) | 0 | 0.3122 | |||
Ter211R_ext* stop lost & splice region | 1 (3.1) | 0 | ~ 1.0000 | |||
Q50*stop | 1 (3.1) | 0 | ~ 1.0000 | |||
W52*stop | 1 (3.1) | 0 | ~ 1.0000 | |||
H97fs | 1 (3.1) | 0 | ~ 1.0000 | |||
S43fs | 1 (3.1) | 0 | ~ 1.0000 | |||
I182fs | 1 (3.1) | 0 | ~ 1.0000 | |||
M120fs | 1 (3.1) | 0 | ~ 1.0000 | |||
R131K | 12 (37.5) | 1 (9.1) | 0.1290 | |||
T31E | 13 (40.6) | 5 (45.4) | 0.7794 | |||
D59N | 30 (93.7) | 9 (81.8) | 0.2665 | |||
frxA (HP_0642) | K18fs | 17 (53.1) | 5 (45.4) | 0.6606 | ||
Y19fs | 1 (3.1) | 0 | ~ 1.0000 | |||
Q27*stop | 1 (3.1) | 0 | ~ 1.0000 | |||
R23fs | 1 (3.1) | 0 | ~ 1.0000 | |||
I44fs | 1 (3.1) | 0 | ~ 1.0000 | |||
A70fs | 1 (3.1) | 0 | ~ 1.0000 | |||
R106fs | 1 (3.1) | 1 (9.1) | 0.4507 | |||
W137*stop | 1 (3.1) | 0 | ~ 1.0000 | |||
Q141*stop | 1 (3.1) | 0 | ~ 1.0000 | |||
V215fs | 10 (31.2) | 6 (54.5) | 0.1679 | |||
fdxB (HP_1508) | N424fs | 1 (3.1) | 1 (9.1) | 0.4507 | ||
K426fs | 1 (3.1) | 1 (9.1) | 0.4507 | |||
fur (HP_1027) | C150Y | 3 (9.4) | 2 (18.2) | 0.5890 | ||
N118Q | 7 (21.9) | 3 (27.3) | 0.6982 |
In total, 56 point mutations in the rdxA gene were identified. Among these, 4 (H97fs, S43fs, I182fs, M120fs) were frameshift mutations, 2 (Q50*stop, W52*stop) were nonsense mutations leading to premature termination of the reading frame, and Ter211R_ext* mutation resulted in the loss of stop codon, and fusion of rdxA gene with the adjacent HP_0953 gene. Two mutations (S108A/P, L62V) identified for the first time in MTZ-R isolates, showed no association with phenotypic resistance (p > 0.05). The R131K, T31E and D59N mutations were found in H. pylori isolates from both phenotypic groups and were not associated with phenotypic resistance to MTZ.
We identified 7 frameshift mutations in the frxA gene, 4 of which (Y19fs, R23fs, I44fs, A70fs) were detected only in MTZ-R isolates, while 3 (K18fs, R106fs, V215fs) were found in isolates from both phenotypic groups. At the same time, 3 nonsense mutations leading to premature protein termination were found exclusively in MTZ-R strains (Table). In the fdxB gene, 2 mutations (N424fs, K426fs) were harbored by both MTZ-R and MTZ-S isolates. None of the missense mutations in the frxA and fdxB genes were associated with resistant phenotype.
A total of 12 missense mutations were identified in the fur gene, of which C150Y and N118Q predominated, however, none of mutations were associated with MTZ-R phenotype . Furthermore, no frameshift or nonsense mutations were detected in the fur gene among the studied isolates.
Discussion
The steady increase in H. pylori antibiotic resistance worldwide significantly impacts the effectiveness of eradication therapy regimens. Meta-analysis data from Russia (2011–2020) indicate that the most significant rise in H. pylori resistance was observed for MTZ (33.95%) and LVX (20.0%) [15]. However, subsequent studies from 2015–2019 and 2020–2024 revealed a slight decrease in LVX resistance from 18.3% to 17.1% [15, 16]. Nevertheless, it must be acknowledged that data on the levels and prevalence of antibiotic resistance are still lacking for most Russian regions [2].
Our study, which included 43 H. pylori clinical isolates from St. Petersburg (2014–2022), demonstrated a high level of MTZ resistance — 74.4%. This finding underscores the necessity for a full-scale survey of H. pylori resistance in this region and calls into question the efficacy of MTZ in local eradication regimens. In contrast, resistance to LVX (a second- and third-line drug) was lower in our sample, at 27.9%. Notably, among the LVX-R isolates, only 15.0% were monoresistant.
[17]. Summarizing the previously presented data on clarithromycin resistance, 20.9% of H. pylori isolates are multidrug-resistant, exhibiting simultaneous resistance to three antibiotics: LVX, MTZ, and clarithromycin [17]. It is well known, that multidrug-resistant H. pylori strains represent a major obstacle to successful eradication therapy and pose a significant challenge to global gastroenterological health. Treatment failure rates can reach 30% with single antibiotic resistance and exceed 70% with dual resistance [18, 19]. Since the choice of eradication regimen is empirical, the data obtained highlight the necessity for global, regional, and local monitoring of H. pylori antibiotic resistance in our country. They also emphasize the need to adapt treatment strategies in each region based on these data and to implement a rational antibiotic use program in eradication therapy regimens [20].
Given that whole-genome sequencing is the most accurate, reliable, rapid and efficient method for identifying known resistance patterns, as well as searching for new ones, this method was used in the study to identify LVX and MTZ resistance determinants and their association with the phenotypic drug resistance of the Russian H. pylori population.
Analysis of the obtained data showed that among all mutations in the gyrA and gyrB genes, only D91Y/N/G in the gyrA gene was significantly associated with phenotypic resistance of H. pylori to LVX (p < 0.05). Another mutation associated with the development of resistance to fluoroquinolones, N87K, was detected in only 16.7% of resistant isolates in our study (p > 0.05). Nevertheless, considering the low detection frequency of the D91Y/N/G mutation, as well as the absence of D91 and N87 mutations in LVX-S isolates, the combined detection of D91Y/N/G and N87K mutations should be considered a more reliable predictor of H. pylori resistance to LVX. On the other hand, given that 50.0% of LVX-resistant strains lack resistance markers in their genome, genotyping the gyrA gene alone may be insufficient to detect phenotypic resistance to LVX, which in turn casts doubt on the rationality of using the D91+N87 gyrA genotype as the sole targets when developing PCR tests for determining H. pylori antibiotic resistance in clinical practice. It should be noted that mutations D91 and N87 were present in the genome of only multidrug-resistant isolates, while these mutations were not detected among LVZ-monoresistant isolates. This could indicate both the existence of other, unstudied resistance mechanisms and the involvement of phenotypic resistance mechanisms, such as changes in the expression levels of efflux systems, biofilm formation and others.Moreover, multidrug-resistant strains may emerge under the selective pressure of combination antibiotic therapy, which promotes the accumulation of mutations, including in the gyrA gene, which is associated with resistance to fluoroquinolones. The obtained data require further investigation using an expanded sample of H. pylori isolates resistant and susceptible to LVX.
Given the high heterogeneity of H. pylori strains, elucidating the mechanisms of MTZ resistance remains a complex challenge. It is generally accepted that H. pylori resistance to MTZ is primarily due to the inactivation of the rdxA and frxA genes, which encode the reduced form of nicotinamide adenine dinucleotide phosphate (NAD(P)H) nitroreductase and flavin nitroreductase, respectively, which catalyze the reduction of MTZ levels within the cell [21]. Numerous international studies have demonstrated that most MTZ-R H. pylori strains carry multiple nonsense and/or frameshift mutations, leading to the loss of nitroreductase functional binding sites.
In the current study, mutations causing stop codon loss, premature termination or frameshifts in the rdxA gene were found in 21.9% of MTZ-R isolates and were absent in the MTZ-S group. This finding suggests a potential role of this gene in the development of antibiotic resistance in the Russian H. pylori population. According to E.G. Chua et al., the R16H/C mutation in the rdxA gene is associated with phenotypic resistance of H. pylori isolates to MTZ [22]. Our results showed that the R16C mutation, as well as the S108A/P and L62V mutations in the rdxA gene, were found only in MTZ-R isolates, with frequencies of 21.9%, 21.9% and 18.7%, respectively. However, due to the uneven distribution of MTZ-R and MTZ-S isolates in our sample (74.4% and 25.6%), the impact of these mutations on the development of phenotypic resistance to MTZ remains to be elucidated.
Similarly, loss-of-function mutations in the frxA and fdxB genes have been proposed as potential predictors of H. pylori phenotypic resistance to MTZ. However, in our study, frameshift mutations in these genes were found in isolates from both resistant and susceptible phenotypic groups. Furthermore, no mutations associated with phenotypic resistance to MTZ were detected in the fur gene. These results suggest that frxA, fdxB, and fur genes are unlikely to play a primary role in the development of H. pylori resistance to MTZ. Nevertheless, for a more precise understanding of the resistance mechanisms, further research is necessary, including a comprehensive analysis of other potential genetic factors in conjunction with possible synergistic or epistatic interactions.
Research limitations. This study has several limitations. The sample size was limited both numerically and geographically, preventing a comprehensive analysis of H. pylori resistance patterns across Russia. Furthermore, an uneven distribution of resistant and susceptible isolates was observed, which significantly complicates the interpretation of the data and reduces the statistical reliability of the results. However, despite these limitations, this study provides important information about the resistance patterns of Russian H. pylori clinical isolates and also highlights the necessity for a systematic, nationwide antibiotic resistance monitoring in our country.
Conclusion
Based on whole-genome sequencing data, this study presents the first comprehensive analysis of phenotypic and genotypic resistance to LVX and MTZ in Russian H. pylori clinical isolates. Our results have demonstrated a high prevalence of resistance to both MTZ and LVX, alongside a high frequency of polyresistant isolates resistant to three antibiotics (LVX + MTZ + clarithromycin). Despite the limited sample size, we were able to confirm the key role of D91 and N87 mutations in the gyrA gene in the development of H. pylori resistance to LVX. However, the high frequency of resistant isolates that do not carry known resistance determinants calls into question the effectiveness of current and emerging molecular diagnostic tests for determining H. pylori susceptibility and dictates the necessity for larger-scale studies to elucidate the full spectrum of antibiotic resistance mechanisms in H. pylori .
Our findings emphasize the exceptional importance of issues related to the continuous monitoring for H. pylori antibiotic resistance in our country, as well as tracking genome dynamics and resistance development mechanisms. Such efforts could become a prerequisite for optimizing eradication therapy regimens and improving treatment effectiveness for H. pylori infections across different regions of Russia.
1 URL: https://github.com/tseemann/snippy
2 URL: https://www.ncbi.nlm.nih.gov/bioproject/PRJNA1011037
About the authors
Daria A. Starkova
St. Petersburg Pasteur institute
Email: dariastarkova13@gmail.com
ORCID iD: 0000-0003-3199-8689
Cand. Sci. (Biol.), senior researcher, Laboratory of pathogen identification, senior researcher, Laboratory of molecular epidemiology and evolutionary genetics
Russian Federation, St. PetersburgNikita S. Gladyshev
Petrovsky National Research Centre of Surgery
Email: krinege@mail.ru
ORCID iD: 0000-0003-2732-5676
Researcher, Laboratory of morphology and pathology of the musculoskeletal system, Avtsyn Research Institute of Human Morphology
Russian Federation, MoscowDmitrii E. Polev
St. Petersburg Pasteur institute
Email: polev@pasteurorg.ru
ORCID iD: 0000-0001-9679-2791
Cand. Sci. (Biol.), senior researcher, Head, Laboratory of metagenomics research
Russian Federation, St. PetersburgAlina T. Saitova
St. Petersburg Pasteur institute
Email: saitova@pasteurorg.ru
ORCID iD: 0000-0002-5921-0745
Junior researcher, Laboratory of metagenomics research
Russian Federation, St. PetersburgKsenia A. Glazunova
Saint Petersburg State University
Email: glazunovak03@mail.ru
ORCID iD: 0009-0009-8001-2143
Student
Russian Federation, St. PetersburgSvetlana A. Egorova
St. Petersburg Pasteur institute
Author for correspondence.
Email: egorova@pasteurorg.ru
ORCID iD: 0000-0002-7589-0234
Dr. Sci. (Med.), Head, Laboratory of pathogen identification
Russian Federation, St. PetersburgAlena V. Svarval
St. Petersburg Pasteur institute
Email: svarval@pasteurorg.ru
ORCID iD: 0000-0001-9340-4132
Cand. Sci. (Med.), senior researcher, Laboratory of pathogen identification
Russian Federation, St. PetersburgReferences
- Moss S.F., Shah S.C., Tan M.C., El-Serag H.B. Evolving concepts in Helicobacter pylori management. Gastroenterology. 2024;166(2):267–83. DOI: https://doi.org/10.1053/j.gastro.2023.09.047.
- Ивашкин В.Т., Маев И.В., Лапина Т.Л. и др. Клинические рекомендации Российской гастроэнтерологической ассоциации по диагностике и лечению инфекции Helicobacter pylori у взрослых. Российский журнал гастроэнтерологии, гепатологии, колопроктологии. 2018;28(1):55–70. Ivashkin V.T., Maev I.V., Lapina T.L., et al. Diagnostics and treatment of Helicobacter pylori infection in adults: Clinical guidelines of the Russian gastroenterological association. Russian Journal of Gastroenterology, Hepatology, Coloproctology. 2018;28(1):55–70. DOI: https://doi.org/10.22416/1382-4376-2018-28-1-55-70 EDN: https://elibrary.ru/oulikb
- Malfertheiner P., Megraud F., Rokkas T., et al. European Helicobacter and Microbiota Study group. Management of Helicobacter pylori infection: the Maastricht VI/Florence consensus report. Gut. 2022;71(9):327745. DOI: https://doi.org/10.1136/gutjnl-2022-327745
- Savoldi A., Carrara E., Graham D.Y., et al. Prevalence of antibiotic resistance in Helicobacter pylori: a systematic review and meta-analysis in World Health Organization regions. Gastroenterology. 2018;155(5):1372–82.e17. DOI: https://doi.org/10.1053/j.gastro.2018.07.007
- Zhao M., Zhang Y., Liu S., et al. Eradication of Helicobacter pylori reshapes gut microbiota and facilitates the evolution of antimicrobial resistance through gene transfer and genomic mutations in the gut. BMC Microbiol. 2025;25(1):90. DOI: https://doi.org/10.1186/s12866-025-03823-w
- Hasanuzzaman M., Bang C.S., Gong E.J. Antibiotic resistance of Helicobacter pylori: mechanisms and clinical implications. J. Korean Med. Sci. 2024;39(4):e44. DOI: https://doi.org/10.3346/jkms.2024.39.e44
- Schuetz A.N., Theel E.S., Cole N.C., et al. Testing for Helicobacter pylori in an era of antimicrobial resistance. J. Clin. Microbiol. 2024;62(2):e0073223. DOI: https://doi.org/10.1128/jcm.00732-23
- Dascălu R.I., Bolocan A., Păduaru D.N., et al. Multidrug resistance in Helicobacter pylori infection. Front. Microbiol. 2023;14:1128497. DOI: https://doi.org/10.3389/fmicb.2023.1128497
- Medakina I., Tsapkova L., Polyakova V., et al. Helicobacter pylori antibiotic resistance: molecular basis and diagnostic methods. Int. J. Mol. Sci. 2023;24(11):9433. DOI: https://doi.org/10.3390/ijms24119433
- Saracino I.M., Pavoni M., Zullo A., et al. Next generation sequencing for the prediction of the antibiotic resistance in Helicobacter pylori: a literature review. Antibiotics (Basel). 2021;10(4):437. DOI: https://doi.org/10.3390/antibiotics10040437
- Zhong Z., Zhang Z., Wang J., et al. A retrospective study of the antibiotic-resistant phenotypes and genotypes of Helicobacter pylori strains in China. Am. J. Cancer Res. 2021;11(10):5027–37.
- Gurevich A., Saveliev V., Vyahhi N., Tesler G. QUAST: quality assessment tool for genome assemblies. Bioinformatics. 2013;29(8):1072–5. DOI: https://doi.org/10.1093/bioinformatics/btt086
- Prjibelski A., Antipov D., Meleshko D., et al. Using SPAdes de novo assembler. Curr. Protoc. Bioinformatics. 2020;70(1):e102. DOI: https://doi.org/10.1002/cpbi.102
- Okonechnikov K., Golosova O., Fursov M. Unipro UGENE: a unified bioinformatics toolkit. Bioinformatics. 2012;28(8):1166–7. DOI: https://doi.org/10.1093/bioinformatics/bts091
- Андреев Д.Н., Маев И.В., Кучерявый Ю.А. Резистентность Helicobacter pylori в Российской Федерации: метаанализ исследований за последние 10 лет. Терапевтический архив. 2020;92(11):24–30. Andreev D.N., Maev I.V., Kucheryavyy Yu.A. Helicobacter pylori resistance in the Russian Federation: a meta-analysis of studies over the past 10 years. Therapeutic Archive. 2020;92(11):24–30. DOI: https://doi.org/10.26442/00403660.2020.11.000795 EDN: https://elibrary.ru/gsmibf
- Andreev D.N., Khurmatullina A.R., Maev I.V., et al. Helicobacter pylori antibiotic resistance in Russia: a systematic review and meta-analysis. Antibiotics (Basel). 2025;14(5):524. DOI: https://doi.org/10.3390/antibiotics14050524
- Starkova D., Gladyshev N., Polev D., et al. First insight into the whole genome sequence variations in clarithromycin resistant Helicobacter pylori clinical isolates in Russia. Sci. Rep. 2024;14(1):20108. DOI: https://doi.org/10.1038/s41598-024-70977-4
- Zou Y., Qian X., Liu X., et al. The effect of antibiotic resistance on Helicobacter pylori eradication efficacy: а systematic review and meta-analysis. Helicobacter. 2020;25(4):e12714. DOI: https://doi.org/10.1111/hel.12714
- Gatta L., Scarpignato C., Fiorini G., et al. Impact of primary antibiotic resistance on the effectiveness of sequential therapy for Helicobacter pylori infection: lessons from a 5-year study on a large number of strains. Aliment. Pharmacol. Ther. 2018;47(9):1261–9. DOI: https://doi.org/10.1111/apt.14597
- Полякова В.В., Бодунова Н.А., Цапкова А., Бордин Д.С. Резистентность Helicobacter pylori к антибиотикам и возможности оптимизации эрадикационной терапии. Эффективная фармакотерапия. 2024;20(46):36–44. Polyakova V.V., Bodunova N.A., Tsapkova L.A., Bordin D.S. Helicobacter pylori resistance to antibiotics and possibilities for optimization of eradication therapy. Effective Pharmacotherapy. 2024;20(46):36–44. DOI: https://doi.org/10.33978/2307-3586-2024-20-46-36-44 EDN: https://elibrary.ru/motwfv
- Tuan V.P., Narith D., Tshibangu-Kabamba E., et al. A next-generation sequencing-based approach to identify genetic determinants of antibiotic resistance in Cambodian Helicobacter pylori clinical isolates. J. Clin. Med. 2019;8(6):858. DOI: https://doi.org/10.3390/jcm8060858
- Chua E.G., Debowski A.W., Webberley K.M., et al. Analysis of core protein clusters identifies candidate variable sites conferring metronidazole resistance in Helicobacter pylori. Gastroenterol. Rep. (Oxf.). 2019;7(1):42–9. DOI: https://doi.org/10.1093/gastro/goy048
Supplementary files