Detection of genetic determinants of potentially oncogenic representatives of the intestinal microbiota as biomarkers of colorectal cancer
- Authors: Shumilova V.N.1, Goncharov A.E.1,2, Azarov D.V.1,2, Sitkin S.I.1,2,3, Latariya E.L.1, Aslanov B.I.1, Bobrakov M.A.1, Topuzov R.E.1
-
Affiliations:
- North-Western State Medical University named after I.I. Mechnikov
- Institute of Experimental Medicine
- Almazov National Medical Research Centre
- Issue: Vol 101, No 5 (2024)
- Pages: 668-678
- Section: ORIGINAL RESEARCHES
- URL: https://microbiol.crie.ru/jour/article/view/18688
- DOI: https://doi.org/10.36233/0372-9311-564
- EDN: https://elibrary.ru/fximcu
- ID: 18688
Cite item
Abstract
Relevance. Colorectal cancer (CRC) is the second leading cause of cancer mortality worldwide. Non-invasive diagnostic methods based on the determination of hidden blood in the stool (fecal immunochemical test, guaiac test), which have been proven to be effective in clinical studies, are used for CRC screening. However, a significant disadvantage of the available non-invasive diagnostic methods is the low sensitivity in detecting the oncological process at the early stages. A number of recent studies discuss the relationship between the disease and various potentially oncogenic microorganisms in the human intestinal tract, which can be used to expand the arsenal of non-invasive methods for diagnosing CRC based on molecular genetic examination of a stool sample to identify oncogenic microorganisms.
The aim of this study was to evaluate the possibility of using genetic determinants of potentially oncogenic microorganisms as markers for colorectal cancer, based on a comparison of their prevalence in groups of patients with colorectal cancer, facultative precancerous diseases and patients without intestinal pathology.
Materials and methods. 215 participants were included in the "case–control" study: 70 patients with newly diagnosed colorectal cancer, 70 patients with inflammatory bowel disease, 75 participants without diagnosed intestinal pathology. Polymerase chain reaction (PCR) was used to identify and detect genes of potentially oncogenic microorganisms.
Results and discussion. An association was found between CRC and the presence of the Bacteroides fragilis fragilisin gene (OR 7.00; 95% CI: 2.55–22.50; p < 0.001), species-specific genes of the periodontal pathogenic microorganisms Fusobacterium nucleatum (OR 5.61; 95% CI: 2.87–11.30; p < 0.001) and Porphyromonas gingivalis (OR 16.3; 95% CI: 4.33–106.00; p < 0.001), the clbB gene of pks pathogenicity island of the Enterobacteria (OR 3.44; 95% CI: 1.39–8.51; p = 0.010).
Conclusion. The presence of genetic markers of potentially oncogenic bacterial species and genotypes in the gut microbiome is associated with colorectal cancer. The results obtained support the possibility of using molecular genetic detection of the studied potentially oncogenic microorganisms as a method for non-invasive diagnosis of CRC.
Full Text
Introduction
Colorectal cancer (CRC) is the 3rd most common type of cancer and the 2nd leading cause of cancer mortality in the world [1]. It is noteworthy that the incidence of colorectal cancer is increasing among people under 50 years of age [2]. Noninvasive (stool blood test, guaiac test and fecal immunochemical test (FIT)) and invasive (flexible rectoromanoscopy, colonoscopy) diagnostic methods are used to detect colorectal cancer. The obvious advantages of non-invasive diagnostic methods are their simplicity and availability for screening examinations of persons at risk.
The use of noninvasive diagnostic methods, such as FIT, in screening examinations can achieve a 27% reduction in mortality from CRC [3]. A meta-analysis of the results of 4 randomized controlled trials demonstrated that the use of guaiac test and flexible rectoromanoscopy contributes to the reduction of CRC mortality by 18% and 26%, respectively [4].
However, a significant disadvantage of noninvasive methods is their low diagnostic sensitivity at early stages of the disease. Thus, the sensitivity of FIT at stage I of CRC is 68% (95% CI 57–78%), at stage II — 92% (95% CI 87–96%), at stage III — 82% (95% CI 73–89%) [5]. Thus, there is a need to expand the arsenal of available non-invasive methods for CRC screening.
Specific features of the quantitative and qualitative composition of the microbiota can act as diagnostic markers of CRC, which is discussed in detail in reviews devoted to the role of the intestinal microbiota in the development and diagnosis of intestinal cancer [6, 7]. Numerous studies have revealed changes in the composition of the intestinal microbiome associated with the development of CRC, which suggests the possibility of using the identification of individual representatives of the intestinal microbiome as an independent method of noninvasive diagnosis of CRC or as an addition to existing noninvasive methods.
The first classification model based on intestinal microbial markers and allowing to distinguish patients with CRC from controls was proposed by G. Zeller et al. [8]. The classification algorithm included data on the relative abundance of 22 microorganism species (MOs), but at least half of the predictive power of the model was determined by only 4 species: two Fusobacterium species, Porphyromonas asaccharolytica and Peptostreptococcus stomatis, the presence of each of which was elevated in CRC.
Authors from the Chinese University of Hong Kong developed a diagnostic model demonstrating specificity of 81.2% and sensitivity of 93.8% when combining FIT and 4 bacterial markers (the “m3” marker gene of “Lachnoclostridium” sp., Fusobacterium nucleatum, Hungatella hathewayi (baseonym: Clostridium hathewayi) and Bacteroides clarus) [9]. In an earlier study, an experimental model based on 23 MOs from the Oscillospiraceae (heterotypic synonym: Ruminococcaceae) and Lachnospiraceae families, the Bacteroides, Porphyromonas, Parabacteroides, Collinsella genera and the Enterobacteriaceae family detected 91.7% of CRC cases in a sample of 490 patients, compared to 75.0% of cases detected by FIT [10].
The MOs most closely associated with CRC include both some oral pathobionts and intestinal bacteria. The former, in addition to the already mentioned representatives of the genera Fusobacterium, Porphyromonas and Peptostreptococcus, include Parvimonas micra, Gemella morbillorum, Tannerella forsythia and some other species [11]. Intestinal bacteria are most often represented by enterotoxigenic strains of Bacteroides fragilis, pathogenic and opportunistic bacteria of the Enterobacteriaceae family (Escherichia coli, Salmonella spp., Klebsiella pneumoniae, Citrobacter rodentium), as well as Campylobacter jejuni, Morganella morganii, Enterococcus faecalis, Clostridioides difficile and others [12].
Based on the results of the literature review, we selected the MOs most frequently associated with CRC according to research data. We included colibactin-producing Enterobacteriaceae carrying the clbA and clbB genes as part of the pks pathogenicity island, fragilysin-producing Bacteroides fragilis carrying the bft gene, periodontal pathogenic bacteria Fusobacterium nucleatum and Porphyromonas gingivalis among the potentially oncogenic MOs [6]. An additional factor in favor of including these MOs in the list of potential oncogenes was their association with the stage (progression) of CRC, prognosis/survival and resistance to therapy in patients with CRC [13, 14].
Taking into account probable regional, ethnic and other peculiarities of quantitative and qualitative composition of intestinal microbiota, it is urgent to assess the prevalence of the above mentioned potentially oncogenic MOs among the Russian population and their role in the development of CRC.
The aim of this study is to evaluate the possibility of using genetic determinants of potentially oncogenic MIs as markers of CRC, based on the comparison of their prevalence in groups of patients with CRC, facultative precancerous conditions and patients without intestinal pathology.
Materials and methods
On the basis of clinical departments of the Peter the Great Clinic of the I.I. Mechnikov NWSMU and the City Oncologic Dispensary of St. Petersburg in 2022–2024 a case-control study was conducted. A case-control study was conducted in 2022–2024 at the Peter the Great Clinic of the I.I. Mechnikov NWSMU and the City Oncologic Dispensary of St. Petersburg. The study included 215 participants: a group of 70 patients with diagnosed CRC (CRC group); a group of 70 patients with inflammatory bowel diseases (ulcerative colitis, Crohn's disease — IBD group); 75 participants without diagnosed bowel pathology (control group). The study was conducted with voluntary informed consent of the patients and was approved at the meeting of the Local Ethical Committee of the I.I. Mechnikov NWSMU (protocol No. 10 of 03.11.2021).
In the group of patients with CRC the distribution by stages of oncologic process taking into account TNM classification was as follows: stage 0 — 1 patient; stage 1 — 17 patients; stage 2 — 13 patients; stage 3 — 37 patients; stage 4 — 12 patients.
Inclusion criteria for patients from the CRC group: age over 18 years; diagnosis of CRC established for the first time on the basis of anamnesis, physical examination, morphological examination of tumor material, data of instrumental and laboratory methods of examination; receipt of clinical material (feces) from the patient.
Inclusion criteria for patients from the IBD group: age over 18 years; diagnosed IBD.
Inclusion criteria for patients from the control group: age over 18 years; no diagnosed IBD and CRC.
Exclusion criteria for all study groups: taking an antibacterial drug for the last 30 days and/or undergoing endoscopic examination (colonoscopy, rectomanoscopy) for the last 14 days before the submission of clinical material (feces).
Samples of clinical material obtained from study participants were stored at –20ºC until molecular genetic studies were performed.
DNA from fecal samples for polymerase chain reaction (PCR) was isolated by magnetic particle sorption using the MetaFec reagent kit (Raissol). The primer sequences used earlier for identification of potentially oncogenic MOs were applied in PCR (Table 1).
Table 1. Nucleotide sequences of primers for identification of potentially oncogenic microorganisms
Target gene | GenBank Accession number | Forward primer/reverse primer | Source |
The clbA pks+ (polyketide synthase) gene of E. coli | CP155641.1 | 5'-CTCCACAGGAAGCTACTAAC-3' 5'-CGTGGTGATAAAGTTGGGAC-3' | [15] |
Тhe clbB pks+ (polyketide synthase) gene of E. coli | CP155641.1 | 5'-GCAACATACTCGCCCAGACT-3' 5'-TCTCAAGGCGTTGTTGTTTG-3' probe FAM (5'-CAAGGTGCGCGCTAGGCTGT-3'); | [16] |
Тhe fragilisin (bft) gene synthesized by enterotoxigenic B. fragilis | AF103902.1 | 5'-GAACCTAAAACGGTATATGT-3' 5'-GTTGTAGACATCCCACTGGC-3' | [15] |
fadA adhesion protein gene of F. nucleatum | DQ012973.1 | 5'-GCAGTTTCTGCTTCAGCATTT-3' 5'-TGCTTGAAGTCTTTGAGCTCTTT-3' | [17] |
fimA gene for biofilm formation of P. gingivalis | AB195793.1 | 5'-TGCGACGCTATATGCAAGAC-3' 5'-TCTTCAAAACCACGCTGATG-3' | [17] |
To identify potentially oncogenic MOs, quantitative PCR with hybridization-fluorescence detection (Bio-Rad CFX96 Thermal Cycler) was performed for clbB pks+ and qualitative PCR (Bio-Rad T100 Thermal Cycler) with electrophoretic detection for other markers.
No cases of nonspecific amplification were detected.
The results of marker genes amplification were validated by capillary sequencing of amplicons on the GenomeLab GeXP device (Beckman Coulter, Inc.), and in all cases the sequences corresponding to the sequences of target fragments of marker genes presented in GenBank were obtained (Table 1). Aliquots of total DNA from the first 5 samples of biological material, in which positive PCR results were obtained, were used as positive controls after confirmation of amplification specificity by sequencing, and sterile deionized water served as a negative control.
R program (RStudio) was used for statistical data processing. To quantify the association of potentially oncogenic MOs with CRC, odds ratios (ORs) and 95% confidence intervals (CI) to them were determined. Sensitivity, specificity and CI were calculated using the Epitools1 program. The results were considered statistically significant at p < 0.05.
Results
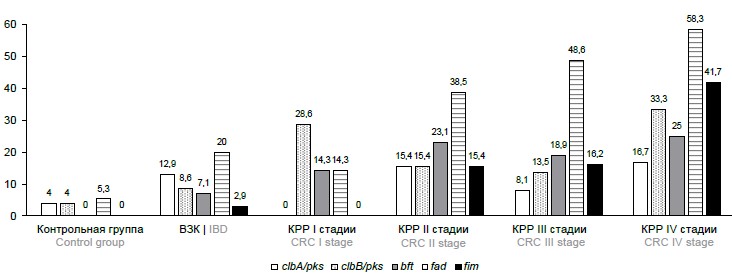
The study of clinical material samples by PCR allowed us to determine the detection rates of genetic markers of potentially oncogenic MOs in patients with CRC at different stages of the disease and in the comparison groups (Fig. 1). A higher prevalence of potentially oncogenic markers was detected in patients with CRC compared to patients with CRC and control group participants. There was a direct correlation between the stage of cancer and the prevalence of potentially oncogenic markers, in particular, F. nucleatum DNA, and a higher frequency of detection of periodontal pathogenic MOs at late stages of the cancer process. Colibactin-producing bacteria carrying the clbB gene of the pks pathogenicity island prevail at early tumor stages.
The prevalence of potentially oncogenic microorganisms in patients with CRC at various stages of the disease and in comparison groups.
The association of some potentially oncogenic representatives of the intestinal microbiome with CRC was observed in the course of the case-control study (Table 2). Thus, the presence of the bft gene of enterotoxigenic B. fragilis was significantly different between the groups of patients with IBD and patients with CRC and associated with the microbiome of patients with CRC (OR = 3.25; 95% CI 1.16–10.6; p = 0.033). Furthermore, a higher prevalence of this gene was found in patients with CRC compared to patients with IBD, while the enterotoxigenic B. fragilis gene was not detected in samples from participants in control group.
Table 2. Representatives of the intestinal microbiome associated with CRC
Target gene | CRC, n (%) | IBD, n (%) | Control, n (%) | OR (95% CI) | |||
CRC/IBD | CRC/Control | CRC + IBD/Control | CRC/IBD + Control | ||||
Fragilisin bft gene of enterotoxigenic B. fragilis | 14 (20%) | 5 (7,1%) | 0 (0%) | 3,25 (1,16–10,6) | ∞ | ∞ | 7 (2,55–22,5) |
fadA adhesion protein gene of F. nucleatum | 31 (44%) | 14 (20%) | 4 (5,3%) | 3,18 (1,52–6,9) | 14,1 (5,13–50,1) | 8,41 (2,89–24,46) | 5,61 (2,87–11,3) |
fimA gene for biofilm formation of P. gingivalis | 13 (19%) | 2 (2,9%) | 0 (0%) | 7,75 (2,03–50,9) | ∞ | ∞ | 16,3 (4,33–106) |
Тhe gene clbA of the pks pathogenicity island of E. coli | 7 (10%) | 9 (13%) | 3 (4,0%) | 0,75 (0,25–2,15) | 2,67 (0,71–12,8) | 3,09 (0,87–10,99) | 1,23 (0,44–3,21) |
Тhe gene clbB of the pks pathogenicity island of E. coli | 13 (19%) | 6 (8,6%) | 3 (4,0%) | 2,43 (0,9–7,31) | 5,47 (1,49–20,14) | 3,77 (1,08–13,18) | 3,44 (1,39–8,51) |
Note. ∞ — due to the zero value of the denominator, it is not possible to calculate the OR indicator.
According to the data of this study, the presence of DNA of the periodontal pathogenic MO F. nucleatum was found to be associated with CRC, and its detection rate differed between the case and control groups. In patients with IBD, CRC was associated with the presence of DNA from another periodontal pathogenic MO, P. gingivalis (OR = 7.75; 95% CI 2.03–50.9; p = 0.009). The frequency of F. nucleatum DNA identification was higher in patients with CRC (44%) compared to patients with IBD (22%) and control group participants (5.3%). A high prevalence of P. gingivalis DNA was found in CRC patients (in 19% of samples) compared to IBD patients (in 2.9% of samples), P. gingivalis DNA was not detected in control group participants.
This study found that the presence of the clbB gene of the pathogenicity island of Enterobacteriaceae pks (OR = 5.47; 95% CI 1.49–20.14; p = 0.015) was associated with CRC and allowed us to separate the microbiome of patients with this pathology from that of control participants. The detectable Enterobacteriaceae pks pathogenicity island clbB gene was found in 19% of samples from patients with CRC, in 8.6% from patients with IBD, and in 4% from control participants. In contrast, the frequency of clbA gene identification did not differ between the comparison groups. The clbA gene was identified in 10% of samples from patients with CRC, in 13% of samples from patients with IBD, and in 4% of samples from control group participants.
In patients with CRC, the frequency of detection of markers of potentially oncogenic MOs did not depend on tumor localization: detection of F. nucleatum in tumors of distal location in 14 cases, proximal location — in 15 cases; P. gingivalis — in 5 and 7 cases; clbB gene of the Enterobacteriaceae pks pathogenicity island — in 6 and 7 cases; clbA gene — in 2 and 5 cases; fragilisin bft gene of enterotoxigenic B. fragilis – in 6 and 7 cases, respectively. No significant differences related to the morphological type of the tumor were revealed (detection of F. nucleatum in 18 cases of highly differentiated tumor, in 13 cases of low-differentiated tumor; P. gingivalis — in 6 and 7 cases; clbB gene of the Enterobacteriaceae pks pathogenicity island — in 7 and 6 cases; clbA gene — in 4 and 3 cases; fragilisin bft gene of enterotoxigenic B. fragilis — in 6 and 8 cases, respectively). Thus, the detection of markers of potentially oncogenic MOs allows to distinguish CRC regardless of the localization and morphological type of tumor.
Taking into account the obtained data, we calculated the sensitivity and specificity of the diagnostic test, which allows us to distinguish the microbiomes of patients with CRC from the microbiomes of patients with IBD and control group participants in case of separate and joint detection of genetic determinants of potentially oncogenic MOs (Table 3). The optimal (in terms of combination of sensitivity and specificity) variant of testing seems to be the one that takes into account the fact of DNA identification of at least 1 out of 5 potential microbial onco-markers.
Table 3. Characterization of sensitivity and specificity of identification of potentially oncogenic microorganisms for the diagnosis of CRC
Parameter of the diagnostic method | CRC, n | IBD + control, n | Sensitivity, % (95% CI) | Specificity, % (95% CI) |
Detection of the clbA gene | 7 | 11 | 10 (5–19) | 92 (86–95) |
Detection of the fragilisin bft gene of enterotoxigenic B. fragilis | 14 | 5 | 20 (12–31) | 97 (92–99) |
Detection the fadA adhesion protein gene of F. nucleatum | 31 | 18 | 44 (33–56) | 88 (81–92) |
Detection the fimA gene for biofilm formation of P. gingivalis | 13 | 2 | 19 (11–29) | 99 (95–99) |
Detection of the clbB gene | 13 | 9 | 19 (11–29) | 94 (89–97) |
Detection of 1 or more microorganisms | 36 | 27 | 51 (39–64) | 81 (74–87) |
Detection of 2 or more microorganisms | 22 | 10 | 31 (21–44) | 93 (88–97) |
Detection of 3 or more microorganisms | 12 | 4 | 17 (9–28) | 97 (93–99) |
Detection of 4 or more microorganisms | 6 | 3 | 9 (3–18) | 98 (94–100) |
Detection of 5 or more microorganisms | 2 | 2 | 3 (0,35–10,00) | 99 (95–100) |
Discussion
In the present study, we evaluated the prevalence of potentially oncogenic CRC-associated MOs among residents of a Russian megacity with diagnosed CRC, IBD and individuals without intestinal tract pathology.
It is known that B. fragilis is a commensal representative of the intestinal microbiota. Both non-toxigenic strains of B. fragilis (not associated with CRC) [18] and enterotoxigenic B. fragilis synthesizing fragilisin, a toxin that cleaves the cell adhesion protein E-cadherin, disrupting the intestinal barrier and contributing to the development of diarrhea, are present in the gastrointestinal tract [19]. In addition, this toxin can activate the implementation of the Wnt/β-catenin signaling pathway, promoting cell proliferation, induction of inflammatory mediator production, and carcinogenesis [20]. The toxin of B. fragilis is encoded by the bft gene with 3 isotypes (bft 1, bft 2, bft 3), which is located in the pathogenicity island (PAI) and flanked by genes encoding mobilization proteins and representing CTn86 and CTn9343 conjugative transposon sequences. Non-toxigenic strains of B. fragilis do not possess a pathogenicity island, but in the presence of conjugative transposons in some strains PAI can transfer from enterotoxigenic B. fragilis to non-toxigenic strains of B. fragilis [21]. The role of enterotoxigenic B. fragilis as a “driver” in the “driver-passenger” model has been established, which consists in the damage of epithelial DNA by “driver” bacteria, which leads to the development of carcinogenesis and changes in the microbial community; further in the process of oncogenesis, “drivers” are displaced by commensal bacteria - “passengers” with tumor-promoting properties [22]. Enterotoxigenic strains of B. fragilis can inhibit the exosomal microRNA miR-149-3p, which mediates intercellular interactions by modulating the differentiation of Th17 cells, contributing to inflammation and carcinogenesis in the intestine [23].
The association of enterotoxigenic B. fragilis with CRC identified in our study was confirmed in different ethnic cohorts. For example, in the Iranian population, the frequency of bft gene detection in colorectal biopsy specimens of patients with CRC ranged from 30.5 to 47% compared with control group participants — up to 6.25% of biopsy specimens [24, 25]. Moreover, a higher prevalence of enterotoxigenic B. fragilis in biopsy specimens was found among patients from Tehran with ulcerative colitis compared to individuals without intestinal pathology [26]. In Canadian and French cohorts of patients with CRC, a high prevalence of enterotoxigenic B. fragilis was found (up to 32% of samples) compared to control subjects [15, 19]. The results of the European Prospective Investigation into Nutrition and Cancer (EPIC) showed that in the European cohort IgA- and IgG-seropositivity to enterotoxigenic B. fragilis and genotoxic E. coli was significantly associated with the development of CRC [27].
F. nucleatum is a Gram-negative non-spore-forming obligate anaerobic MO of the family Fusobacteriaceae and is the dominant MO in dental plaque biofilms [28]. F. nucleatum promotes carcinogenesis and metastasis through multiple mechanisms: promotes proliferation of myeloid suppressor cells; accelerates T cell apoptosis, suppresses T cell proliferation, thereby orchestrating a tumor microenvironment that promotes oncogenesis and metastasis; induces expression of the molecular structure protein S100A9 and triggers activation of M2 macrophages via nuclear factor-κB, thereby activating tumor cell proliferation and migration; stimulates the proliferation of Foxp3+ regulatory T cells and inhibits the proliferation and function of effector T cells, impeding the antitumor immune response; induces the secretion of circulating exosomes, enhancing tumor invasion; furthermore, a possible role of F. nucleatum in resistance to tumor immunotherapy and chemotherapy [29]. A key virulence/oncogenicity factor of F. nucleatum is the adhesin FadA, which regulates annexin A1 expression via E-cadherin. Induction of annexin A1, which is a modulator of Wnt/β-catenin, specifically stimulates colorectal carcinoma cells, contributing to the progression of CRC [30]. Furthermore, F. nucleatum stimulates inflammatory and antiapoptotic responses in CRC cells through the release of ADP-heptose and activation of the ALPK1/TIFA axis [31]. Recently, a distinct Fna C2 clade of F. nucleatum associated with CRC has been characterized, which exhibits increased virulence [32].
According to a study conducted by J. Jones et al., it is F. nucleatum and enterotoxigenic B. fragilis represent two key pathobionts that promote oncogenic reprogramming of intestinal epithelial cells [11].
P. gingivalis is an anaerobic oral bacterium that causes chronic periodontitis. Over the past decade, the mechanisms by which P. gingivalis promotes tumor progression and stimulates cell invasion and metastasis of tumor cells have been identified. These mechanisms include increased expression of proinflammatory factors and matrix metalloproteinases that degrade the basal membranes and extracellular matrix of the intestinal epithelium [33].
According to these studies, an association was found between the presence of periodontopathogenic bacteria in fecal samples and biopsy material from CRC. A case-control study using metagenomic sequencing revealed that the detection rate of Fusobacterium was higher in CRC patients (31.9% vs. 11.7% in controls) and the development of CRC was associated with the presence of F. nucleatum (OR = 4.11; 95% CI 1.62–10.47; p = 0.004) and P. gingivalis (OR = 5.17; 95% CI 1.75–15.25; p = 0.001) [34]. A study conducted in France revealed that the prevalence of F. nucleatum was higher among patients with CRC (70.4%) compared to individuals without intestinal pathology [19].
P. gingivalis and F. nucleatum, oral bacteria belonging to the so-called red and orange complexes, can not only induce chronic inflammation but also promote oncogenesis in both the oral cavity and intestine, possibly having a synergistic effect [35].
It should also be noted that all three oncogenic bacteria (F. nucleatum, B. fragilis and P. gingivalis) are potentially associated not only with the development of CRC, but also with a worse prognosis for patients (lower survival rate) [13, 14]. Furthermore, the results of our study indicate a direct relationship between the stage of the cancer process and the prevalence of periodontal pathogenic MOs F. nucleatum and P. gingivalis.
Various authors have also found colibactin-producing genotypes of Enterobacteriaceae to be associated with CRC. Colibactin is a genotoxin that causes double-stranded DNA breaks, cell cycle arrest, and chromosomal instability in eukaryotic cells. It is synthesized by an assembly line of non-ribosomal polyketide synthase (pks) consisting of 19 genes (clbA to clbS) located on a 54 bp genomic island [36]. The pks island is present in the genomes of K. pneumoniae, K. aerogenes (basonym: Enterobacter aerogenes), Citrobacter koseri, and in the phylogenetic groups of E. coli [19]. Infection may occur at early stages of ontogenesis: it is known that transmission of colibactin-producing genotypes of Enterobacteriaceae occurs in the perinatal period during passage through the birth canal (the clbB gene was detected in 87.5% of children born naturally, in 12.5% — by cesarean section) and as a result of breastfeeding [37]. The frequency of clbN gene detection was higher in CRC patients (49.4%) compared to control participants (24%; p < 0.005), with a high prevalence (72.2%) found in the last stage IV compared to stage I/II CRC (42.3%; p < 0.05) and stage III CRC (43.2%; p < 0.05) [19]. In a Japanese cohort study (543 colorectal neoplasia cases (22 CRC and 521 adenomas), 425 control participants), pks+-E. coli was found in 32.6% of fecal samples from patients with colorectal neoplasia and in 30.8% from control participants [38]. Also among the Canadian cohort, the frequency of colonization with pks+ bacteria was found not to differ between control group participants (42%) and CRC patients (46%), notably, pks+ bacteria were common in advanced stages of CRC (40/79; 52%) compared to early tumor stages (3/15; 20%; p < 0.05) [15]. In a cohort study, pks+-E. coli was detected in 9.44% (111/1175) of biopsy specimens from patients with CRC, with DNA pks+-E. coli was inversely associated with the stage of tumor process (p = 0.008) [16]. Our study revealed a high prevalence of the clbB gene of the pathogenicity island of pks+-E. coli at the I stage of the cancer process.
Conclusion
The results of this study allow us to conclude that CRC in patients of a large metropolitan area is associated with the presence of genes of potentially oncogenic bacteria in the intestinal microbiome, in particular, species-specific genes of periodontal pathogens F. nucleatum, P. gingivalis, toxin-fragilisin bft gene of B. fragilis, polyketide synthase clbB gene of Enterobacteriaceae pks pathogenicity island. The results obtained are consistent with the current ideas about the pathogenetic role of these bacteria and/or their toxin-producing strains in CRC.
Molecular genetic detection of the above genetic determinants of potentially oncogenic MOs can be applied as a method of non-invasive diagnosis of CRC regardless of the localization and morphological type of tumor, both separately and together with other recommended tests.
Given the available data on the association of these bacteria with the stage/progression of CRC, prognosis/survival and resistance to immunotherapy and chemotherapy, we can assume the possibility of their use as non-invasive biomarkers for predicting the course and outcomes of CRC, response to antitumor therapy, and in the future for the development of appropriate cancer prevention measures, including personalized correction of oral and intestinal dysbiosis and/or their sanitation.
1 URL: http://epitools.ausvet.com.au
About the authors
Viktoriya N. Shumilova
North-Western State Medical University named after I.I. Mechnikov
Author for correspondence.
Email: tori.lovas@gmail.com
ORCID iD: 0009-0004-4271-6530
postgraduate student, Department of epidemiology, parasitology and desinfectology, epidemiologist, Epidemiological department
Россия, Saint PetersburgArtemij E. Goncharov
North-Western State Medical University named after I.I. Mechnikov; Institute of Experimental Medicine
Email: tori.lovas@gmail.com
ORCID iD: 0000-0002-5206-6656
D. Sci. (Med.), Head, Laboratory of functional genomics and proteomics of microorganisms, Professor, Department of epidemiology, parasitology and desinfectology
Россия, Saint Petersburg; Saint PetersburgDaniil V. Azarov
North-Western State Medical University named after I.I. Mechnikov; Institute of Experimental Medicine
Email: tori.lovas@gmail.com
ORCID iD: 0000-0003-2483-5144
Cand. Sci. (Med.), assistant, Department of epidemiology, parasitology and desinfectology, research associate, Laboratory of functional genomics and proteomics of microorganisms
Россия, Saint Petersburg; Saint PetersburgStanislav I. Sitkin
North-Western State Medical University named after I.I. Mechnikov; Institute of Experimental Medicine; Almazov National Medical Research Centre
Email: tori.lovas@gmail.com
Scopus Author ID: 0000-0003-0331-0963
Cand. Sci. (Med.), Head, Epigenetics & metagenomics research group, Institute of Perinatology and Pediatrics, Associate Professor, Department of propaedeutics of internal diseases, gastroenterology and dietetics named after S.M. Ryss, leading researcher
Россия, Saint Petersburg; Saint Petersburg; Saint PetersburgElgudzha L. Latariya
North-Western State Medical University named after I.I. Mechnikov
Email: tori.lovas@gmail.com
ORCID iD: 0000-0002-9569-8485
Cand. Sci. (Med.), Associate Professor, Department of faculty surgery named after I.I. Grekov, Vice-rector for clinical work
Россия, Saint PetersburgBatyrbek I. Aslanov
North-Western State Medical University named after I.I. Mechnikov
Email: tori.lovas@gmail.com
ORCID iD: 0000-0002-6890-8096
D. Sci. (Med.), Associate Professor, Head, Department of epidemiology, parasitology and disinfectology; Head, Research laboratory of molecular epidemiology and bacteriophage research
Россия, Saint PetersburgMikhail A. Bobrakov
North-Western State Medical University named after I.I. Mechnikov
Email: tori.lovas@gmail.com
ORCID iD: 0009-0003-3003-8886
surgeon, oncologist, assistant, Department of hospital surgery named after V.A. Oppel
Россия, Saint PetersburgRustem E. Topuzov
North-Western State Medical University named after I.I. Mechnikov
Email: tori.lovas@gmail.com
ORCID iD: 0000-0001-8168-6187
Cand. Sci. (Med.), surgeon, oncologist, Associate Professor, Department of hospital surgery named after V.A. Oppel
Россия, Saint PetersburgReferences
- Baidoun F., Elshiwy K., Elkeraie Y., et al. Colorectal cancer epidemiology: recent trends and impact on outcomes. Curr. Drug Targets. 2021;22(9):998–1009. DOI: https://doi.org/10.2174/1389450121999201117115717
- Patel S.G., Karlitz J.J., Yen T., et al. The rising tide of early-onset colorectal cancer: a comprehensive review of epidemiology, clinical features, biology, risk factors, prevention, and early detection. Lancet Gastroenterol. Hepatol. 2022;7(3):262–74. DOI: https://doi.org/10.1016/S2468-1253(21)00426-X
- Giorgi Rossi P., Vicentini M., Sacchettini C., et al. Impact of screening program on incidence of colorectal cancer: a cohort study in Italy. Am. J. Gastroenterol. 2015;110(9):1359–66. DOI: https://doi.org/10.1038/ajg.2015.240
- Fitzpatrick-Lewis D., Ali M.U., Warren R., et al. Screening for colorectal cancer: a systematic review and meta-analysis. Clin. Colorectal Cancer. 2016;15(4):298–313. DOI: https://doi.org/10.1016/j.clcc.2016.03.003
- Niedermaier T., Tikk K., Gies A., et al. Sensitivity of fecal immunochemical test for colorectal cancer detection differs according to stage and location. Clin. Gastroenterol. Hepatol. 2020;18(13):2920–8.e6. DOI: https://doi.org/10.1016/j.cgh.2020.01.025
- Шумилова В.Н., Гончаров А.Е., Латария Э.Л., Асланов Б.И. Роль кишечной микробиоты в диагностике колоректального рака. Фундаментальная и клиническая медицина. 2024;9(1):112–23. Shumilova V.N., Goncharov A.E., Latariya E.L., Aslanov B.I. The role of the gut microbiota in the development of colorectal cancer. Fundamental and Clinical Medicine. 2024;9(1):112–23. DOI: https://doi.org/10.23946/2500-0764-2024-9-1-112-123 EDN: https://elibrary.ru/mzdotq
- Костин Р.К., Малюгин Д.А., Соленова Л.Г., Кулаева Е.Д. Микробиота желудочно-кишечного тракта и канцерогенез в различных органах человека. Журнал микробиологии, эпидемиологии и иммунобиологии. 2023;100(1):110–25. Kostin R.K., Malyugin D.A., Solenova L.G., Kulaeva E.D. Gut microbiota and carcinogenesis in various human organs. Journal of Microbiology, Epidemiology and Immunobiology. 2023;100(1):110–25. DOI: https://doi.org/10.36233/0372-9311-310 EDN: https://elibrary.ru/cybybs
- Zeller G., Tap J., Voigt A.Y., et al. Potential of fecal microbiota for early-stage detection of colorectal cancer. Mol. Syst. Biol. 2014;10(11):766. DOI: https://doi.org/10.15252/msb.20145645
- Liang J.Q., Li T., Nakatsu G., et al. A novel faecal Lachnoclostridium marker for the non-invasive diagnosis of colorectal adenoma and cancer. Gut. 2020;69(7):1248–57. DOI: https://doi.org/10.1136/gutjnl-2019-318532
- Baxter N.T., Ruffin M.T. 4th, Rogers M.A., Schloss P.D. Microbiota-based model improves the sensitivity of fecal immunochemical test for detecting colonic lesions. Genome Med. 2016;8(1):37. DOI: https://doi.org/10.1186/s13073-016-0290-3
- Jones J., Shi Q., Nath R.R., Brito I.L. Keystone pathobionts associated with colorectal cancer promote oncogenic reprograming. PLoS One. 2024;19(2):e0297897. DOI: https://doi.org/10.1371/journal.pone.0297897
- Dougherty M.W., Jobin C. Intestinal bacteria and colorectal cancer: etiology and treatment. Gut Microbes. 2023;15(1):2185028. DOI: https://doi.org/10.1080/19490976.2023.2185028
- Wei Z., Cao S., Liu S., et al. Could gut microbiota serve as prognostic biomarker associated with colorectal cancer patients' survival? A pilot study on relevant mechanism. Oncotarget. 2016;7(29):46158–72. DOI: https://doi.org/10.18632/oncotarget.10064
- Kerdreux M., Edin S., Löwenmark T., et al. Porphyromonas gingivalis in colorectal cancer and its association to patient prognosis. J. Cancer. 2023;14(9):1479–85. DOI: https://doi.org/10.7150/jca.83395
- Oliero M., Hajjar R., Cuisiniere T., et al. Prevalence of pks + bacteria and enterotoxigenic Bacteroides fragilis in patients with colorectal cancer. Gut Pathog. 2022;14(1):51. DOI: https://doi.org/10.1186/s13099-022-00523-y
- Arima K., Zhong R., Ugai T., et al. Western-style diet, pks Island-carrying Escherichia coli, and colorectal cancer: analyses from two large prospective cohort studies. Gastroenterology. 2022;163(4):862–74. DOI: https://doi.org/10.1053/j.gastro.2022.06.054
- de la Fuente C., Flores S., Moraga M. DNA from human ancient bacteria: a novel source of genetic evidence from archaeological dental calculus. Archaeometry. 2013;55(4):767–78. DOI: https://doi.org/10.1111/j.1475-4754.2012.00707.x
- Chan J.L., Wu S., Geis A.L., et al. Non-toxigenic Bacteroides fragilis (NTBF) administration reduces bacteria-driven chronic colitis and tumor development independent of polysaccharide A. Mucosal Immunol. 2019;12(1):164–77. DOI: https://doi.org/10.1038/s41385-018-0085-5
- Périchon B., Lichtl-Häfele J., Bergsten E., et al. Detection of Streptococcus gallolyticus and four other CRC-associated bacteria in patient stools reveals a potential "driver" role for enterotoxigenic Bacteroides fragilis. Front. Cell. Infect. Microbiol. 2022;12:794391. DOI: https://doi.org/10.3389/fcimb.2022.794391
- Sears C.L., Geis A.L., Housseau F. Bacteroides fragilis subverts mucosal biology: from symbiont to colon carcinogenesis. J. Clin. Invest. 2014;124(10):4166–72. DOI: https://doi.org/10.1172/JCI72334
- Buckwold S.L., Shoemaker N.B., Sears C.L., Franco A.A. Identification and characterization of conjugative transposons CTn86 and CTn9343 in Bacteroides fragilis strains. Appl. Environ. Microbiol. 2007;73(1):53–63. DOI: https://doi.org/10.1128/AEM.01669-06
- Tjalsma H., Boleij A., Marchesi J.R., Dutilh B.E. A bacterial driver-passenger model for colorectal cancer: beyond the usual suspects. Nat. Rev. Microbiol. 2012;10(8):575–82. DOI: https://doi.org/10.1038/nrmicro2819
- Cao Y., Wang Z., Yan Y., et al. Enterotoxigenic Bacteroides fragilis promotes intestinal inflammation and malignancy by inhibiting exosome-packaged miR-149-3p. Gastroenterology. 2021;161(5):1552–66.e12. DOI: https://doi.org/10.1053/j.gastro.2021.08.003
- Jasemi S., Emaneini M., Fazeli M.S., et al. Toxigenic and non-toxigenic patterns I, II and III and biofilm-forming ability in Bacteroides fragilis strains isolated from patients diagnosed with colorectal cancer. Gut Pathog. 2020;12:28. DOI: https://doi.org/10.1186/s13099-020-00366-5
- Zamani S., Taslimi R., Sarabi A., et al. Enterotoxigenic Bacteroides fragilis: a possible etiological candidate for bacteriallyinduced colorectal precancerous and cancerous lesions. Front. Cell. Infect. Microbiol. 2020;9:449. DOI: https://doi.org/10.3389/fcimb.2019.00449
- Zamani S., Hesam Shariati S., Zali M.R., et al. Detection of enterotoxigenic Bacteroides fragilis in patients with ulcerative colitis. Gut Pathog. 2017;9:53. DOI: https://doi.org/10.1186/s13099-017-0202-0
- Butt J., Jenab M., Werner J., et al. Association of pre-diagnostic antibody responses to Escherichia coli and Bacteroides fragilis toxin proteins with colorectal cancer in a European cohort. Gut Microbes. 2021;13(1):1–14. DOI: https://doi.org/10.1080/19490976.2021.1903825
- Signat B., Roques C., Poulet P., Duffaut D. Fusobacterium nucleatum in periodontal health and disease. Curr. Issues Mol. Biol. 2011;13(2):25–36. DOI: https://doi.org/10.21775/cimb.013.025
- Ye C., Liu X., Liu Z., et al. Fusobacterium nucleatum in tumors: from tumorigenesis to tumor metastasis and tumor resistance. Cancer Biol. Ther. 2024;25(1):2306676. DOI: https://doi.org/10.1080/15384047.2024.2306676
- Rubinstein M.R., Baik J.E., Lagana S.M., et al. Fusobacterium nucleatum promotes colorectal cancer by inducing Wnt/β-catenin modulator Annexin A1. EMBO Rep. 2019;20(4):e47638. DOI: https://doi.org/10.15252/embr.201847638
- Martin-Gallausiaux C., Salesse L., Garcia-Weber D., et al. Fusobacterium nucleatum promotes inflammatory and antiapoptotic responses in colorectal cancer cells via ADP-heptose release and ALPK1/TIFA axis activation. Gut Microbes. 2024;16(1):2295384. DOI: https://doi.org/10.1080/19490976.2023.2295384
- Zepeda-Rivera M., Minot S.S., Bouzek H., et al. A distinct Fusobacterium nucleatum clade dominates the colorectal cancer niche. Nature. 2024;628(8007):424–32. DOI: https://doi.org/10.1038/s41586-024-07182-w
- Chang C., Wang H., Liu J., et al. Porphyromonas gingivalis infection promoted the proliferation of oral squamous cell carcinoma cells through the miR-21/PDCD4/AP-1 negative signaling pathway. ACS Infect. Dis. 2019;5(8):1336–47. DOI: https://doi.org/10.1021/acsinfecdis.9b00032
- Ahn J., Sinha R., Pei Z., et al. Human gut microbiome and risk for colorectal cancer. J. Natl Cancer Inst. 2013;105(24):1907–11. DOI: https://doi.org/10.1093/jnci/djt300
- Леонов Г.Е., Вараева Ю.Р., Ливанцова Е.Н., Стародубова А.В. Особенности микробиома ротовой полости при различных соматических заболеваниях. Вопросы питания. 2023;92(4):6–19. Leonov G.E., Varaeva Yu.R., Livantsova E.N., Starodubova A.V. The oral microbiome in the context of systemic disease. Problems of Nutrition. 2023;92(4):6–19. DOI: https://doi.org/10.33029/0042-8833-2023-92-4-6-19
- Johnson J.R., Johnston B., Kuskowski M.A., et al. Molecular epidemiology and phylogenetic distribution of the Escherichia coli pks genomic island. J. Clin. Microbiol. 2008;46(12):3906–11. DOI: https://doi.org/10.1128/JCM.00949-08
- Tsunematsu Y., Hosomi K., Kunisawa J., et al. Mother-to-infant transmission of the carcinogenic colibactin-producing bacteria. BMC Microbiol. 2021;21(1):235. DOI: https://doi.org/10.1186/s12866-021-02292-1
- Iwasaki M., Kanehara R., Yamaji T., et al. Association of Escherichia coli containing polyketide synthase in the gut microbiota with colorectal neoplasia in Japan. Cancer Sci. 2022;113(1):277–86. DOI: https://doi.org/10.1111/cas.15196
Supplementary files