Prevalence of qacEΔ1, qacE, oqxA, oqxB, acrA, cepA and zitB genes among multidrug-resistant Klebsiella pneumoniae isolated in a cardiac hospital
- Authors: Mihailovskaya V.S.1, Selivanova P.A.1, Kuznetsova M.V.1
-
Affiliations:
- Institute of Ecology and Genetics of Microorganisms Ural Branch Russian Academy of Sciences
- Issue: Vol 101, No 4 (2024)
- Pages: 502-511
- Section: ORIGINAL RESEARCHES
- URL: https://microbiol.crie.ru/jour/article/view/18600
- DOI: https://doi.org/10.36233/0372-9311-548
- EDN: https://elibrary.ru/fcoyee
- ID: 18600
Cite item
Full Text
Abstract
Background. Infections caused by multidrug-resistant (MDR) Klebsiella pneumoniae are the leading cause of mortality worldwide. The widespread use of disinfectants and antiseptics has caused the emergence of K. pneumoniae with reduced sensitivity to them, which, in combination with MDR, can pose a significant epidemiological threat.
The aim of the study was to assess the prevalence of efflux pump and transporter genes associated with biocide resistance and their association with antibiotic resistance among K. pneumoniae isolated in a cardiac surgical hospital.
Materials and methods. K. pneumoniae isolates (n = 50) from the patients and medical equipment were tested by polymerase chain reaction for the presence of genes of 4 types of efflux pumps (qacEΔ1, qacE, oqxA, oqxB, acrA) and 2 transporters involved in the outflow of cations (cepA) and zinc ions (zitB). Spearman's rank correlation test was used to assess the strength of the association between the efflux pumps, beta-lactamase genes and mobile genetic elements.
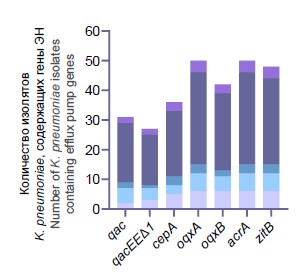
Results. The occurrence of K. pneumoniae containing qacEΔ1, qacE, oqxA, oqxB, acrA, cepA and zitB was high: 54, 62, 100, 84, 100, 72 и 96% respectively. K. pneumoniae with a combination of all the studied pumps was most often detected (32%), and these isolates were MDR in 100% of cases. The qacE, qacEΔ1 genes were closely associated with resistance to cephalosporins, carbapenems, fluoroquinolones, carbapenemase genes, and integrons. The results of the study showed that the genes of various efflux pumps associated with biocide resistance and their combinations were widely represented among the clinical isolates of MDR K. pneumoniae.
Conclusion. The high prevalence of efflux pump genes associated with resistance to quaternary ammonium compounds, chlorhexidine and zinc salts and their significant association with antibiotic resistance in nosocomial K. pneumoniae underlines the importance of further studying the mechanisms of cross-resistance to biocides to improve methods of combating MDR nosocomial pathogens.
Full Text
Введение
Klebsiella pneumoniae является одним из ведущих возбудителей инфекций, связанных с оказанием медицинской помощи в кардиохирургических стационарах. Представители этого вида, согласно классификации Всемирной организации здравоохранения, относятся к группе приоритетных патогенов, поскольку инфекции, вызванные мульти- и панрезистентными K. pneumoniae, ассоциированы с высокой смертностью пациентов [1]. Необходимой частью программы инфекционного контроля и предотвращения внутрибольничных инфекций является обработка поверхностей дезинфектантами и антисептиками [2]. Часто использующиеся в качестве дезинфицирующих средств четвертичные аммониевые соли (ЧАС) обладают способностью прикрепляться к клеточной стенке бактерий благодаря положительному заряду, что вызывает её структурную дезорганизацию и лизис клеток. Хлоргексидин может ковалентно связываться с мембраной, что в конечном итоге приводит к деполяризации и гибели клеток бактерий [3]. Широкое применение дезинфицирующих средств и антисептиков (в том числе в общественных и медицинских учреждениях во время пандемии COVID-19) вызвало возникновение резистентных к ним K. pneumoniae, что в сочетании с множественной лекарственной устойчивостью (МЛУ) может представлять существенную эпидемиологическую угрозу [4, 5].
Одним из механизмов резистентности K. pneumoniae к биоцидам является экспрессия эффлюксных насосов (ЭН). Существуют 6 семейств эффлюксных систем: основное суперсемейство мембранных транспортеров (MFS), суперсемейство оттока лекарственных и токсичных веществ (MATE), суперсемейство АТФ-связывающих бактериальных кассетных транспортеров (ABC), суперсемейство малых транспортеров множественной лекарственной устойчивости (SMR), суперсемейство связывающе-транспортирующих протеинов (RND) и недавно описанная протеобактериальная антимикробная эффлюксная структура (PACE) [6, 7]. AcrAB, OqxAB, EefAB и KexD относятся к белкам суперсемейства RND, которое является наиболее важным у грамотрицательных бактерий. Среди ЭН наибольшую значимость представляют AcrAB и OqxAB [8]. Гены qacE и qacEΔ1 впервые были описаны у K. pneumoniae в 3'-консервативном сегменте интегрона плазмиды R751 [9]. Ген qacEΔ1 представляет собой модифицированную форму qacE, которая возникла в результате вставки сегмента ДНК, содержащего ген устойчивости к сульфаниламиду вблизи 3'-конца гена qacE. упомянутые ЭН относятся к суперсемейству SMR и обусловливают устойчивость к органическим катионам. Известно, что cepA, кодирующий насос оттока катионов, связан с устойчивостью к хлоргексидину у K. pneumoniae [10]. ZitB участвует в конститутивном пути экспорта цинка, способствуя гомеостазу клетки во время воздействия низких и умеренных концентраций ионов металла [11].
Гены группы qac часто выявляются в ассоциации с генами, кодирующими устойчивость к антибиотикам разных групп, в том числе к бета-лактамам [12]. В нескольких работах отмечается, что присутствие насоса AcrAB-TolC способствовало снижению минимальной ингибирующей концентрации ципрофлоксацина [13] и бета-лактамов [4] и формированию МЛУ. Важно, что гены ЭН, связанных с устойчивостью к биоцидам, располагаются не только на хромосоме (emrE, mdfA, sugE, ydgE, ydgF), но и на мобильных генетических элементах (oqxA, oqxB, qacEΔ1, qacE, qacF/H/I, qacG, sugE), таких как плазмиды, интегроны и транспозоны [14]. Более того, гены ЭН могут быть локализованы на тех же мобильных элементах, что и гены устойчивости к антибиотикам, что приводит к перекрестной или ко-резистентности к дезинфектантам, антисептикам и антимикробным препаратам [14].
Ранее мы представили молекулярно-генетическую характеристику изолятов K. pneumoniae, выделенных от пациентов и проб окружающей среды в кардиохирургическом стационаре [15].
Цель исследования — оценка распространённости генов ЭН и транспортёров, ассоциированных с устойчивостью к биоцидам, и их связи с резистентностью к антибиотикам среди изолятов K. pneumoniae, выделенных в условиях кардиохирургического стационара.
Материалы и методы
Бактериальные изоляты
Исследовали 50 изолятов K. pneumoniae, выделенных в 2021–2022 гг. из клинического материала пациентов (мокрота, кровь, моча, содержимое ран) и смывов с медицинского оборудования кардиохирургического стационара. Исследование проводилось при добровольном информированном согласии пациентов. Протокол исследования одобрен Институциональным наблюдательным советом Института экологии и генетики микроорганизмов (протокол № 26, дата утверждения 22.05.2024).
Бактериологические исследования были выполнены на автоматическом анализаторе «WalkAway-96 Plus» («Beckman Сoulter») с использованием панели NBC 41. Чувствительность к антибиотикам (ампициллин, цефотаксим, цефтазидим, цефтриаксон, цефепим, меропенем, имипенем, ампициллин/сульбактам, ампициллин/клавуланат, ципрофлоксацин, левофлоксацин, амикацин, гентамицин) и присутствие генов бета-лактамаз (blaTEM, blaCTX-M, blaSHV, blaOXA, blaKPC, blaVIM-2, blaIMP-1, blaNDM-1) оценивали ранее [15]. Фенотип МЛУ определяли как нечувствительность штаммов хотя бы к одному препарату 3 и более классов антибиотиков.
Детекция генов ЭН и мобильных генетических элементов
ДНК изолятов K. pneumoniae экстрагировали путём прогрева суспензии клеток бактерий в течение 15 мин при 97ºС в твёрдотельном термостате «Термит», пробы охлаждали, центрифугировали 5 мин при 13 000 об/мин, супернатанты переносили в чистые пробирки Эппендорф и хранили при ‒20ºC. Методом ПЦР детектировали гены ЭН, ассоциированных с устойчивостью к ЧАС и хлоргексидину (qacEΔ1, qacE, oqxA, oqxB, acrA), ген транспортёра катионов (cepA), ген zitB, кодирующий транспортёр цинка, ген интегронов класса 1 (int1), а также плазмиды IncQ, распространённой в клинических K. pneumoniae. Праймеры, синтезированные ООО «Cинтол» согласно рекомендациям авторов, условия проведения ПЦР и размеры ампликонов указаны в табл. 1. Амплификацию проводили в 25 мкл реакционной смеси с использованием реагентов производства ООО «Синтол» на термоциклере «DNA Engine Dyad Thermal Cycler» («Bio-Rad»). Электрофоретическое разделение продуктов реакции проводили в 1,2–2,0% агарозном геле в трисборатном буфере при напряжении электрического поля 6 В/см. Визуализацию полос и документирование данных осуществляли с помощью системы гель-документации «Gel-DocXR» («Bio-Rad»).
Таблица 1. Использованные в исследовании последовательности праймеров, условия проведения ПЦР и ожидаемый размер ампликона
Table 1. Primer sequences used in the study, the conditions for PCR and the expected size of the amplicons
Ген Gene | Нуклеотидная последовательность (5’–3’) Nucleotide sequence (5’–3’) | Условия ПЦР PCR conditions | Размер, п.н. Size, bp | Ссылка Reference |
ЭН и транспортёры | Efflux pumps and transporters | ||||
qacEΔ1 | F: TAGCGAGGGCTTTACTAAGC R: ATTCAGAATGCCGAACACCG | 93ºC, 2 m; 35 [93ºC, 30 s; 55ºC, 30 s; 72ºC, 1 m]; 72ºC, 5 m | 300 | [16] |
qacE | F: CCCGAATTCATGAAAGGCTGGCTT R: AAGCTTTCACCATGGCGTCGG | 350 | ||
cepA | F: CAACTCCTTCGCCTATCCCG R: TCAGGTCAGACCAAACGGCG | 94ºC, 5 m; 30 [94ºC, 30 s; 53ºC, 60 s; 72ºC, 2 m]; 72ºC, 7 m | 1051 | |
oqxA | F: CTCGGCGCGATGATGCT R: CCACTCTTCACGGGAGACGA | 95ºC, 1 m; 35 [95ºC, 45 s; 60ºC, 45 s; 72ºC, 1 m]; 72ºC, 5 m | 392 | [17] |
oqxB | F: TTCTCCCCCGGCGGGAAGTAC R: CTCGGCCATTTTGGCGCGTA | 512 | ||
acrA | F: GCTGTCGACGGTTAATGACTTTACAGAGG R: ACATCCGAGAATTCCAGCGT | 94ºC, 3 m; 34 [94ºC, 45 s; 52ºC, 45 s; 68ºC, 1 m]; 72ºC, 5 m | 107 | [18] |
zitB | F: TACGACGCTTCAGTTCAGC R: CACTTTCGGTTGGCTAAGAC | 95ºC, 5 m; 30 [94ºC, 30 s; 53ºC, 60 s; 72ºC, 60 s]; 72ºC, 7 m | 449 | [19] |
Мобильные генетические элементы | Mobile genetic elements | ||||
intI | F: GGCATCCAAGCAGCAAG R: AAGCAGACTTGACCTGA | 94ºC, 5 m; 35 [94ºC, 30 s; 55ºC, 30 s; 72ºC, 2 m]; 72ºC, 5 m | –* | [20] |
IncQ | F: CTCCCGTACTAACTGTCACG R: ATCGACCGAGACAGGCCCTGC | 94ºC, 5 m; 35 [94ºC, 30 s; 61ºC, 30 s; 72ºC, 2 m]; 72ºC, 5 m | 436 | [21] |
Примечание. *Продукт амплификации может быть представлен несколькими последовательностями разного размера.
Note. *The amplification product may be represented by several sequences of different size.
Статистический анализ
Для выявления значимых различий между качественными показателями выборок определяли точный критерий Фишера (двусторонний). Значения p < 0,05 считали достоверными. Тест ранговой корреляции Спирмена использовали для оценки силы ассоциации между генами ЭН и транспортёров, генами бета-лактамаз и мобильных генетических элементов. Сила связи была классифицирована по значению коэффициента rS на очень слабую (0,00–0,19), слабую (0,20–0,39), среднюю (0,40–0,59), сильную (0,60–0,79) и очень сильную (0,80–1,0).
Результаты
Все протестированные гены ЭН встречались с высокой частотой среди изолятов K. pneumoniae, выделенных из биологического материала пациентов и медицинского оборудования кардиохирургического стационара (рис. 1, а). Распространённость генов qacE, qacEΔ1, oqxB и cepA, ассоциированных с устойчивостью к хлоргексидину и ЧАС, оказалась значительной и составила 62% (31/50), 54% (27/50), 84% (42/50) и 72% (36/50) соответственно. Гены oqxA и acrA были обнаружены у всех изолятов. Встречаемость K. pneumoniae, содержащих ген системы выброса ионов цинка (zitB), составила 96% (48/50). В целом в геномах K. pneumoniae было обнаружено 3–7 генов разных ЭН и транспортёров одновременно, причём в исследуемой выборке более половины культур имели 6 и более ЭН (58%, 29/50). Важно отметить, что изоляты с МЛУ содержали больше разных эффлюксных систем, чем чувствительные K. pneumoniae (p < 0,05, t-тест) (рис. 1, б).
Рис. 1. Распространённость K. pneumoniae, содержащих гены ЭН, с учётом источника выделения (а); количество детектированных генов ЭН среди МЛУ и не-МЛУ K. pneumoniae (б). *p < 0,05 (t-тест).
Fig. 1. The prevalence of K. pneumoniae isolates containing efflux pump genes by the source of isolation (a); the number of detected efflux pump genes among multidrug-resistant (MDR) and non-multidrug-resistant (non-MDR) K. pneumoniae (b). *p < 0,05 (t-test).
Детектировано 14 индивидуальных паттернов, среди которых комбинация всех исследуемых генов (qacE + qacEΔ1 + cepA + zitB + oqxA + oqxB + acrA) встречалась наиболее часто (32%, 16/50). Эти 16 культур были в 100% случаев с МЛУ, в частности устойчивы ко всем протестированным бета-лактамным антибиотикам и фторхинолонам. Каждая из последующих комбинаций встречалась с частотой менее 12% (табл. 2).
Таблица 2. Встречаемость индивидуальных комбинаций генов ЭН, ассоциированных с устойчивостью к биоцидам, среди изолятов K. pneumoniae
Table 2. The occurrence of individual combinations of efflux pump genes associated with biocide resistance among K. pneumoniae
Комбинации генов ЭН Efflux pump genes combinations | n (% от всех культур) (% of all isolates) |
qacE + qacEΔ1 + cepA + zitB + oqxB + oqxA + acrA | 16 (32) |
cepA + zitB + oqxB + oqxA + acrA | 6 (12) |
cepA + zitB + oqxA + acrA | 5 (10) |
qacE + qacEΔ1 + zitB + oqxB + oqxA + acrA | 5 (10) |
qacE + cepA + zitB + oqxB + oqxA + acrA | 4 (8) |
zitB + oqxB + oqxA + acrA | 3 (6) |
qacE + zitB + oqxB + oqxA + acrA | 3 (6) |
qacEΔ1 + cepA + zitB + oqxB + oqxA + acrA | 2 (4) |
zitB + oqxA + acrA | 1 (2) |
qacE + qacEΔ1 + cepA + zitB + oqxA + acrA | 1 (2) |
qacE + qacEΔ1 + zitB + oqxA + acrA | 1 (2) |
qacE + qacEΔ1 + cepA + oqxB + oqxA + acrA | 1 (2) |
cepA + oqxB + oqxA + acrA | 1 (2) |
qacEΔ1 + zitB + oqxB + oqxA + acrA | 1 (2) |
В табл. 3 показана ассоциация между генами ЭН и транспортёров и фенотипом резистентности к антибиотикам K. pneumoniae. Гены qacE, qacEΔ1 были тесно связаны с устойчивостью к цефалоспоринам, карбапенемам и фторхинолонам, а oqxB — к цефалоспоринам. Важно отметить, что только K. pneumoniae с МЛУ кодировали qacE и qacEΔ1. Не выявлено значимых ассоциаций генов cepA и zitB с фенотипом устойчивости к антибиотикам.
Таблица 3. Ассоциация между резистентностью к антибиотикам и генами ЭН изолятов K. pneumoniae
Table 3. Association between antibiotic resistance and efflux pump genes of K. pneumoniae isolates
Антибиотик/группа Antibiotic/group | Профиль Profile | Количество изолятов K. pneumoniae, позитивных на гены ЭН, n (%) The number of K. pneumoniae positive for the efflux pump genes, n (%) | ||||
qacEΔ1 | qacE | cepA | oqxB | zitB | ||
Ампициллин Ampicillin | R (n = 50) | 27 (54) | 31 (62) | 36 (72) | 42 (84) | 48 (96) |
S (n = 0) | 0 | 0 | 0 | 0 | 0 | |
Цефалоспорины Cephalosporins | R (n = 45) | 27 (60)* | 31 (68,9)* | 34 (75,6) | 38 (84,4) | 44 (97,8) |
S (n = 5) | 0 | 0 | 2 (40) | 4 (80) | 4 (80) | |
Карбапенемы Carbapenems | R (n = 38) | 27 (71,1)* | 30 (78,9)* | 29 (76,3) | 35 (92,1)* | 37 (97,4) |
S (n = 12) | 0 | 1 (8,3) | 7 (58,3) | 7 (58,3) | 11 (91,7) | |
Ампициллин/сульбактам Ampicillin/sulbactam | R (n = 45) | 27 (60)* | 31 (68,9)* | 34 (75,6) | 38 (84,4) | 44 (97,8) |
S (n = 5) | 0 | 0 | 2 (40) | 4 (80) | 4 (80) | |
Амоксициллин/клавуланат Amoxicillin/clavulanic acid | R (n = 42) | 26 (61,9)* | 29 (69,1)* | 32 (76,2) | 36 (85,7) | 41 (97,6) |
S (n = 8) | 1 (12,5) | 2 (25) | 4 (50) | 6 (75) | 7 (87,5) | |
Фторхинолоны Fluoroquinolones | R (n = 44) | 27 (61,4)* | 31 (70,5)* | 33 (75) | 38 (86,4) | 43 (97,7) |
S (n = 6) | 0 | 0 | 3 (50) | 4 (66,7) | 5 (83,3) | |
Гентамицин Gentamicin | R (n = 29) | 20 (69)* | 21 (72,4) | 19 (65,5) | 25 (86,2) | 28 (96,6) |
S (n = 21) | 7 (33,3) | 10 (47,6) | 17 (81) | 17 (81) | 20 (95,2) | |
Амикацин Amikacin | R (n = 23) | 16 (69,6) | 15 (65,2) | 17 (73,9) | 20 (87) | 22 (95,7) |
S (n = 27) | 11 (40,7) | 16 (59,3) | 19 (70,4) | 22 (81,5) | 26 (96,3) | |
Фенотип МЛУ MDR phenotype | МЛУ | MDR (n = 44) | 27 (61,4)* | 31 (70,5)* | 33 (75) | 38 (86,4) | 43 (97,7) |
не-МЛУ non-MDR (n = 6) | 0 | 0 | 3 (50) | 4 (66,7) | 5 (83,3) | |
Примечание. *p < 0,05 — разница между выборками статистически значима (F-тест).
Note. *p < 0,05 — difference between the samples is statistically significant (F-test).
С помощью корреляционного анализа выявлена позитивная связь между qacEΔ1 и генами бета-лактамаз: blaOXA (rS = 0,31), blaVIM-2 (rS = 0,68), blaNDM-1 (rS = 0,64), blaCTX-M (rS = 0,51), blaSHV (rS = 0,51). Эти же гены показали значимую связь с qacE (рис. 2). В нашем исследовании гены cepA и zitB не показали позитивных корреляций с генами устойчивости к антибиотикам. Интегроны класса 1 длиной от 800 п.н. до 2500 п.н. были выявлены у 32 (64%) изолятов. Плазмида группы несовместимости Q была обнаружена у 90% (45/50) K. pneumoniae. Корреляционный анализ показал значимую позитивную связь между генами насосов qacE, qacEΔ1 и интегронами (rS = 0,70 и rS = 0,65; рис. 2).
Рис. 2. Корреляционная матрица, отражающая силу связи между генами ЭН, генами бета-лактамаз и мобильными генетическими элементами. Цветовое значение каждой ячейки соответствует коэффициенту корреляции Спирмена и пропорционально силе корреляции. *p < 0,05, **p < 0,01. Цветной вариант рисунка см. на сайте журнала.
Fig. 2. A correlation matrix reflecting the strength of the relationship between the efflux pumps genes, beta-lactamase genes and mobile genetic elements. The color of each cell corresponds to the Spearman correlation coefficient and is proportional to the correlation strength. *p < 0.05, **p < 0.01. For a color version of the picture, see the journal’s website.
Обсуждение
K. pneumoniae — распространённый патоген, вызывающий инфекции, связанные с оказанием медицинской помощи. МЛУ этих бактерий к антибиотикам первой линии существенно затрудняет лечение. К тому же широкое использование биоцидов (особенно в сублетальных концентрациях) может вызвать резистентность к ним и, как следствие, способствовать сохранению K. pneumoniae в окружающей среде, в том числе на поверхностях медицинских приборов. Сниженная чувствительность бактерий к ЧАС, хлоргексидину и солям цинка в высокой степени связана с экспрессией ЭН. Во многих исследованиях показана важность ЭН для повышения устойчивости штаммов K. pneumoniae и к различным классам антибиотиков [22–24]. Важно отметить, что гены устойчивости к противомикробным препаратам часто находятся на мобильных генетических элементах, которые могут передаваться путём горизонтального переноса от одного штамма к другому. В данной работе у наиболее значимого нозокомиального патогена — K. pneumoniae мы изучили распространённость ЭН, связанных с устойчивостью к ЧАС и хлоргексидину, транспортёров, ассоциированных со сниженной чувствительностью к солям цинка, а также их связь с резистентностью к антибиотикам, наличием генов бета-лактамаз и мобильными генетическими элементами.
Одним из механизмов устойчивости к биоцидам грамотрицательных бактерий является экспрессия эффлюксных систем семейства SMR, которые кодируются генами qacE и qacEΔ1 [25, 26]. С устойчивостью K. pneumoniae к хлоргексидину тесно связан cepA, кодирующий белки системы оттока катионов [27]. В выборочных исследованиях по изучению резистентности клинических штаммов K. pneumoniae к бензалкония хлориду ген qacΔE1 обнаруживался в диапазоне от 53,1 до 68,0% [28, 29]. Согласно А. Abuzaid и соавт., среди K. pneumoniae ген cepA встречался в 87,5% случаев [28]. В исследовании К.Г. Косяковой и соавт. частота встречаемости qacE, qacEΔ1 и cepA составила 33,3, 23,3 и 83,3% соответственно [25]. В нашем исследовании qacEΔ1 и qacE были распространены в группе нозокомиальных K. pneumoniae с более высокой частотой: 62 и 54% соответственно. Встречаемость cepA оказалась несколько ниже — 72%. В последнее время возрастает роль OqxA- и OqxB-насосов, входящих в семейство связывающе-транспортирующих белков [22]. Ранее J. Yuan и соавт. обнаружили гены oqxAB во всех исследованных штаммах K. pneumoniae, что позволило сделать предположение о геноме K. pneumoniae как возможном резервуаре oqxAB [32]. У клебсиелл в основном эти гены расположены в хромосоме, но могут находиться на плазмидах и часто ассоциированы с устойчивостью к фторхинолонам, тигециклину, а также ЧАС и бигуанидным дезинфицирующим средствам [30, 31]. Меньший процент встречаемости данных детерминант показали M. Dehnamaki и соавт.: 57 и 56% изолятов несли гены oqxA и oqxB соответственно [17]. В исследовании L. Ni и соавт. частоты обнаружения oqxA и oqxB составили 60,9 и 17,2% [24]. В нашем исследовании ген oqxA детектирован у всех изолятов кардиохирургического стационара, тогда как oqxB содержали 84% культур. ЭН AcrAB-TolC обеспечивает толерантность бактерий к различным антибиотикам, включая фторхинолоны, а также к биоцидам, включая этанол, хлоргексидина ацетат и бензалкония бромид [33]. Помимо вклада в фенотип МЛУ, AcrAB может представлять собой новый фактор вирулентности, необходимый K. pneumoniae для сопротивления иммунным защитным механизмам в лёгких, способствуя развитию пневмонии [34]. Ген acrA, кодирующий белок, соединяющий 2 интегральных мембранных белка, считается более консервативным, и его часто используют для обнаружения данного ЭН. В нашем исследовании все изоляты содержали acrA, тогда как, по данным W. Guo и соавт. [33], только 19% резистентных к карбапенемам K. pneumoniae включали данный ген. Важно отметить, что соли и наночастицы тяжёлых металлов активно используются в антисептических композициях. Сегодня ведётся активная разработка противомикробных поверхностных веществ на основе меди, серебра и цинка для создания бинарных или тройных контактно-нейтрализующих поверхностных покрытий [35]. Было доказано, что ZnO более токсичен для бактерий в форме наночастиц, что делает его более перспективным. В данной работе распространённость культур, кодирующих транспортёр, опосредующий отток ионов цинка [36], — ZitB, составила 96%.
В нескольких работах отмечается, что резистентность к антибиотикам и биоцидам может многократно увеличиваться за счёт одновременной экспрессии ЭН разных семейств [4, 19, 22]. Важно отметить, что в исследуемой выборке наиболее часто встречались K. pneumoniae с комбинацией всех исследуемых насосов (32%), причём такие культуры были в 100% случаев с МЛУ. Ранее мы показали высокую распространённость в изолятах кардиохирургического стационара blaOXA, blaVIM-2 и blaNDM-1. В этом исследовании выявлено, что гены группы qac чаще встречались в ассоциации с генами, кодирующими устойчивость к карбапенемам, — blaVIM-2 (rS = 0,68) и blaNDM-1 (rS = 0,64), что может быть обусловлено их локализацией в плазмид-опосредованных интегронах класса 1 и требует дальнейшего изучения. В исследовании Y. Chen и соавт. также было показано, что гены qacEΔ1 и cepA были достоверно чаще представлены среди карбапенеморезистентных культур [16]. Глобальное распространение детерминант устойчивости к бета-лактамным антибиотикам, в том числе карбапенемам, является результатом их распространения посредством конъюгативных плазмид. Известны 5 групп несовместимости плазмид (Inc), которые описаны в литературе как потенциально ответственные за распространение генов blaKPC и blaNDM у изолятов K. pneumoniae [21]. Другие мобильные генетические элементы — интегроны — могут включать кассеты устойчивости к противомикробным препаратам и дезинфектантам [37]. По данным F. Firoozeh и соавт., 100% изолятов K. pneumoniae (n = 150) с МЛУ несли гены intI [38]. В исследовании É.M. Oliveira и соавт. у клинических изолятов K. pneumoniae в госпитале г. Ресифи (Бразилия) наиболее часто обнаруживались плазмиды групп несовместимости Q и FIB [21]. Данные, полученные W.M.B.S. Martins и соавт., свидетельствуют о том, что K. pneumoniae с высококопийной плазмидой IncQ, несущей blaKPC-2, может эффективно передавать её путём конъюгативного переноса другим штаммам [39]. Плазмида IncQ, способная реплицироваться в широком круге хозяев, была обнаружена в клиническом изоляте K. pneumoniae, выделенном от пациентов с COVID-19 в России [40]. В нашем исследовании интегроны класса 1 были детектированы у 64% K. pneumoniae, а плазмиды IncQ — у 90%. При этом выявлена значимая позитивная связь между генами ЭН qacE, qacEΔ1 и интегронами.
Заключение
Проведено исследование по распространённости генов ЭН, а также транспортёров, участвующих в оттоке ионов металлов, оценена их связь с антибиотикоустойчивостью и генами, ассоциированными с резистентностью к бета-лактамам, среди культур K. pneumoniae с МЛУ, выделенных от пациентов кардиохирургического стационара с нозокомиальной инфекцией. Показана значимая связь между устойчивостью к антибиотикам и присутствием генов ЭН, ассоциированных с устойчивостью к ЧАС, хлоргексидину и ионам цинка, что подчёркивает важность дальнейшего изучения механизмов кросс-резистентности к биоцидам для совершенствования методов борьбы с патогенами с МЛУ.
About the authors
Veronika S. Mihailovskaya
Institute of Ecology and Genetics of Microorganisms Ural Branch Russian Academy of Sciences
Email: mar@iegm.ru
ORCID iD: 0000-0002-4264-8177
engineer, Laboratory of molecular biotechnology
Russian Federation, PermPolina A. Selivanova
Institute of Ecology and Genetics of Microorganisms Ural Branch Russian Academy of Sciences
Email: mar@iegm.ru
ORCID iD: 0009-0009-3718-8602
engineer, Laboratory of molecular biotechnology
Russian Federation, PermMarina V. Kuznetsova
Institute of Ecology and Genetics of Microorganisms Ural Branch Russian Academy of Sciences
Author for correspondence.
Email: mar@iegm.ru
ORCID iD: 0000-0003-2448-4823
D. Sci. (Med.), leading researcher, Laboratory of molecular biotechnology
Russian Federation, PermReferences
- Ntshonga P., Gobe I., Koto G., et al. Biocide resistance in Klebsiella pneumoniae: a narrative review. Infect. Prev. Pract. 2024;6(2):100360. DOI: https://doi.org/10.1016/j.infpip.2024.100360
- Gerba C.P. Quaternary ammonium biocides: efficacy in application. Appl. Environ. Microbiol. 2015;81(2):464–9. DOI: https://doi.org/10.1128/AEM.02633-14
- Зверьков А.В., Зузова А.П. Хлоргексидин: прошлое, настоящее и будущее одного из основных антисептиков. Клиническая микробиология и антимикробная химиотерапия. 2013;15(4):279–85. Zverkov A.V., Zouzova A.P. Chlorhexidine: past, present, and future of the famous antiseptic agent. Clinical Microbiology and Antimicrobial Chemotherapy. 2013;15(4):279–85. EDN: https://elibrary.ru/roxohj
- Maurya N., Jangra M., Tambat R., Nandanwar H. Alliance of efflux pumps with β-lactamases in multidrug-resistant Klebsiella pneumoniae isolates. Microb. Drug Resist. 2019;25(8):1155–63. DOI: https://doi.org/10.1089/mdr.2018.0414
- Chen B., Han J., Dai H., Jia P. Biocide-tolerance and antibiotic-resistance in community environments and risk of direct transfers to humans: Unintended consequences of community-wide surface disinfecting during COVID-19? Environ. Pollut. 2021;283:117074. DOI: https://doi.org/10.1016/j.envpol.2021.117074
- Иванов М.Э., Фурсова Н.К., Потапов В.Д. Суперсемейства эффлюксных насосов Pseudomonas aeruginosa (обзор литературы). Клиническая лабораторная диагностика. 2022;67(1):53–8. Ivanov M.E., Fursova N.K., Potapov V.D. Pseudomonas aeruginosa efflux pump superfamily (review of literature). Clinical Laboratory Diagnostics. 2022;67(1):53–8. DOI: https://doi.org/10.51620/0869-2084-2022-67-1-53-58 EDN: https://elibrary.ru/tkfgmf
- Ковальчук С.Н., Федорова Л.С., Ильина Е.Н. Молекулярные механизмы микробной устойчивости к дезинфицирующим средствам. Антибиотики и химиотерапия. 2023;68(1-2): 45–56. Kovalchuk S.N., Fedorova L.S., Ilina E.N. Molecular mechanisms of microbial resistance to disinfectants. Antibiotics and Chemotherapy. 2023;68(1-2):45-56. DOI: https://doi.org/10.37489/0235-2990-2023-68-1-2-45-56 EDN: https://elibrary.ru/hycybo
- Ntshonga P., Gobe I., Koto G., et al. Biocide resistance in Klebsiella pneumoniae: a narrative review. Infect. Prev. Pract. 2024;6(2):100360. DOI: https://doi.org/10.1016/j.infpip.2024.100360
- Stokes H.W., Hall R.M. A novel family of potentially mobile DNA elements encoding site-specific gene-integration functions: integrons. Mol. Microbiol. 1989;3(12):1669–83. DOI: https://doi.org/10.1111/j.1365-2958.1989.tb00153.x
- Fang C.T., Chen H.C., Chuang Y.P., et al. Cloning of a cation efflux pump gene associated with chlorhexidine resistance in Klebsiella pneumoniae. Antimicrob. Agents Chemother. 2002;46(6):2024–8. DOI: https://doi.org/10.1128/AAC.46.6.2024-2028.2002
- Wang D., Hosteen O., Fierke C.A. ZntR-mediated transcription of zntA responds to nanomolar intracellular free zinc. J. Inorg. Biochem. 2012;111:173–81. DOI: https://doi.org/10.1016/j.jinorgbio.2012.02.008
- Kücken D., Feucht H., Kaulfers P.M. Association of qacE and qacEΔ1 with multiple resistance to antibiotics and antiseptics in clinical isolates of Gram-negative bacteria. FEMS Microbiol. Lett. 2000;183(1):95–8. DOI: https://doi.org/10.1111/j.1574-6968.2000.tb08939.x
- Pakzad I., Zayyen Karin M., Taherikalani M., et al. Contribution of AcrAB efflux pump to ciprofloxacin resistance in Klebsiella pneumoniae isolated from burn patients. GMS Hyg. Infect. Control. 2013;8(2):Doc15. DOI: https://doi.org/10.3205/dgkh000215
- Hrovat K., Zupančič J.Č., Seme K., Avguštin J.A. QAC resistance genes in ESBL-producing E. coli isolated from patients with lower respiratory tract infections in the central Slovenia region — a 21-year survey. Trop. Med. Infect. Dis. 2023;8(5):273. DOI: https://doi.org/10.3390/tropicalmed8050273
- Кузнецова М.В., Сергевнин В.И., Михайловская В.С. и др. Микробиологическая и молекулярно-генетическая характеристика изолятов Klebsiella pneumoniae, выделенных в условиях кардиохирургического стационара. Инфекция и иммунитет. 2024;14(1):103–14. Kuznetsova M.V., Sergevnin V.I., Mikhailovskaya V.S., et al. Microbiological and molecular genetic characteristics of Klebsiella pneumoniae isolates, extracted under conditions of cardiac surgery hospital. Russian Journal of Infection and Immunity. 2024;14(1):103–14. DOI: https://doi.org/10.15789/2220-7619-MAM-15631 EDN: https://elibrary.ru/dwusky
- Chen Y., Liao K., Huang Y., et al. Determining the susceptibility of carbapenem resistant Klebsiella pneumoniae and Escherichia coli strains against common disinfectants at a tertiary hospital in China. BMC Infect. Dis. 2020;20(1):88. DOI: https://doi.org/10.1186/s12879-020-4813-6
- Dehnamaki M., Ghane M., Babaeekhou L. Detection of OqxAB and QepA efflux pumps and their association with antibiotic resistance in Klebsiella pneumoniae isolated from urinary tract infection. Int. J. Infect. 2020;7(4):e107397. DOI: https://doi.org/10.5812/iji.107397
- Li D.W., Onishi M., Kishino T., et al. Properties and expression of a multidrug efflux pump AcrAB-KocC from Klebsiella pneumoniae. Biol. Pharm. Bull. 2008;31(4):577–82. DOI: https://doi.org/10.1248/bpb.31.577
- Deus D., Krischek C., Pfeifer Y., et al. Comparative analysis of the susceptibility to biocides and heavy metals of extended-spectrum β-lactamase-producing Escherichia coli isolates of human and avian origin, Germany. Diagn. Microbiol. Infect. Dis. 2017;88(1):88–92. DOI: https://doi.org/10.1016/j.diagmicrobio.2017.01.023
- Lévesque C., Piché L., Larose C., Roy P.H. PCR mapping of integrons reveals several novel combinations of resistance genes. Antimicrob. Agents Chemother. 1995;39(1):185–91. DOI: https://doi.org/10.1128/aac.39.1.185
- Oliveira É.M., Beltrão E.M.B., Scavuzzi A.M.L., et al. High plasmid variability, and the presence of IncFIB, IncQ, IncA/C, IncHI1B, and IncL/M in clinical isolates of Klebsiella pneumoniae with blaKPC and blaNDM from patients at a public hospital in Brazil. Rev. Soc. Bras. Med. Trop. 2020;53:e20200397. DOI: https://doi.org/10.1590/0037-8682-0397-2020
- Ni R.T., Onishi M., Mizusawa M., et al. The role of RND-type efflux pumps in multidrug-resistant mutants of Klebsiella pneumoniae. Sci. Rep. 2020;10(1):10876. DOI: https://doi.org/10.1038/s41598-020-67820-x
- Gaurav A., Bakht P., Saini M., et al. Role of bacterial efflux pumps in antibiotic resistance, virulence, and strategies to discover novel efflux pump inhibitors. Microbiology (Reading). 2023;169(5):001333. DOI: https://doi.org/10.1099/mic.0.001333
- Ni L., Zhang Z., Shen R., et al. Disinfection strategies for carbapenem-resistant Klebsiella pneumoniae in a healthcare facility. Antibiotics (Basel). 2022;11(6):736. DOI: https://doi.org/10.3390/antibiotics11060736
- Косякова К.Г., Эсауленко Н.Б., Каменева О.А. и др. Распространенность генов карбапенемаз, qacE, qacEΔ1 и cepA у множественно-резистентных грамотрицательных бактерий с различной чувствительностью к хлоргексидину. Эпидемиология и вакцинопрофилактика. 2020;19(5):49–60. Kosyakova K.G., Esaulenko N.B., Kameneva O.A., et al. Prevalence of carbapenemase genes, qacE, qacEΔ1 and cepA in multidrug-resistant gram-negative bacteria with different susceptibility to chlorhexidine. Epidemiology and Vaccinal Prevention. 2020;19(5):49–60. DOI: https://doi.org/10.31631/2073-3046-2020-19-5-49-60 EDN: https://elibrary.ru/avfflm
- Kazama H., Hamashima H., Sasatsu M., Arai T. Distribution of the antiseptic-resistance genes qacE and qacE delta 1 in gram-negative bacteria. FEMS Microbiol. Lett. 1998;159(2):173–8. DOI: https://doi.org/10.1111/j.1574-6968.1998.tb12857.x
- Fang C.T., Chen H.C., Chuang Y.P., et al. Cloning of a cation efflux pump gene associated with chlorhexidine resistance in Klebsiella pneumoniae. Antimicrob. Agents Chemother. 2002;46(6):2024–8. DOI: https://doi.org/10.1128/AAC.46.6.2024-2028.2002
- Abuzaid A., Hamouda A., Amyes S. Klebsiella pneumoniae susceptibility to biocides and its association with cepA, qacΔE and qacE efflux pump genes and antibiotic resistance. J. Hosp. Infect. 2012;81(2):87–91. DOI: https://doi.org/10.1016/j.jhin.2012.03.003
- Pastrana-Carrasco J., Garza-Ramos J.U., Barrios H., et al. Pastrana-Carrasco J., Garza-Ramos J.U., Barrios H., et al. QacEdelta1 gene frequency and biocide resistance in extended-spectrum beta-lactamase producing enterobacteriaceae clinical isolates. Rev. Invest. Clin. 2012;64(6 Pt. 1):535–40.
- Zhong X., Xu H., Chen D., et al. First emergence of acrAB and oqxAB mediated tigecycline resistance in clinical isolates of Klebsiella pneumoniae pre-dating the use of tigecycline in a Chinese hospital. PLoS One. 2014;9(12):e115185. DOI: https://doi.org/10.1371/journal.pone.0115185
- Li J., Zhang H., Ning J., et al. The nature and epidemiology of OqxAB, a multidrug efflux pump. Antimicrob. Resist. Infect. Control. 2019;8:44. DOI: https://doi.org/10.1186/s13756-019-0489-3
- Yuan J., Xu X., Guo Q., et al. Prevalence of the oqxAB gene complex in Klebsiella pneumoniae and Escherichia coli clinical isolates. J. Antimicrob. Chemother. 2012;67(7):1655–9. DOI: https://doi.org/10.1093/jac/dks086
- Guo W., Shan K., Xu B., Li J. Determining the resistance of carbapenem-resistant Klebsiella pneumoniae to common disinfectants and elucidating the underlying resistance mechanisms. Pathog. Glob. Health. 2015;109(4):184–92. DOI: https://doi.org/10.1179/2047773215Y.0000000022
- Padilla E., Llobet E., Doménech-Sánchez A., et al. Klebsiella pneumoniae AcrAB efflux pump contributes to antimicrobial resistance and virulence. Antimicrob. Agents Chemother. 2010;54(1):177–83. DOI: https://doi.org/10.1128/AAC.00715-09
- Birkett M., Dover L., Cherian Lukose C., et al. Recent advances in metal-based antimicrobial coatings for high-touch surfaces. Int. J. Mol. Sci. 2022;23(3):1162. DOI: https://doi.org/10.3390/ijms23031162
- Grass G., Fan B., Rosen B.P., et al. ZitB (YbgR), a member of the cation diffusion facilitator family, is an additional zinc transporter in Escherichia coli. J. Bacteriol. 2001;183(15):4664–7. DOI: https://doi.org/10.1128/jb.183.15.4664-4667.2001
- Karampatakis T., Tsergouli K., Behzadi P. Carbapenem-resistant Klebsiella pneumoniae: virulence factors, molecular epidemiology and latest updates in treatment options. Antibiotics (Basel). 2023;12(2):234. DOI: https://doi.org/10.3390/antibiotics12020234
- Firoozeh F., Mahluji Z., Khorshidi A., Zibaei M. Molecular characterization of class 1, 2 and 3 integrons in clinical multi-drug resistant Klebsiella pneumoniae isolates. J. Antimicrob. Res. Inf. Control. 2019;8:59. DOI: https://doi.org/10.1186/s13756-019-0509-3
- Martins W.M.B.S., Nicolas M.F., Yu Y., et al. Clinical and molecular description of a high-copy IncQ1 KPC-2 plasmid harbored by the international ST15 Klebsiella pneumoniae. Clone. mSphere. 2020;5(5):e00756-20. DOI: https://doi.org/10.1128/mSphere.00756-20.
- Shelenkov A., Petrova L., Mironova A., et al. Long-read whole genome sequencing elucidates the mechanisms of amikacin resistance in multidrug-resistant Klebsiella pneumoniae isolates obtained from COVID-19 patients. Antibiotics. 2022;11:1364. DOI: https://doi.org/10.3390/antibiotics11101364
Supplementary files