Molecular epidemiological study of clinical cases of acute hepatitis E in Belarus
- Authors: Davydov V.V.1, Zhavoronok S.V.1, Znovets T.V.2, Tsyrkunov V.M.3, Babenkа A.S.1, Marchuk S.I.1, Gasich E.L.4, Novik T.P.1, Alatortseva G.I.5, Mikhailov M.I.5,6, Zverev V.V.5,7
-
Affiliations:
- Belarusian State Medical University
- Third City Clinical Hospital named after E.V. Klumov
- Grodno State Medical University
- Republican Scientific and Practical Center of Epidemiology and Microbiology
- I. Mechnikov Research Institute of Vaccines and Sera
- Russian Medical Academy of Continuous Professional Education
- I.M. Sechenov First Moscow State Medical University (Sechenov University)
- Issue: Vol 99, No 6 (2022)
- Pages: 625-636
- Section: ORIGINAL RESEARCHES
- URL: https://microbiol.crie.ru/jour/article/view/1355
- DOI: https://doi.org/10.36233/0372-9311-328
- ID: 1355
Cite item
Full Text
Abstract
Relevance. The frequency of occurrence of anamnestic antibodies to the hepatitis E virus (HEV) in the general population of the Republic of Belarus is 7.3%, which is clearly not consistent with the low incidence of hepatitis E (HE). Most of primary HEV infections remain undiagnosed. The intensive epidemic process of HEV in the Belarusian population is hidden. Conducting epidemiological studies, including genotyping of HEV sequences isolated on the territory of the republic, makes it possible to more accurately characterize the sources of HEV infection and the mechanisms of its transmission.
Aim — molecular epidemiological study of two cases of acute hepatitis E detected in patients from Belarus.
Materials and methods. During 2021–2022, samples of biological material were obtained from two patients undergoing treatment with an established diagnosis of acute hepatitis E. Serum samples were tested to detect antibodies to HEV using enzyme immunoassay, HEV RNA was detected in fecal samples using nested RT-PCR. The nucleotide sequence was determined by an automatic sequencer using the Sanger method. Analysis of nucleotide sequences, their genotyping, and calculation of evolutionary distances were performed using MEGA X software.
Results. The HEV sequence isolated from a pregnant woman who had an epidemiological episode of alimentary contact with raw pork meat is clustered into a common phylogenetic clade with HEV sequence obtained from the patient from Belarus with a history of kidney transplantation and HEV sequences isolated from a domestic pigs. The HEV sequence isolated from a patient with a history of travel to Pakistan belongs to the HEV genotype 1 and joins a clade of HEV sequences isolated in Pakistan, India, Nepal and Mongolia.
Full Text
Введение
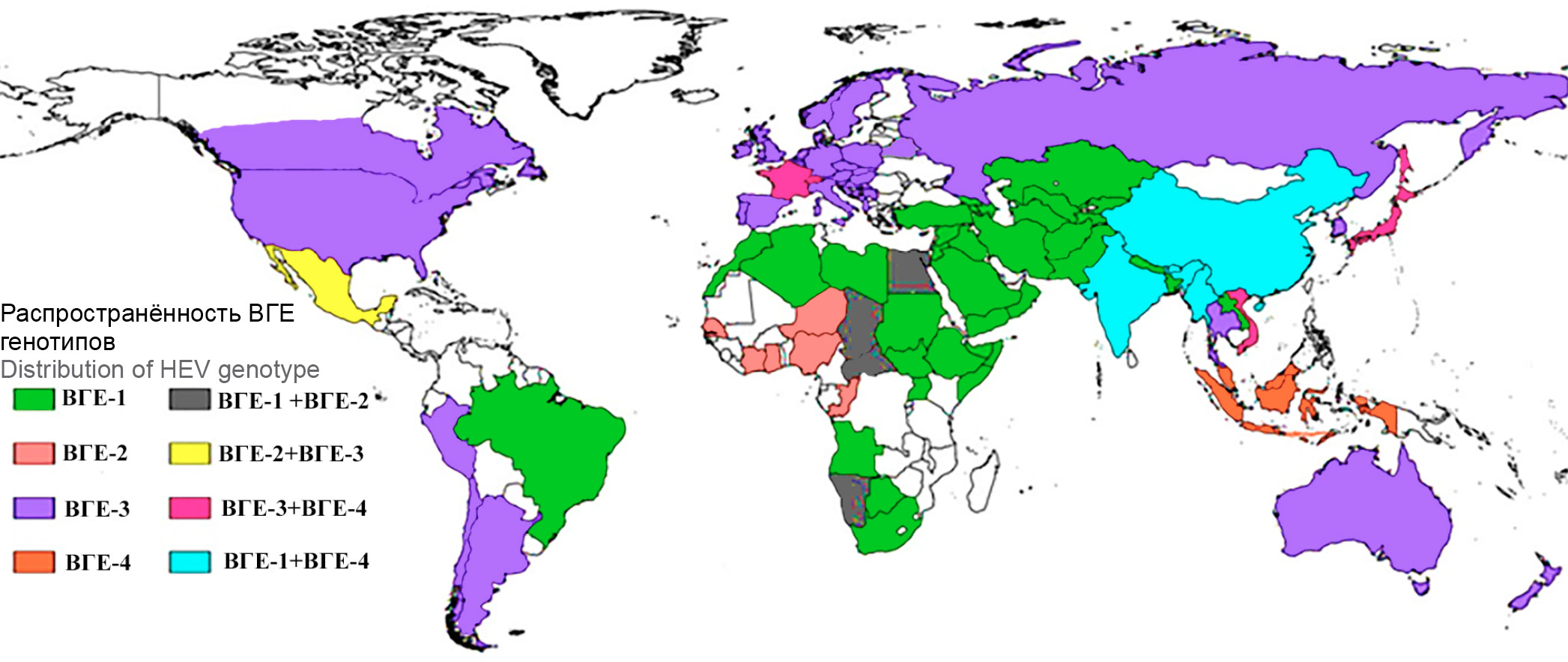
Вирус гепатита Е (ВГЕ) (Paslahepevirus balayani), названный в честь первооткрывателя вируса — советского вирусолога М.С. Балаяна [1], относится к семейству Hepeviridae рода Paslahepevirus. Префикс «Pasla» является аббревиатурой и происходит от первых букв латинских названий таксонов известных хозяев вируса: Primates, Artiodactyla, Scandentia, Lagomorpha [2]. Корень «hepe» в родовом названии показывает тропность вируса, который в основном способен к репликации в клетках печени. Это РНК-содержащий вирус, вызывающий острый вирусный гепатит у человека. На основе филогенетического анализа выделяют 8 генотипов вируса (ВГЕ-1–ВГЕ-8). Известно, что только 5 генотипов ВГЕ из 8 известных могут инфицировать человека. Генотипы 1 и 2 имеют фекально-оральный механизм передачи и вызывают крупные вспышки заболеваний в развивающихся странах. Генотипы ВГЕ-3 и ВГЕ-4 вызывают зоонозную инфекцию, передающуюся человеку от заражённых животных: свиней, оленей и диких кабанов. Передача обычно происходит при употреблении в пищу сырого или недостаточно термически обработанного мяса или печени этих животных. ВГЕ-5 и ВГЕ-6 до сих пор были обнаружены только у диких кабанов в Японии. ВГЕ-7 и ВГЕ-8 недавно были найдены у верблюдов на Ближнем Востоке и в Китае. Из этих 4 генотипов было описано только одно заражение человека ВГЕ-7, связанное с употреблением заражённого верблюжьего мяса и молока [3] пациентом с ослабленным иммунитетом после трансплантации. В странах Европы наиболее часто гепатит Е (ГЕ) вызывается генотипом ВГЕ-3. Глобальная распространённость доминирующих генотипов ВГЕ представлена на рис. 1.
Рис. 1. Глобальное распределение основных генотипов ВГЕ. Различные цвета на карте указывают на распределение генотипов ВГЕ (ВГЕ-1–ВГЕ-4) по всему миру [4]. / Fig. 1. Global distribution of major HEV genotypes. The different colors on the map indicate the distribution of HEV genotypes (HEV-1 to HEV-4) around the world [4].
ВГЕ является гепатотропным инфекционным агентом, который обычно вызывает самокупирующийся острый гепатит у здоровых взрослых и может вызывать хронический гепатит у лиц с ослабленным иммунитетом [5]. По оценкам ВОЗ, ежегодно происходит более 20 млн случаев инфицирования, что приводит примерно к 3 млн симптоматических случаев ГЕ и около 70 000 смертей, связанных с ним1. Истинное глобальное бремя данного заболевания, вероятно, является ещё бóльшим [6]. Исследования распространённости антител к ВГЕ у здоровых людей, проведённые в различных странах, показывают, что до половины населения может иметь анамнестические антитела против ВГЕ [7]. Однако у беременных женщин острый ГЕ может быть очень тяжёлым и приводить в 30% случаев к летальному исходу в случае заражения в III триместре [8]. Кроме того, инфицирование ВГЕ во время беременности часто приводит к преждевременным родам, вертикальной передаче, значительной перинатальной заболеваемости и мертворождениям [9]. Эта проблема осложняется невозможностью применения рибавирина и интерферона I типа из-за риска тератогенного действия, поэтому беременным женщинам с ГЕ обычно назначается только поддерживающая терапия [10].
Механизмы тяжёлого поражения печени при инфицировании ВГЕ у беременных точно неизвестны. Развитие фульминантного гепатита может быть обусловлено изменённым иммунитетом, высоким уровнем гормонов и факторами, связанными с самим вирусом. Факторы, связанные с вирусом, включают варианты генотипа ВГЕ и его белки, например малый многофункциональный белок, кодируемый третьей открытой рамкой считывания вирусного генома. Иммунные факторы организма-хозяина, такие как CD8+-Т-, NK- и NKT-клетки, могут быть вовлечены в патогенез ГЕ во время беременности. Кроме того, в этот процесс могут быть вовлечены некоторые цитокины, такие как фактор некроза опухоли-α, интерлейкин-6, интерферон-γ и трансформирующий фактор роста-β1. Резко повышенные уровни гормонов, включая прогестерон, эстроген и хорионический гонадотропин, во время беременности также могут способствовать повреждению печени [11]. Кроме того, частый неблагоприятный исход ГЕ у беременных, вероятно, связан с возможностью репликации ВГЕ в мезенхимальных стромальных клетках эндометрия [12].
ВГЕ представляет собой РНК-содержащий вирус с капсидом с икосаэдрической структурой размером 27–34 нм. ВГЕ существует в виде квазиоболочечных вирусных частиц в крови и супернатанте клеточных культур и в виде безоболочечных вирионов в желчи и фекалиях [13]. Инфицирование организма-хозяина происходит алиментарно через заражённую пищу или воду. При попадании в желудочно-кишечный тракт вирионы сначала инфицируют энтероциты, где они размножаются, выделяются в виде квазиоболочечных вирусных частиц в кровоток и инфицируют гепатоциты [14]. При выходе из гепатоцитов в результате контакта с желчью оболочка «срывается», вирус обнажается и более контагиозные вирионы снова выделяются через фекалии.
Геном ВГЕ имеет размер 7,2 kb и представляет собой одноцепочечную смысловую РНК, содержащую в большинстве случаев 3 открытые рамки считывания (ОРС) [15]. ОРС1 обеспечивает синтез неструктурного полипротеина, необходимого для репликации вируса. ОРС2 образует субгеномную бицистронную РНК размером 2,2 kb и кодирует белок (pORF2), состоящий из 660 а.о. Белок pORF2 процессируется в три формы: две из них секретируются из поражённой клетки в виде гликопротеинов, которые не связаны с инфекционными частицами, и третью, негликозилированную форму, которая является структурным компонентом инфекционных частиц [16]. ОРС3 является наименьшей из трех и перекрывается с ОРС2 примерно на 300 нуклеотидов в альтернативной рамке считывания [17]. Существует также ОРС4, перекрывающаяся с ОРС1, она присутствует только в геноме ВГЕ-1 [18]. ОРС4 кодирует белок (pORF4) из 124 а.о., который усиливает активность вирусной полимеразы, способствует репликации вируса и необходим для жизненного цикла ВГЕ-1. Вероятно данная особенность определяет эпидемический характер распространения ВГЕ 1-го генотипа.
Процесс систематики генетического разнообразия ВГЕ имеет ряд трудностей. В нашей работе мы придерживались подходов, описанных D.B. Smith и соавт. [19]. Систематика субгенотипов ВГЕ основана на изучении различий эволюционного расстояния (p-distance) последовательностей генома вируса и их филогенетического положения. Эволюционные расстояния нуклеотидных последовательностей среди полных геномных последовательностей ВГЕ-1 формируют непрерывное распределение до 0,122. Расстояния внутри субгенотипов колеблются до 0,06, перекрывая диапазон субгенотиповых расстояний (> 0,037), включая расстояния 0,056–0,064 между субгенотипами 1b и 1c и 0,044–0,082 между субгенотипами 1a и 1f. Перекрытие диапазонов меж- и внутрисубгенотиповых сравнений является ещё более выраженным для расстояний, рассчитанных для аминокислотных последовательностей. Систематика субгенотипов ВГЕ-3 демонстрирует ещё более сложный паттерн с формированием разных филогенетических групп. Так, субгенотипы 3a, 3b, 3c, 3h, 3i и 3j и субгенотипы 3e, 3f и 3g образуют две главные филогенетические клады 3abchij и 3efg. Третью кладу формируют субгенотипы кроличьего ВГЕ-3.
Целью настоящего исследования явилось молекулярно-эпидемиологическое исследование двух случаев острого ГЕ, выявленных у пациентов из Беларуси.
Материалы и методы
В 2021–2022 гг. были получены образцы биологического материала от двух пациентов, проходящих лечение в учреждениях здравоохранения Республики Беларусь, с установленным диагнозом острого ГЕ. Предварительно пациенты дали добровольное информированное согласие для участия в исследовании. Протокол исследования одобрен Комитетом по биомедицинской этике Белорусского государственного медицинского университета (протокол № 8 от 26.04.2016).
Полученные образцы тестировали на присутствие специфических иммуноглобулинов классов M (анти-ВГЕ IgM) и G (анти-ВГЕ IgG) при помощи иммуноферментного анализа и на присутствие РНК ВГЕ при помощи ПЦР-анализа. Анти-ВГЕ IgG и анти-ВГЕ IgM определяли с использованием наборов реагентов «ДС-ИФА-АНТИ-HEV-G» и «ДС-ИФА-АНТИ-HEV-M» (НПО «Диагностические системы») согласно инструкциям производителя. Использовали также разработанные нами новые тест-системы для выявления анти-ВГЕ IgG и анти-ВГЕ IgM в сыворотке крови, основанные на непрямом варианте иммуноферментного анализа [20] и применении в качестве антигенов рекомбинантных белков ORF2 и ORF3 ВГЕ-3, предоставленных НИИВС им. И.И. Мечникова [21]. Все положительные результаты, полученные при помощи тест-систем, являющихся нашей разработкой, были верифицированы повторным исследованием образцов в тест-системах сравнения производства НПО «Диагностические системы».
Набор для выделения нуклеиновых кислот («Jena Bioscience») использовали в соответствии с протоколом производителя для выделения тотальной РНК. Для выявления РНК ВГЕ применяли адаптированный нами метод с вырожденными праймерами, ориентированными на участок ОРС2 генома ВГЕ с 5905 по 6635 нт. Условия проведения ОТ-ПЦР соответствовали описанным ранее [22]. Подтверждение положительных результатов проводили коммерческим набором «HEV RT-PCR Kit 2.0» («RealStar®», «Altona»).
Набор «QIAquick Gel Extraction Kit» («QIAGEN») использовали для экстрагирования из агарозы продуктов амплификации, содержащихся в геле. Нуклеотидную последовательность фрагмента генома ВГЕ определяли в ходе прямого секвенирования ампликонов на автоматическом секвенаторе «3500 GeneticAnalyzer» («ABI») с использованием набора «BigDye Terminator v 3.1 Cycle Sequencing Kit» («Thermo Scientific»).
Анализ нуклеотидных последовательностей ВГЕ, их генотипирование и расчёт эволюционных расстояний выполняли с помощью программного обеспечения «MEGA X» [23]. В анализ были включены 59 нуклеотидных последовательностей, являющихся фрагментами ОРС2 ВГЕ величиной 273 нуклеотида (нуклеотидные позиции 6193–6466 относительно штамма Burma, номер в GenBank M73218), 11 последовательностей, выделенных из биологического материала человека и животных в Республике Беларусь, 40 референсных последовательностей для 1–8 генотипов и субгенотипов ВГЕ, предложенных D.B. Smith и соавт. [19], а также 8 наиболее близких по результатам BLAST-анализа последовательностей к выделенным в Беларуси. Последовательность птичьего ВГЕ была включена как внешняя группа для отрицательного контроля. Филогенетический анализ был проведён методом максимального правдоподобия и модели Хасегава–Кишино–Яно [24].
Результаты
Клинический случай 1
Женщина, 33 года (код на дендрограмме Patient_Сх_BY_2021|g3), поступила в отделение патологии беременности 22.02.2021. Диагноз при поступлении: «Беременность 38–39 нед. Хроническая гипоксия плода, компенсация. Краевое прикрепление пуповины». В течение последнего месяца пациентка отмечала появление кожного зуда стоп, ладоней и живота, усиливающегося к вечеру. Зуд усилился с 15.02.2021. В общем анализе мочи билирубин — 10 мкмоль/л. УЗИ органов брюшной полости — гепатомегалия. Результаты биохимического исследования крови: прямой билирубин — 5,23 мкмоль/л, аланинаминотрансфераза (АЛТ) — 289,7 ЕД/л, аспартатаминотрансфераза (АСТ) — 306,8 ЕД/л, γ-глутамилтранспептидаза (ГГТП) — 53,1 ЕД/л. Маркеры вирусов гепатита А, В и С не выявлены. Тест на анти-ВГЕ IgM — положительный, на анти-ВГЕ IgG — отрицательный.
Эпидемиологический анамнез: проживает в городской квартире, сырокопчёные и кровяные колбасы в домашних условиях не готовит, свинину, выращенную на собственном подворье, приобретает у родителей. Воду употребляет из централизованного водопровода. В течение последнего года за пределы Беларуси не выезжала. Переливание компонентов крови и парентеральные манипуляции не проводились. Гепатитом не болела, контакта с желтушными больными не было. Примерно за месяц до госпитализации готовила котлеты из фарша домашней вьетнамской свинины и свиного фарша в соотношении 1 : 1. Во время приготовления котлет пробовала на вкус сырой фарш.
В сроке гестации 280 дней произошли срочные быстрые роды. Родился живой доношенный мальчик с оценкой по шкале Апгар 8/9 баллов. Выписана домой с ребёнком на 10-е сутки после родов.
Клинический случай 2
Мужчина, 28 лет (код на дендрограмме Patient_Sh_BY_2022|g1), поступил в стационар 07.04.2022. Заболел 04.04.2022, когда на фоне утомляемости проявились желтушная окраска кожи, тёмная моча и «светлый» стул. Результаты лабораторного обследования: билирубин общий — 111,8 мкмоль/л, АЛТ — 1532 ЕД/л, АСТ — 2223 ЕД/л, щелочная фосфатаза — 275 ЕД/л, ГГТП — 347 ЕД/л. При ультразвуковом обследовании органов брюшной полости выявлена гепатомегалия. Маркеры вирусов гепатита А, В и С не выявлены. Тест на анти-ВГЕ IgM — положительный, на анти-ВГЕ IgG — положительный.
Эпидемиологический анамнез: в последнее время проживает в Гродно, является уроженцем Пакистана. За последние 6 мес выезжал за пределы Беларуси в Италию и Пакистан, вернулся из Пакистана за 2 нед до начала клинических проявлений заболевания.
Диагноз: острый ГЕ, желтушная форма, средней тяжести.
Из образцов биологического материала, полученных от пациентов, была выделена РНК ВГЕ (рис. 2).
Рис. 2. Фрагменты электрофореграмм результатов гнездовой ОТ-ПЦР. 1–8 — номера образцов; М — дорожка маркера молекулярного веса. а — Patient_Сх_BY_2021|g3: 1 — грудное молоко; 2 — сыворотка крови; 3 и 4 — кал; 5 и 6 — моча от 24.02.2021; 7 и 8 — кал от 20.03.2021; б — Patient_Sh_BY_2022|g1: 1 и 3 — сыворотка крови; 2 и 4 — кал. / Fig. 2. Fragments of electrophoregrams of the results of nested RT-PCR. 1–8 sample numbers; M — lane of the molecular weight marker. a — Patient_Сх_BY_2021|g3: 1 — breast milk; 2 — blood serum; 3 and 4 — feces; 5 and 6 — urine from 02/24/2021; 7 and 8 — feces from 03/20/2021; b — Patient_Sh_BY_2022|g1: 1 and 3 — blood serum; 2 and 4 — feces.
На основе филогенетического анализа последовательностей, кодирующих фрагмент белка капсида вируса, построено филогенетическое дерево, которое позволило оценить степень генетического родства последовательностей ВГЕ, выделенных из биологического материала, полученного из организмов изучаемых пациентов, с последовательностями, выделенными из организма человека и животных в Беларуси в более ранний период, а также с референсными последовательностями ВГЕ, установленными для генотипов и субгенотипов, и гомологичными последовательностями из базы данных GenBank (рис. 3).
Рис. 3. Филогенетическое дерево для частичной последовательности ОРС2. ▲ — последовательности, выделенные в Беларуси. Расшифровка кода последовательности: AAAAAAAA_BB_C.c_DDDD|EEF: А — код доступа в базе GenCode NCBI; B — код страны происхождения последовательности; C — сокращённое название вида хозяина; D — год выделения последовательности; E — генотип вируса; F — субгенотип. / Fig. 3. Phylogenetic tree for the partial ORF2 sequence. ▲— sequences identified in Belarus. Sequence ID decoding: AAAAAAAA_BB_C.c_DDDD|EEF: А — accession number in the GenBank NCBI database; B — code of the country of the sequence origin; C — abbreviated name of the host species; D — year of the sequence isolation; E — virus genotype; F — subgenotype.
Последовательность, выделенная из организма пациента Patient_Сх_BY_2021|g3, относится к генотипу ВГЕ-3 и кластеризуется в пределах клады 3efg. Эта последовательность в 100% репликаций образует общую филогенетическую ветвь с двумя другими, выделенными из организма человека в Беларуси: MT518198_H.s_BY_2021|g3 и Patient_Pf_BY_2019|3. Величина р-эволюционной дистанции между последовательностью, выделенной из организма пациента Patient_Сх_BY_2021|g3, и последовательностью Patient_Pf_BY_2019|3, выделенной из организма пациента, являющегося реципиентом почечного трансплантата в Беларуси в 2019 г. (код в базе GenBank: MT325974) составляет 0,015 ± 0,008, что свидетельствует о высокой степени их генетического сходства. Данный факт позволяет утверждать о наличии общего источника заражения ВГЕ обоих пациентов и эпидемиологической связи между этими случаями заболевания ГЕ. Наличие в эпидемиологическом анамнезе пациента эпизода алиментарного контакта с сырым свиным фаршем и высокой степени генетической однородности последовательностей ВГЕ, выделенных из его организма с последовательностями, выделенными из организма домашней свиньи в России и в Эстонии (рис. 2; таблица), что подтверждается незначительными эволюционными дистанциями между ними, позволяет констатировать зоонозный характер источника заражения ГЕ. Ввиду отсутствия выезда пациента за пределы Беларуси в рамках инкубационного периода заболевания, данный случай следует считать автохтонным.
Значения попарных эволюционных дистанций между нуклеотидными последовательностями ВГЕ, выделенными из организма пациентов в Республике Беларусь, а также последовательностями ВГЕ для сравнения, являющимися наиболее близкими, выявленными при помощи BLAST-анализа / Values of pairwise evolutionary distances between HEV nucleotide sequences isolated from the body of patients in the Republic of Belarus, as well as HEV sequences for comparison, which are the closest identified by BLAST analysis
№ No. | Коды последовательностей на дендрограмме Sequence IDs on the dendrogram | Эволюционное расстояние p-distance | Коды последовательностей, выявленных при помощи BLAST-анализа IDs of sequences identified by BLAST analysis |
1 | Patient_Сх_BY_2021|g3 | 0,015 ± 0,008 | MT325974_H.s_BY_2019|3 |
2 | Patient_Сх_BY_2021|g3 | 0,015 ± 0,008 | MT518198_H.s_BY_2021|g3 |
3 | Patient_Сх_BY_2021|g3 | 0,120 ± 0,023 | HQ380086_S.s_RU_2010|g3 |
4 | Patient_Сх_BY_2021|g3 | 0,128 ± 0,023 | KP871817_S.s_EST_2013|g3 |
5 | Patient_Pf_BY_2019|3 | 0,000 ± 0,000 | MT518198_H.s_BY_2021|g3 |
6 | Patient_Sh_BY_2022|g1 | 0,073 ± 0,017 | HM446620_H.s._PK_2016|g1 |
7 | Patient_Sh_BY_2022|g1 | 0,094 ± 0,019 | AB986069_H.s_NP_2014|g1a |
8 | Patient_Sh_BY_2022|g1 | 0,098 ± 0,020 | LC225387_MO_H.s_2015|g1g |
Последовательность РНК, выделенной из организма пациента Patient_Sh_BY_2022|g1, относится к генотипу ВГЕ-1. Возможность завоза на территорию Беларуси штаммов ВГЕ, имеющих высокий эпидемический потенциал, обсуждалась нами ранее [25]. Генотипирование данной последовательности не имеет однозначной трактовки. На online-платформе «Automated genotyping and interpretation HEV»2 были установлены весовые коэффициенты клады для данной последовательности. С вероятностью 32,8% эту последовательность можно отнести к субгенотипу ВГЕ-1a и с вероятностью 25,23% — к субгенотипу ВГЕ-1f. Эта последовательность в 54% репликаций образует общую филогенетическую ветвь с рядом последовательностей, выделенных из организма человека в странах Юго-Восточной Азии (таблица). Наименьшее значение р-эволюционной дистанции, составляющее 0,073 ± 0,017, разделяет последовательности Patient_Sh_BY_2022|g1, выделенную из организма пациента в Беларуси, и HM446620_H.s._PK_2016|g1, выделенную из организма пациента в Пакистане. Незначительная величина эволюционной дистанции между этими двумя последовательностями, свидетельствующая о высокой степени их гомологичности, а также наличие в эпидемиологическом анамнезе пациента эпизода выезда за пределы Беларуси в Пакистан позволяют утверждать, что данный случай ГЕ является завозным.
Обсуждение
Как отмечалось нами ранее, частота встречаемости анамнестических антител к ВГЕ в общей популяции условно здорового населения Беларуси составляет 7,3% [26], что эквивалентно примерно 10 тыс. ежегодных случаев ВГЕ-инфекции и явно не согласуется с официальными показателями заболеваемости ГЕ в Беларуси, составляющими 0,02–0,04 на 100 тыс. населения. Это означает, что 99,9% первичных инфекций ВГЕ остаются недиагностированными. Подобная особенность ещё в большей степени выражена в некоторых странах Западной Европы (Франция, Германия, Нидерланды и Польша), являющихся анклавами по ГЕ, где показатель частоты выявления анти-ВГЕ IgG в общей популяции почти двукратно превышает его значение в Беларуси [27]. Факты, объясняющие существование «парадокса Балаяна» [28], несомненно, лежат в плоскости молекулярно-генетической организации ВГЕ. Одно из возможных объяснений низкой частоты симптоматической инфекции ВГЕ заключается в том, что разные варианты ВГЕ-3 обладают разным патогенным потенциалом. Хотя в настоящее время не получено достоверных данных, подтверждающих это, всё же ряд исследований показывает, что штаммы ВГЕ-3, относящиеся к кладе 3efg, являются более патогенными [29]. Недавнее исследование образцов биологического материала, полученных от белорусских доноров, позволило выявить РНК ВГЕ в АЛТ-положительной сыворотке с высоким титром анти-ВГЕ IgM, что доказывает существование интенсивного эпидемического процесса ГЕ в стране. Таким образом, проведение молекулярно-эпидемиологических исследований, включающих генотипирование вариантов ВГЕ, циркулирующих на территории Беларуси, имеет высокую актуальность.
Проведённые нами исследования показали, что на территории Беларуси циркулируют возбудители ГЕ, относящиеся к разным генотипам и субгенотипам, имеющие различный механизм распространения и отличающиеся по эпидемическому потенциалу. Нами выявлен случай острого ГЕ у беременной, сопровождающийся выраженной клинической манифестацией. Возбудитель был генотипирован как ВГЕ-3 клада 3efg. К счастью, данный случай гепатита беременной не привёл к осложнениям для плода и благополучно завершился для матери. Однако нами был ранее описан клинический случай с аналогичным эпидемическим анамнезом, завершившийся антенатальной гибелью плода [30], что придаёт данному исследованию больший вес.
Учитывая, что ГЕ у беременных может прогрессировать до фульминантного гепатита, скрининг ГЕ у этой группы пациентов должен стать нормой оказания акушерско-гинекологической помощи. Необходимо информировать беременных о потенциальном воздействии ВГЕ на их организм и плод, о существовании вероятных неблагоприятных исходов для беременной и плода. Им следует рекомендовать избегать употребления пищевых продуктов и воды, являющихся возможными источниками ВГЕ. В случае осложнённого течения беременности и родов необходимо использовать только отрицательную по РНК ВГЕ донорскую кровь и её компоненты. Наконец, существует также существенная потребность в разработке новых методов лечения ГЕ во время беременности.
Исходя из имеющихся данных, нельзя утверждать о преобладании циркуляции того или иного субгенотипа ВГЕ на территории Беларуси. Из 11 последовательностей РНК, выделенных в Беларуси, 7 было получено из организма человека, из которых 6 отнесены к ВГЕ-3, 1 — к ВГЕ-1. Три из 6 «белорусских» последовательностей ВГЕ-3 относятся к кладе 3efg, 3 — к кладе 3abchij. Из 4 последовательностей ВГЕ-3, полученных от животных, 2 от кроликов генотипированы как ВГЕ-3 rabbit, 2 от свиней отнесены к кладе 3abchij [31].
Результаты проведённых исследований свидетельствуют о существовании значительных рисков завоза ВГЕ на территорию Беларуси. Доказан эпизод завоза ВГЕ с гиперэндемичной по ГЕ территории. Последовательность, выделенная из организма пациента с клиническими проявлениями острого ГЕ, с вероятностью 100% отнесена к ВГЕ-1. Данная последовательность на 94,3% гомологична последовательности, ранее выделенной от пациента в Пакистане. При формировании соответствующих условий генотип ВГЕ, обладающий значительным эпидемическим потенциалом, может стать причиной развития вспышек ГЕ. Это может быть подтверждено описанным нами ранее случаем заболевания острым ГЕ в безжелтушной форме иностранной студентки [30], которая проживала в общежитии для иностранных студентов и не выезжала за пределы Беларуси в течение последних 2 лет. Существование рисков завоза ВГЕ в Беларусь с гиперэндемичных территорий обусловливает необходимость организации мониторинга иностранных граждан на анти-ВГЕ IgM и РНК ВГЕ при их прибытии в Беларусь и прохождении ими медицинских осмотров.
1 ВОЗ. Гепатит Е. Основные факты. Available at: https://www.who.int/ru/news-room/fact-sheets/detail/hepatitis-e/
2 HEV-GLUE. MRC-University of Glasgow Centre for Virus Research. Available at: http://hev.glue.cvr.ac.uk/#/analysisTool
About the authors
Vladimir V. Davydov
Belarusian State Medical University
Author for correspondence.
Email: davidovvv@bsmu.by
ORCID iD: 0000-0002-5672-9509
Cand. Sci. (Biol.), Associate Professor, Head, Department of biology
Belarus, MinskSergey V. Zhavoronok
Belarusian State Medical University
Email: davidovvv@bsmu.by
ORCID iD: 0000-0001-9727-1103
D. Sci. (Med.), Professor, Department of infectious diseases
Russian Federation, MinskTatyana V. Znovets
Third City Clinical Hospital named after E.V. Klumov
Email: davidovvv@bsmu.by
ORCID iD: 0000-0002-5603-490X
Cand. Sci. (Med.), Deputy chief physician for obstetrics
Belarus, MinskVladimir M. Tsyrkunov
Grodno State Medical University
Email: davidovvv@bsmu.by
ORCID iD: 0000-0002-9366-6789
D. Sci. (Med.), Professor, Department of infectious diseases
Belarus, GrodnoAndrei S. Babenkа
Belarusian State Medical University
Email: davidovvv@bsmu.by
ORCID iD: 0000-0002-5513-970X
Cand. Sci. (Chem.), Associate Professor, Department of bioorganic chemistry
Belarus, MinskSvetlana I. Marchuk
Belarusian State Medical University
Email: davidovvv@bsmu.by
ORCID iD: 0000-0002-2291-4538
researcher, Laboratory of biochemical research methods of the scientific part
Belarus, MinskElena L. Gasich
Republican Scientific and Practical Center of Epidemiology and Microbiology
Email: davidovvv@bsmu.by
ORCID iD: 0000-0002-3662-3045
D. Sci. (Biol.), Associate Professor, Head, Laboratory for diagnosis of HIV and concomitant infections
Belarus, MinskTatiana P. Novik
Belarusian State Medical University
Email: davidovvv@bsmu.by
ORCID iD: 0000-0002-3220-9592
Cand. Sci. (Biol.), Associate Professor, Department of biology
Belarus, MinskGalina I. Alatortseva
I. Mechnikov Research Institute of Vaccines and Sera
Email: davidovvv@bsmu.by
ORCID iD: 0000-0001-9887-4061
Cand. Sci. (Biol.), Head, Laboratory for cloning viral genomes
Russian Federation, MoscowMikhail I. Mikhailov
I. Mechnikov Research Institute of Vaccines and Sera; Russian Medical Academy of Continuous Professional Education
Email: davidovvv@bsmu.by
ORCID iD: 0000-0002-6636-6801
D. Sci. (Med.), Professor, Corresponding member of the Russian Academy of Sciences, Head, Laboratory for viral hepatitis, Scientific Director, Institute of Molecular and Personalized Medicine
Russian Federation, Moscow; MoscowVitaly V. Zverev
I. Mechnikov Research Institute of Vaccines and Sera; I.M. Sechenov First Moscow State Medical University (Sechenov University)
Email: davidovvv@bsmu.by
ORCID iD: 0000-0002-0017-1892
D. Sci. (Biol.), Professor, Full Member of the Russian Academy of Sciences, scientific director, Head, Department of microbiology, virology, immunology
Russian Federation, Moscow; MoscowReferences
- Balayan M.S., Andjaparidze A.G., Savinskaya S.S., Ketiladze E.S., Braginsky D.M., Savinov A.P., et al. Evidence for a virus in non-A, non-B hepatitis transmitted via the fecal-oral route. Intervirology. 1983; 20(1): 23–31. https://doi.org/10.1159/000149370
- Purdy M.A., Harrison T.J., Jameel S., Meng X.J., Okamoto H., Van der Poel W.H.M., et al. ICTV virus taxonomy profile: Hepeviridae. J. Gen. Virol. 2017; 98(11): 2645–6. https://doi.org/10.1099/jgv.0.000940
- Lee G.H., Tan B.H., Teo E.C., Lim S.G., Dan Y.Y., Wee A., et al. Chronic infection with camelid hepatitis E virus in a liver transplant recipient who regularly consumes camel meat and milk. Gastroenterology. 2016; 150(2): 355–7.e3. https://doi.org/10.1053/j.gastro.2015.10.048
- Pallerla S.R., Harms D., Johne R., Todt D., Steinmann E., Schemmerer M., et al. Hepatitis E virus infection: circulation, molecular epidemiology, and impact on global health. Pathogens. 2020; 9(10): 856. https://doi.org/10.3390/pathogens9100856
- Nimgaonkar I., Ding Q., Schwartz R.E., Ploss A. Hepatitis E virus: advances and challenges. Nat. Rev. Gastroenterol. Hepatol. 2018; 15(2): 96–110. https://doi.org/10.1038/nrgastro.2017.150
- European Association for the Study of the Liver. EASL Clinical Practice Guidelines on hepatitis E virus infection. J. Hepatol. 2018; 68(6): 1256–71. https://doi.org/10.1016/j.jhep.2018.03.005
- Li P., Liu J., Li Y., Su J., Ma Z., Bramer W.M., et al. The global epidemiology of hepatitis E virus infection: A systematic review and meta-analysis. Liver. Int. 2020; 40(7): 1516–28. https://doi.org/10.1111/liv.14468
- Pérez-Gracia M.T., Suay-García B., Mateos-Lindemann M.L. Hepatitis E and pregnancy: current state. Rev. Med. Virol. 2017; 27(3): e1929. https://doi.org/10.1002/rmv.1929
- Patra S., Kumar A., Trivedi S.S., Puri M., Sarin S.K. Maternal and fetal outcomes in pregnant women with acute hepatitis E virus infection. Ann. Intern. Med. 2007; 147(1): 28–33. https://doi.org/10.7326/0003-4819-147-1-200707030-00005
- Kinast V., Burkard T.L., Todt D., Steinmann E. Hepatitis E virus drug development. Viruses. 2019; 11(6): 485. https://doi.org/10.3390/v11060485
- Wu C., Wu X., Xia J. Hepatitis E virus infection during pregnancy. Virol. J. 2020; 17(1): 73. https://doi.org/10.1186/s12985-020-01343-9
- El-Mokhtar M.A., Othman E.R., Khashbah M.Y., Ismael A., Ghaliony M.A., Seddik M.I., et al. Evidence of the extrahepatic replication of hepatitis e virus in human endometrial stromal cells. Pathogens. 2020; 9(4): 295. https://doi.org/10.3390/pathogens9040295
- Nagashima S., Takahashi M., Kobayashi T., Nishizawa T., Nishiyama T., Primadharsini P.P., et al. Characterization of the Quasi-Enveloped Hepatitis E Virus Particles Released by the Cellular Exosomal Pathway. J. Virol. 2017; 91(22): e00822-17. https://doi.org/10.1128/jvi.00822-17
- Marion O., Lhomme S., Nayrac M., Dubois M., Pucelle M., Requena M., et al. Hepatitis E virus replication in human intestinal cells. Gut. 2020; 69(5): 901–10. https://doi.org/10.1136/gutjnl-2019-319004
- Debing Y., Moradpour D., Neyts J., Gouttenoire J. Update on hepatitis E virology: Implications for clinical practice. J. Hepatol. 2016; 65(1): 200–12. https://doi.org/10.1016/j.jhep.2016.02.045
- Montpellier C., Wychowski C., Sayed I.M., Meunier J.C., Saliou J.M., Ankavay M., et al. Hepatitis E virus lifecycle and identification of 3 forms of the ORF2 capsid protein. Gastroenterology. 2018; 154(1): 211–23.e8. https://doi.org/10.1053/j.gastro.2017.09.020
- Graff J., Torian U., Nguyen H., Emerson S.U. A bicistronic subgenomic mRNA encodes both the ORF2 and ORF3 proteins of hepatitis E virus. J. Virol. 2006; 80(12): 5919–26. https://doi.org/10.1128/jvi.00046-06
- Yadav K.K., Boley P.A., Fritts Z., Kenney S.P. Ectopic expression of genotype 1 hepatitis E virus ORF4 increases genotype 3 HEV viral replication in cell culture. Viruses. 2021; 13(1): 75. https://doi.org/10.3390/v13010075
- Smith D.B., Izopet J., Nicot F., Simmonds P., Jameel S., Meng X.J., et al. Update: proposed reference sequences for subtypes of hepatitis E virus (species Orthohepevirus A). J. Gen. Virol. 2020; 101(7): 692–8. https://doi.org/10.1099/jgv.0.001435
- Krasochko P.A., Zhavoronok S.V., Borisovets D.S., Krasochko P.P., Alatortseva G.I., Prokopenkova T.M., et al. Development of elisa test kits for semi-quantitative detection of anti-hev antibodies in pigs in the republic of Belarus. Izvestiya Natsional'noy akademii nauk Belarusi. Seriya agrarnykh nauk. 2020; (3): 339–51. https://doi.org/10.29235/1817-7204-2020-58-3-339-351 (in Russian)
- Alatortseva G.I., Sidorov A.V., Nesterenko L.N., Lukhverchik L.N., Amiantova I.I., Dotsenko V.V., et al. Recombinant protein containing antigenically significant fragments of hepatitis E virus proteins used in test systems for hepatitis E serodiagnosis (versions). Patent RF 2711907 C2; 2020. (in Russian)
- Arabey A.A., Marchuk S.I., Zhavoronok S.V., Davydov V.V., Kyuregyan K.K., Mikhaylov M.I. Adapted polymerase chain reaction method for detecting hepatitis E virus in humans and animals. Voennaya meditsina. 2018; (3): 86–92. (in Russian)
- Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms. Mol. Biol. Evol. 2018; 35(6): 1547–9. https://doi.org/10.1093/molbev/msy096
- Hasegawa M., Kishino H., Yano T. Dating of the human-ape splitting by a molecular clock of mitochondrial DNA. J. Mol. Evol. 1985; 22(2): 160–74. https://doi.org/10.1007/bf02101694
- Davydov V.V. Zhavoronok S.V. Anis'ko L.A. Gasich E.L. Marchuk S.I. Semizhon P.A. et al. Study of the risks of hepatitis E importation to the Republic of Belarus. BSMU in the Vanguard of Medical Science and Practice: The Scientific Works Collection. 2020; (10): 285–90. (in Russian)
- Davydov V.V., Zhavoronok S.V., Rogacheva T.A., Novik T.P., Alatortseva G.I., Nesterenko L.N. et al. Prevalence of antibodies to the hepatitis E virus in the population of the Republic of Belarus. Zhurnal mikrobiologii, epidemiologii i immunobiologii. 2022; 99(2): 160–71. https://doi.org/10.36233/0372-9311-236 (in Russian)
- Li P., Liu J., Li Y., Su J., Ma Z., Bramer W.M., et al. The global epidemiology of hepatitis E virus infection: A systematic review and meta-analysis. Liver Int. 2020; 40(7): 1516–28. https://doi.org/10.1111/liv.14468
- Mikhaylov M.I., Malinnikova E.Yu., Kyuregyan K.K., Potemkin I.A., Alsalikh N.D., Isaeva O.V. et al. Balayan paradox. Zhurnal mikrobiologii, epidemiologii i immunobiologii. 2019; 96(1): 80–5. https://doi.org/10.36233/0372-9311-2019-1-80-85 (in Russian)
- Smith D.B., Ijaz S., Tedder R.S., Hogema B., Zaaijer H.L., Izopet J., et al. Variability and pathogenicity of hepatitis E virus genotype 3 variants. J. Gen. Virol. 2015; 96(11): 3255–64. https://doi.org/10.1099/jgv.0.000264
- Zhavoronok S.V., Karpov I.A., Mikhaylov M.I., Arabey A.A., Kashkur Yu.V., Kyuregyan K.K., et al. The intensity of the epidemic and epizootic processes of infection caused by the hepatitis E virus in the territory of the Republic of Belarus. Infektsionnye Bolezni: Novosti, Mneniya, Obuchenie. 2019; 8(1): 11–22. https://doi.org/10.24411/2305-3496-2019-11001 (in Russan)
- Davydov V.V. Genetic diversity of hepatitis e virus in the republic of Belarus. Klinicheskaya infektologiya i parazitologiya. 2020; 9(3): 297–305. https://doi.org/10.34883/PI.2020.9.3.029 (in Russian)
Supplementary files