Competitive ability of Escherichia coli strains in the intestinal microbiota of patients with Crohn's disease and healthy volunteers: physiological, biochemical and genetic characteristics
- Authors: Siniagina M.N.1, Laikov A.V.1, Markelova M.I.1, Boulygina E.A.1, Khusnutdinova D.R.1, Abdulkhakov S.R.1, Grigoryeva T.V.1
-
Affiliations:
- Kazan Federal University
- Issue: Vol 99, No 6 (2022)
- Pages: 669-681
- Section: ORIGINAL RESEARCHES
- URL: https://microbiol.crie.ru/jour/article/view/1081
- DOI: https://doi.org/10.36233/0372-9311-192
- ID: 1081
Cite item
Full Text
Abstract
Introduction. Crohn's disease (CD) is a chronic inflammation of various parts of the gastrointestinal tract with an increased proportion of Escherichia coli. However, the role of E. coli in disease remains unclear.
This study aims to evaluate the competitive abilities of E. coli strains from CD patients and healthy volunteers, and to identify the biochemical and genetic determinants underlying these features.
Materials and methods. The antagonistic activity was assessed by co-cultivation of 11 clinical E. coli strains inhibiting the growth of the K-12, with Enterobacter cloacae, Klebsiella pneumonia and Salmonella enterica. To elucidate the mechanism of antagonistic activity, the evaluation of biochemical properties and a comparative genomic analysis were used.
Results and discussion. Genes of bacteriocin production systems were identified in genomes of 11 strains from CD patients and healthy volunteers active against the E. coli K-12 strain. Three strains from healthy individuals demonstrated activity against several Enterobacteriaceae bacteria. The strains’ biochemical properties were typical of representatives of E. coli. Strains 1_34_12, active against E. cloacae, and 1_45_11, inhibiting all tested enterobacteria, are phylogenetically related to the laboratory strain K-12. Strain 1_39_1, active against K. pneumonia and S. enterica, is phylogenetically close to the Nissle1917, contains the genes for colibactin biosynthesis and a variant of the fimH gene that increases the adhesive ability of bacteria.
Conclusion. The identified E. coli strains are able to displace Enterobacteriaceae bacteria and can be used to study the bacteria-bacteria and host-bacteria interactions, to understand their role in gut homeostasis and intestinal inflammation.
Full Text
Введение
Желудочно-кишечный тракт человека содержит таксономически и функционально сложную микробную экосистему [1]. Микроорганизмы, заселяющие кишечник, находятся в разнообразных взаимоотношениях как с макроорганизмом, так и между собой [2]. В условиях ограниченного пространства кишечника преимуществом обладают микроорганизмы, способные использовать одно или несколько питательных веществ более эффективно, чем его конкуренты [3]. У комменсалов развились различные механизмы, позволяющие им выживать и успешно колонизировать кишечник [4]. При конкуренции за использование ресурса без прямого взаимодействия между конкурентами организмы секретируют специализированные метаболиты, такие как бактериоцины [5], нацеленные, как правило, на близкородственные организмы. При прямом контактном взаимодействии важную роль играют системы, которым не нужны специфические мишени, — система контактзависимого ингибирования (contact-dependent growth inhibition — CDI) [6, 7] и система секреции 6-го типа (type 6 secretion system — T6SS) [8, 9]. Показано, что конкурентное вытеснение комменсалами микроорганизмов обеспечивает колонизационную резистентность кишечника и защиту организма хозяина от патогенных микроорганизмов [10–12]. Однако нарушение гомеостаза под действием различных факторов (приём антибиотиков, бактериальная или вирусная инфекция, нарушения иммунной системы) могут приводить к дисбалансу, сопровождающемуся снижением разнообразия и сменой занимаемых экологических ниш бактериями, вызывая различные патологические процессы в кишечнике [13, 14].
Болезнь Крона (БК) — хроническое воспаление различных отделов желудочно-кишечного тракта, прогрессирующее с течением времени и характеризующееся чередой обострений и ремиссий [15, 16]. БК сопровождается нарушениями микробиоты, в частности увеличением доли Escherichia coli, однако определённые причины данного явления не выявлены. В то же время кишечная палочка является важным компонентом микробиоты и заселяет кишечник человека в течение 1-го года жизни [17, 18]. Предполагают, что патогенные штаммы используют альтернативные источники питания и занимают ниши, не занятые комменсальными бактериями [19]. Кроме того, рассматривают возможность проявления комменсальными штаммами E. coli патогенных свойств [20], поскольку бактерии содержат генетические детерминанты (факторы вирулентности), которые не только помогают им адаптироваться к новым условиям окружающей среды, но и воздействовать на организм человека, вызывая патологические процессы [21].
Цель данной работы — характеристика конкурентных свойств штаммов E. coli, выделенных из кишечника пациентов с БК и здоровых добровольцев, а также выявление биохимических и генетических детерминант, лежащих в их основе.
Материалы и методы
Нами проанализированы 11 штаммов E. coli, продемонстрировавших ранее ингибирующую активность в отношении E. coli K-12 substr. MG1655 [22] и входящих в коллекцию из 63 штаммов, выделенных из кала 32 людей (14 пациентов с БК и 18 здоровых добровольцев) и охарактеризованных нами ранее [22, 23]. Образцы кала были собраны в Университетской клинике Казанского (Приволжского) федерального университета (Медико-санитарная часть). Все пациенты были проинформированы об участии в исследовании и дали своё согласие. Исследование было рассмотрено и одобрено локальным этическим комитетом Казанского (Приволжского) федерального университета (протокол № 6 от 13.10.2017).
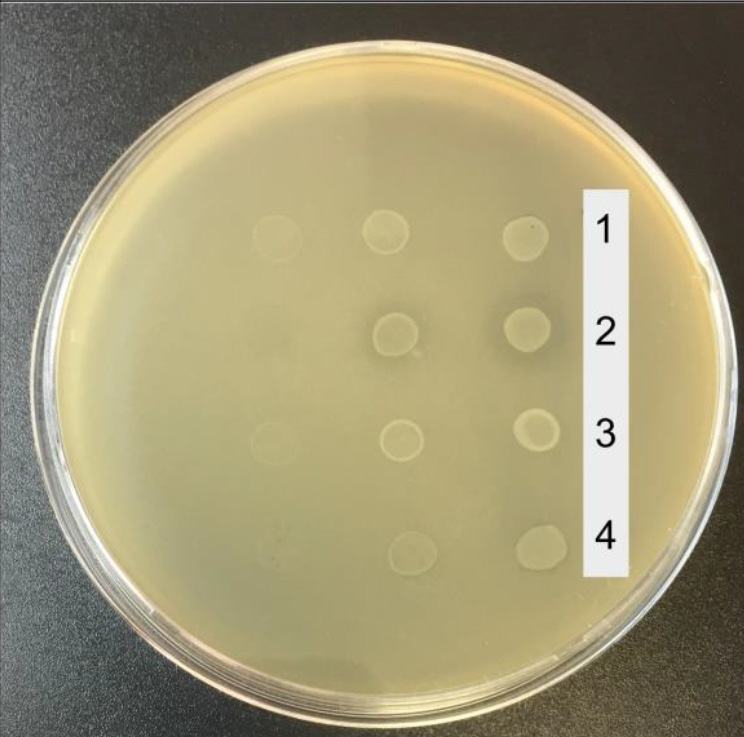
Для оценки антагонистической активности использовали представителей семейства Enterobacteriaceae — изоляты Enterobacter cloacae 3_222_2 и Klebsiella pneumonia 3_219_1, выделенные из кала пациентов с БК, а также культуру Salmonella enterica, предоставленную сотрудниками Республиканской клинической инфекционной больницы им. проф. А.Ф. Агафонова (Казань, Россия). Антагонистическую активность оценивали на плотной питательной среде согласно [24] с некоторыми модификациями. Для этого тестовую культуру бактерий смешивали с расплавленным 0,8% LB-агаром, наносили на 2% LB-агар и инкубировали в течение 30 мин при комнатной температуре до полного затвердения. По 3 мкл исходной культуры каждого штамма E. coli (OD600 = 1,0) и её разведений в 10 и 100 раз наносили на подготовленные чашки с тестовой культурой и инкубировали в течение ночи при 37°C. Эксперимент проводили в 2 повторах. Активность считали антагонистической при размере зон задержки роста тестовой культуры более 1 мм (рис. 1).
Рис. 1. Оценка антагонистической активности исследуемых штаммов E. coli на примере совместного культивирования с E. coli K-12 substr. MG1655. / Fig. 1. E. coli antagonistic activity evaluation using co-cultivation with E. coli K-12 substr. MG1655 as an example.
Биохимические реакции проводили с использованием коммерческого набора «ENTEROtest 24 N» («Lachema»), представляющего собой планшет с субстратами для определения утилизации аргинина, орнитина, лизина, малоната, салицина, сорбитола, мелибиозы, целлобиозы, лактозы, трегалозы, маннитола, дульцита, адонитола, арабитола, сахарозы, инозитола, раффинозы и эскулина, роста на цитрате Симмонса, наличия β-галактозидазы, β-глюкуронидазы, β-ксилоксидазы, уреазной активности и продукции сероводорода.
Штаммы E. cloacae и K. pneumonia выделены нами из образцов фекалий пациентов с БК и являются потенциальными конкурентами E. coli в микробиоте кишечника. S. enterica служит причиной кишечных расстройств и опасна для здоровья человека. Интерпретация результатов теста по активности штаммов пациентов и здоровых людей затруднена ввиду большого разнообразия обнаруженных генов бактериоцинов у E. coli и непредсказуемого влияния их друг на друга. Поэтому для оценки был использован модельный лабораторный штамм E. coli К-12 substr. MG1655.
Поиск генов, кодирующих ферменты продукции бактериоцинов и колибактина, а также T6SS и CDI в геномах штаммов E. coli проводили путём выравнивания на нуклеотидные последовательности референсных генов с использованием BLASTn (идентичность не менее 80%, длина гена более 80%, E < 10–16). Поиск нуклеотидных замен в гене fimH (адгезин) исследуемых штаммов проводили путём множественного выравнивания с использованием Clustal Omega1. Последовательность гена fimH штамма E. coli K-12 substr. MG1655 была взята в качестве референсной. Последовательности полных геномов 63 штаммов, выделенных из образцов кала 32 людей (14 пациентов с БК и 18 здоровых добровольцев), загружены в NCBI (проект PRJNA560176).
Для филогенетического анализа последовательности геномов были загружены на платформу Type (Strain) Genome Server2 [25]. При построении дерева использовали референсные геномы E. coli K-12 substr. MG1655 (GCF_904425475.1), E. coli Nissle 1917 (GCF_003546975.1), Salmonella enterica subsp. enterica str. 92-0392 (GCF_002761055.1), Enterobacter cloacae GGT036 (GCF_000770155.1), Klebsiella pneumonia subsp. pneumoniae str. HS11286 (GCF_000240185.1), доступные в базе данных NCBI3. Попарные сравнения всех геномных последовательностей производили с использованием метода Genome BLAST Distance Phylogeny (GBDP) и оценки точных межгеномных расстояний, подсчитанных с помощью алгоритма «trimming» и формулы расстояния d5 [26]. Полученные межгеномные расстояния были использованы для построения минимального дерева эволюции с поддержкой ветвей (с использованием 100 pseudo-bootstrap повторов) с по- мощью программы FASTME 2.1.6.1 [27]. Филограмма была визуализирована с помощью PhyD3 [28].
Статистический анализ частоты встречаемости биохимических свойств и генов бактериоцинов проводили с помощью точного критерия Фишера; р < 0,05 принимали за уровень статистической достоверности.
Результаты и обсуждение
По результатам первичного скрининга 63 штам- мов [22] антагонистическую активность в отношении лабораторного штамма E. coli K-12 substr. MG1655 проявили 11 штаммов: 4 — от пациентов с БК и 7 — от здоровых добровольцев (p > 0,05, точный критерий Фишера). В настоящей работе в качестве модели межвидовой конкуренции было проведено совместное культивирование данных штаммов с бактериями E. cloacae, K. pneumonia и S. enterica. Антагонистическую активность в отношении всех протестированных культур энтеробактерий проявили только штаммы, выделенные из фекалий здоровых добровольцев (табл. 1). В отношении E. cloacae были активны штаммы 1_34_12 и 1_45_11, рост K. pneumonia и S. enterica подавляли штаммы 1_39_1 и 1_45_11. Таким образом, были отобраны 3 штамма E. coli от здоровых добровольцев, проявившие способность вытеснять конкурентные микроорганизмы in vitro.
Таблица 1. Антагонистическая активность 11 штаммов E. coli в отношении представителей семейства Enterobacteriaceae / Table 1. Antagonistic activity of 11 E. coli strains against other members of Enterobacteriaceae family
Конкурентный штамм Competitive strain | Штамм E. coli | E. coli strain | ||||||||||
1_18_1 | 1_25_1 | 1_25_14 | 1_34_12 | 1_39_1 | 1_45_11 | 36k_8 | 3_77_1 | 3_188_1 | 3_200_11 | 3_225_1 | |
Enterobacter cloacae | – | – | – | + | – | + | – | – | – | – | – |
Klebsiella pneumonia | – | – | – | – | + | + | – | – | – | – | – |
Salmonella enterica | – | – | – | – | + | + | – | – | – | – | – |
Микробиота кишечника в значительной степени детерминирована спектром питательных веществ, зависящих от диеты хозяина [29]. В ряде работ продемонстрированы различия в структуре микробиоты кишечника сельских и городских жителей [30, 31]. Питательные вещества в кишечнике варьируют во времени и пространстве [32], поэтому метаболическая гибкость и способность использовать разные субстраты являются важными стратегиями для выживания бактерий в условиях постоянных колебаний состава питательных веществ [33]. По результатам анализа биохимических свойств не выявлено статистически значимых различий в способности утилизировать субстраты среди штаммов от пациентов с БК и здоровых добровольцев. В обеих группах присутствовали штаммы, утилизирующие адонитол и арабитол, но не способные утилизировать лактозу. Данные биохимические свойства являются редкими для E. coli и встречаются у 5% представителей вида согласно определителю бактерий Берджи [34]. Стоит отметить, что три штамма, способные подавлять рост E. cloacae, K. pneumonia и/или S. enterica, обладают типичными биохимическими свойствами, характерными для вида E. coli. Штаммы 1_45_11 и 1_34_12 способны ферментировать сорбит, лактозу, маннит, мелибиозу, трегалозу, а также обладают β-галактозидазной и β-глюкуронидазной активностью и содержат аргининдигидролазу и лизиндекарбоксилазу (рис. 2). Штамм 1_39_1, кроме того, способен утилизировать орнитин.
Рис. 2. Биохимические свойства клинических штаммов E. coli, проявивших антагонистическую активность в отношении представителей семейства Enterobacteriaceae.*Штаммы от пациентов с БК. E. coli K-12 substr. MG1655 — контрольный штамм. / Fig. 2. Biochemical properties of clinical E. coli strains showing antagonistic activity against Enterobacteriaceae. *Strains from patients with Crohn’s disease. E. coli K-12 substr. MG1655 is a control strain.
Согласно результатам подсчёта КОЕ, 8 из 11 штаммов доминируют в микробном сообществе кишечника участников исследования (табл. 2). Минорную долю в кишечном сообществе составляют штаммы 1_25_14 и 3_200_11, что, вероятно, связано с неспособностью утилизировать данными штаммами лактозу, являющуюся важнейшим источником углерода для E. coli [34], а также штамм 3_225_1, который, как и штамм 3_200_11, не утилизирует мелибиозу. Можно предположить, что мелибиоза даёт конкурентное преимущество бактериям при колонизации кишечника.
Таблица 2. Доля штаммов, проявивших антагонистическую активность в отношении E. coli К-12, в кишечном сообществе участников исследования / Table 2. The proportion of strains that showed antagonistic activity against E. coli K-12 in the intestinal community of study participants
Штамм E. coli | E. coli strain | Источник выделения штамма | Source of strain isolation | Доля в сообществе, % Proportion in the community, % |
1_18_1 | Здоровый доброволец | Healthy volunteer | 96 |
1_25_1 | Здоровый доброволец | Healthy volunteer | 97,3 |
1_25_14 | Здоровый доброволец | Healthy volunteer | 0,8 |
1_34_12 | Здоровый доброволец | Healthy volunteer | 79,5 |
1_39_1 | Здоровый доброволец | Healthy volunteer | 95 |
1_45_11 | Здоровый доброволец | Healthy volunteer | 84,5 |
36k_8 | Здоровый доброволец | Healthy volunteer | 84,7 |
3_77_1 | Пациент с БК | CD patient | 100 |
3_188_1 | Пациент с БК | CD patient | 100 |
3_200_11 | Пациент с БК | CD patient | 24 |
3_225_1 | Пациент с БК | CD patient | 16 |
В ходе анализа геномов у всех конкурентных штаммов обнаружены гены систем продукции бактериоцинов, дающих внутри- и межвидовое конкурентное преимущество при колонизации кишечника [5, 35] (табл. 3). Геномы штаммов пациентов с БК содержат полные кластеры генов продукции микроцинов J25 (штамм 3_77_1) и H47 (штамм 3_188_1), а также гены, необходимые для продукции колицинов А (штамм 3_200_11) и К (штамм 3_225_1). Штаммы здоровых добровольцев содержат полные кластеры генов продукции микроцинов С7 (штамм 1_39_1), В17 (штамм 36к_8), J25 (штамм 36к_8), H47 (штаммы 1_34_12, 1_39_1, 36к_8) и М (штамм 1_34_12), а также колицинов А (штаммы 1_25_1, 1_25_14, 1_34_12), В (штамм 3_77_1), Е1 (штамм 1_45_11), Ia (штамм 1_45_11), Ib (штамм 1_45_11), К (штамм 1_18_1) и М (штамм 1_45_11). Микроцин J25 неэффективен в отношении E. cloacae, K. pneumonia и S. enterica [36]. Кроме того, синтез микроцинов J25, I47 и H47 ингибируется в условиях культивирования на богатой питательной среде [37, 38], а синтез микроцина В17 индуцируется в условиях азотного голодания [39], что может объяснять неспособность штаммов 36к_8, 3_77_1 и 3_188_1, содержащих данные системы, подавлять рост E. cloacae, K. pneumonia и S. enterica. Таким образом, можно предположить, что активность штамма 1_39_1 в отношении K. pneumonia и S. enterica связана с кластером генов продукции микроцина С7, что соответствует данным литературы [40]. Способность штамма 1_34_12 ингибировать рост E. cloacae может быть опосредована системой продукции микроцина М. Интересно, что штамм 1_45_11 не содержит систем продукции микроцинов, однако ингибирует рост S. enterica, что, вероятно, связано с наличием у него генов колицинов Ia, Ib и M, которые, согласно данным литературы, эффективны в отношении сальмонелл [41]. Кроме того, в недавнем исследовании показано, что E. coli, содержащие гены продукции данных колицинов, подавляют рост S. enterica и K. pneumoniae [42].
Таблица 3. Гены систем продукции бактериоцинов, обнаруженные у 11 штаммов E. coli, проявивших антагонистическую активность в отношении представителей семейства Enterobacteriaceae / Table 3. Genes for bacteriocin production systems found in E. coli strains that showed antagonistic activity against Enterobacteriaceae
Фактор Bacteriocin | Ген Gene | Функция Function | Штаммы E. coli | E. coli strain | ||||||||||
от здоровых добровольцев from healthy individuals | от пациентов с БК from patients with CD | ||||||||||||
1_34_12 | 1_39_1 | 1_45_11 | 1_18_1 | 1_25_1 | 1_25_14 | 36k_8 | 3_77_1 | 3_188_1 | 3_200_11 | 3_225_1 | |||
Колицин | Colicin | |||||||||||||
A | caa | Колицин A (неполный) | Colicin A (incomplete) | + | – | – | – | + | + | – | – | + | + | – |
cai | Иммунитет к колицину | Colicin А immunity | + | – | + | – | + | + | – | + | + | + | – | |
B | cba | Колицин B | Colicin B | – | – | + | – | – | – | – | + | – | – | – |
cbi | Иммунитет к колицину | Colicin B immunity | – | – | + | – | – | – | – | + | – | – | – | |
E1 | cea | Колицин E1 | Colicin E1 | – | – | + | – | – | – | – | – | – | – | – |
imm | Иммунитет к колицину | Colicin E1 immunity | – | – | + | – | – | – | – | – | – | – | – | |
lys | Белок лизиса | Lysis | – | – | + | – | – | – | – | – | – | – | – | |
Ia | cia | Колицин Ia | Colicin Ia | – | – | + | – | – | – | – | – | – | – | – |
iia, imm | Иммунитет к колицину | Colicin Ia immunity | – | – | + | – | – | – | – | – | – | – | – | |
Ib | cib | Колицин Ib | Colicin Ib | – | – | + | – | – | – | – | – | – | – | – |
Иммунитет к колицину | Colicin Ib immunity | + | – | + | – | + | – | – | – | + | – | |||
K | cka | Колицин K | Colicin K | – | – | – | + | – | – | – | – | – | – | + |
cki | Иммунитет к колицину | Colicin K immunity | – | – | – | + | – | – | – | – | – | – | + | |
ckl | Белок лизиса | Lysis | – | – | – | + | – | – | – | – | – | – | + | |
M | cma | Колицин M | Colicin M | – | – | + | – | – | – | – | + | – | – | – |
cmi | Иммунитет к колицину | Colicin M immunity | – | – | + | – | – | – | – | + | – | – | – | |
Микроцин | Microcin | |||||||||||||
Класс I | Class I | |||||||||||||
B17 | mcbA | Микроцин B17 | Microcin B17 | – | – | – | – | – | – | + | – | – | – | – |
mcbG | Иммунитет к микроцину | Self-immunity | – | – | – | + | + | – | + | – | – | – | – | |
mcbF | Иммунитет к микроцину | Self-immunity | – | – | – | – | – | – | + | – | – | – | – | |
mcbE | Иммунитет к микроцину | Self-immunity | – | – | – | – | – | – | + | – | – | – | – | |
mcbD | Процессинг микроцина | Processing | – | – | – | – | – | – | + | – | – | – | – | |
mcbC | Процессинг микроцина | Processing | – | – | – | – | – | – | + | – | – | – | – | |
mcbB | Процессинг микроцина | Processing | – | – | – | – | – | – | + | – | – | – | – | |
C7 | mccA | Микроцин C7 | Microcin C7 | – | + | – | – | – | – | – | – | – | – | – |
mccF | Иммунитет к микроцину | Self-immunity | – | + | – | – | – | – | – | – | – | – | – | |
mccD | Посттрансляционные модификации Post-translation modifications | – | + | – | – | – | – | – | – | – | – | – | |
mccC | Экспорт микроцина | Export | – | + | – | – | – | – | – | – | – | – | – | |
mccE | Посттрансляционные модификации Post-translation modifications | – | + | – | – | – | – | – | – | – | – | – | |
mccB | Посттрансляционные модификации Post-translation modifications | – | + | – | – | – | – | – | – | – | – | – | |
J25 | mcjA | Микроцин J25 | Microcin J25 | – | – | – | – | – | – | + | + | – | – | – |
mcjD | Экспорт микроцина, иммунитет Export, self-immunity | – | – | – | – | – | – | + | + | – | – | – | |
Фактор Bacteriocin | Ген Gene | Функция Function | Штаммы E. coli | E. coli strain | ||||||||||
от здоровых добровольцев from healthy individuals | от пациентов с БК from patients with CD | ||||||||||||
1_34_12 | 1_39_1 | 1_45_11 | 1_18_1 | 1_25_1 | 1_25_14 | 36k_8 | 3_77_1 | 3_188_1 | 3_200_11 | 3_225_1 | |||
mcjB | Процессинг микроцина | Processing | – | – | – | – | – | – | + | + | – | – | – | |
mcjC | Процессинг микроцина | Processing | – | – | – | – | – | – | + | + | – | – | – | |
Класс IIa | Class IIa | |||||||||||||
V | cvaC | Колицин V | Colicin V | + | + | – | – | – | – | + | + | – | – | – |
cvi | Иммунитет к колицину | Self-immunity | – | – | – | – | – | – | – | – | – | – | – | |
cvaB | Синтез колицина | Synthesis | – | – | – | – | – | – | – | – | – | – | – | |
cvaA | Секреция колицина | Secretion | – | – | – | – | – | – | + | + | + | – | – | |
Класс IIb | Class IIb | |||||||||||||
H47 | mchB | Предшественник микроцина H47 Microcin H47 precursor | + | + | – | – | – | – | + | – | + | – | – |
mchC | Процессинг микроцина | Processing | + | + | – | – | – | – | + | – | + | – | – | |
mchD | Посттрансляционные модификации Post-translation modifications | + | + | – | – | – | – | + | – | + | – | – | |
mchE | Секреция микроцина | Secretion | + | + | – | – | – | – | + | – | + | – | – | |
mchF | Секреция микроцина | Secretion | + | + | – | – | – | – | + | – | + | – | – | |
mchX | Регуляции экспресии микроцина Regulation of expression | + | + | – | – | – | – | + | – | + | – | – | |
mchI | Иммунитет к микроцину | Self-immunity | + | + | – | – | – | – | + | – | + | – | – | |
I47 | mchS2 | Предшественник микроцина I47 Microcin I47 precursor | + | – | – | – | – | – | + | – | + | – | – |
mchS3 | Белок иммунитета к микроцину Self-immunity | + | – | – | – | – | – | + | – | + | – | – | |
mchS4 | Регуляция экспрессии микроцина Regulation of expression | + | + | – | – | – | – | + | – | + | – | – | |
M | mcmA | Предшественник микроцина M Microcin M precursor | + | + | – | – | – | – | – | – | – | – | – |
mcmI/ mcmA | Иммунитет к микроцину | Self-immunity | + | + | – | – | – | – | – | – | – | – | – | |
mcmK | Посттрансляционные модификации Post-translation modifications | + | – | – | – | – | – | + | – | + | – | – | |
mcmL | Посттрансляционные модификации Post-translation modifications | + | – | – | – | – | – | + | – | + | – | – | |
mcmM | Процессинг микроцина и экспорт Processing and export | + | + | – | – | – | – | + | – | + | – | – | |
Геномный анализ показал, что ни один из 11 штаммов не несёт кластеры генов систем прямого контактного взаимодействия CDI [7], а кластеры генов T6SS [43] обнаружены только в 1 штамме от здорового добровольца (36k_8). Поскольку данный штамм не содержит гены колицинов, а имеющиеся кластеры продукции микроцинов (В17, H47) неактивны на богатой питательной среде, использованной в исследовании, можно предположить, что конкуренция штамма с другими бактериями опосредована T6SS. Однако связь генов продукции бактериоцинов и T6SS с антагонистической активностью требует дальнейшего подтверждения путём оценки экспрессии генов обнаруженных систем либо путём детекции конечного продукта.
Среди других факторов, ассоциированных с выживанием бактерий в сообществе, только у штамма 1_39_1 обнаружен кластер генов pks, необходимый для биосинтеза и транспорта колибактина — вторичного метаболита, описанного преимущественно для патогенных штаммов семейства Enterobacteriaceae [44–46] и индуцирующего двуцепочечные разрывы ДНК в эукариотических клетках [47]. Однако инактивация гена clbA pks-острова в геноме пробиотического штамма E. coli Nissle 1917 приводит к существенному ослаблению его терапевтических свойств [48].
Для оценки адгезивного потенциала 11 штаммов были изучены варианты гена адгезина fimH, способствующего прикреплению бактериальной клетки к эпителиоцитам, поскольку некоторые точечные мутации в гене fimH (G73A/E/R/W, T158A/P, R166C/H/S) ассоциированы с фенотипической адгезией бактерий [49]. Среди всех штаммов только в гене fimH штамма 1_39_1 была обнаружена замена R166C/H, дающая конкурентное преимущество при колонизации кишечника.
Проведён филогенетический анализ штаммов, подавляющих рост нескольких протестированных представителей семейства Enterobacteriaceae. Оказалось, что штаммы 1_34_12 и 1_45_11 филогенетически близки лабораторному штамму K-12 substr. MG1655, а штамм 1_39_1 родственен известному пробиотическому штамму Nissle 1917 (рис. 3).
Рис. 3. Филограмма, построенная с помощью расстояний GBDP, рассчитанных на основе геномных последовательностей. Цифры представляют собой значения поддержки pseudo-bootstrap GBDP > 60% из 100 повторов при средней поддержке ветвей 62,2%. / Fig. 3. Phylogram constructed using the GBDP distances calculated from genomic sequences. Values in tree nodes represent pseudo-bootstrap GBDP support > 60% out of 100 repeats, with an average branch support of 62.2%.
Таким образом, в настоящем исследовании был проведён анализ микробных взаимодействий 11 клинических штаммов (4 от пациентов с БК и 7 от здоровых добровольцев) с некоторыми представителями семейства Enterobacteriaceae. В отличие от штаммов здоровых добровольцев, штаммы пациентов с БК не проявили антагонистической активности в отношении E. cloacae, K. pneumonia и S. enterica, несмотря на то что содержат гены, необходимые для продукции некоторых бактериоцинов, что, вероятно, связано с особенностями метода проведения анализа. Гены систем продукции бактериоцинов являются важными факторами колонизации кишечника бактерий и связаны с их пробиотическими свойствами [50]. В то же время данные кластеры генов обнаружены в геномах патогенных штаммов [5, 51], а также у пациентов с БК [51], что было подтверждено в данном исследовании. Штаммы здоровых добровольцев, проявившие антагонистическую активность, содержат не только гены систем продукции бактериоцинов, но и T6SS, осуществляющих прямое контактное взаимодействие между бактериями. Кроме того, выявлены 3 штамма (1_34_12, 1_39_1 и 1_45_11), способные подавлять рост нескольких протестированных представителей энтеробактерий. Данные штаммы обладают высоким конкурентным потенциалом и, вероятно, играют важную роль в колонизационной резистентности кишечника. Среди штаммов 1_34_12 и 1_45_11, филогенетически родственных штамму K-12 substr. MG1655, наибольший интерес представляет штамм 1_45_11, способный подавлять рост всех протестированных представителей энтеробактерий. Штамм 1_39_1, проявивший антагонистическую активность в отношении K. pneumonia и S. enterica, оказался филогенетически родственным известному пробиотическому штамму Nissle 1917. Кроме того, он содержит мутацию в гене адгезина fimH, повышающую адгезивную способность бактерий, и кластер генов системы продукции колибактина, обнаруженный ранее у Nissle 1917 [48].
Заключение
На примере E. coli были продемонстрированы различия в конкурентном потенциале 11 филогенетически родственных штаммов, заселяющих кишечник пациентов с БК и здоровых добровольцев. Интересно, что способность вытеснять in vitro несколько представителей конкурентных энтеробактерий была обнаружена у штаммов, выделенных у здоровых добровольцев. Вероятно, данные штаммы обладают важными свойствами, необходимыми для обеспечения колонизационной резистентности кишечника человека. Анализ биохимических характеристик не продемонстрировал различий в способности утилизировать субстраты среди E. coli пациентов и здоровых добровольцев, однако выявил свойства, отсутствующие у минорной группы штаммов и, вероятно, необходимые бактериям для успешной конкуренции в сообществе. Кроме того, в обеих группах были идентифицированы редко встречающиеся среди кишечных палочек штаммы. Интересно, что высококонкурентные штаммы не обладают уникальными особенностями, которые могли бы давать штаммам преимущество при колонизации кишечника. Сравнительный геномный анализ позволил выявить генетические детерминанты, дающие конкурентные преимущества бактериям обеих исследованных групп, а также выявить некоторые характеристики геномов штаммов здоровых добровольцев, обладающих высоким конкурентным потенциалом. Для понимания роли E. coli в поддержании кишечного гомеостаза и развитии воспалительных реакций желудочно-кишечного тракта необходимо дальнейшее изучение комплекса взаимодействий штаммов друг с другом и с организмом хозяина.
1 URL: https://www.ebi.ac.uk/Tools/msa/clustalo/
2 URL: https://tygs.dsmz.de
3 URL: https://www.ncbi.nlm.nih.gov/ (дата обращения: 01.07.2021).
About the authors
Maria N. Siniagina
Kazan Federal University
Author for correspondence.
Email: marias25@mail.ru
ORCID iD: 0000-0002-8138-9235
researcher, Research laboratory “Omics technologies”
Russian Federation, KazanAlexander V. Laikov
Kazan Federal University
Email: marias25@mail.ru
ORCID iD: 0000-0002-4250-2645
researcher, Research laboratory “Omics technologies”, Institute of Fundamental Medicine and Biology
Russian Federation, KazanMaria I. Markelova
Kazan Federal University
Email: marias25@mail.ru
ORCID iD: 0000-0001-7445-2091
researcher, Research laboratory “Omics technologies”, Institute of Fundamental Medicine and Biology
Russian Federation, KazanEugenia A. Boulygina
Kazan Federal University
Email: marias25@mail.ru
ORCID iD: 0000-0003-3458-1176
researcher, Research laboratory “Omics technologies”, Institute of Fundamental Medicine and Biology
Russian Federation, KazanDilyara R. Khusnutdinova
Kazan Federal University
Email: marias25@mail.ru
ORCID iD: 0000-0002-9982-9059
researcher, Research laboratory “Omics technologies”, Institute of Fundamental Medicine and Biology
Russian Federation, KazanSayar R. Abdulkhakov
Kazan Federal University
Email: marias25@mail.ru
ORCID iD: 0000-0001-9542-3580
Cand. Sci. (Med.), Head, Department of fundamentals of clinical medicine, senior researcher, Research laboratory “Omics technologies”, Institute of Fundamental Medicine and Biology
Russian Federation, KazanTatyana V. Grigoryeva
Kazan Federal University
Email: marias25@mail.ru
ORCID iD: 0000-0001-5314-7012
Cand. Sci. (Biol.), senior researcher, Research laboratory “Omics technologies”, Institute of Fundamental Medicine and Biology
Russian Federation, KazanReferences
- Das B., Nair G.B. Homeostasis and dysbiosis of the gut microbiome in health and disease. J. Biosci. 2019; 44(5): 1–8. https://doi.org/10.1007/s12038-019-9926-y
- Pessione E. The Russian doll model: how bacteria shape successful and sustainable inter-kingdom relationships. Front. Microbiol. 2020; 11: 2325. https://doi.org/10.3389/fmicb.2020.573759
- Freter R. Factors affecting the microecology of the gut. In: Probiotics. Dordrecht: Springer; 1992: 111–44. https://doi.org/10.1007/978-94-011-2364-8_6
- Stubbendieck R.M., Straight P.D. Multifaceted interfaces of bacterial competition. J. Bacteriol. 2016; 198(6): 2145–55. https://doi.org/10.1128/JB.00275-16
- Massip C., Oswald E. Siderophore-microcins in Escherichia coli: determinants of digestive colonization, the first step toward virulence. Front. Cell. Infect. Microbiol. 2020; 10: 381. https://doi.org/10.3389/fcimb.2020.00381
- Hayes C.S., Koskiniemi S., Ruhe Z.C., Poole S J., Low D.A. Mechanisms and biological roles of contact-dependent growth inhibition systems. Cold Spring Harb. Perspect. Med. 2014; 4(2): a010025. https://doi.org/10.1101/cshperspect.a010025
- Aoki S.K., Pamma R., Hernday A.D., Bickham J.E., Braaten B.A., Low D.A. Contact-dependent inhibition of growth in Escherichia coli. Science. 2005; 309(5738): 1245–8. https://doi.org/10.1101/cshperspect.a010025
- Russell A.B., Peterson S.B., Mougous J.D. Type VI secretion system effectors: poisons with a purpose. Nat. Rev. Microbiol. 2014; 12(2): 137–48. https://doi.org/10.1038/nrmicro3185
- Anderson M.C., Vonaesch P., Saffarian A., Marteyn B.S., Sansonetti P.J. Shigella sonnei encodes a functional T6SS used for interbacterial competition and niche occupancy. Cell Host Microbe. 2017; 21(6):769–76. https://doi.org/10.1016/j.chom.2017.05.004
- Endt K., Stecher B., Chaffron S., Slack E., Tchitchek N., Benecke A., et al. The microbiota mediates pathogen clearance from the gut lumen after non-typhoidal salmonella diarrhea PLoS Pathog. 2010; 6(9): e1001097. https://doi.org/10.1371/journal.ppat.1001097
- Kamada N., Seo S.U., Chen G.Y., Nunez G. Role of the gut microbiota in immunity and inflammatory disease Nat. Rev. Immunol. 2013; 13(5): 321–35. https://doi.org/10.1038/nri3430
- Abt M.C., Pamer E.G. Commensal bacteria mediated defenses against pathogens. Curr. Opin. Immunol. 2014; 29: 16–22. https://doi.org/10.1016/j.coi.2014.03.003
- Manichanh C., Rigottier-Gois L., Bonnaud E., Gloux K., Pelletier E., Frangeul L., et al. Reduced diversity of faecal microbiota in Crohn’s disease revealed by a metagenomic approach. Gut. 2006; 55(2): 205–11. https://doi.org/10.1136/gut.2005.073817
- Dicksved J., Halfvarson J., Rosenquist M., Järnerot G., Tysk C., Apajalahti J., et al. Molecular analysis of the gut microbiota of identical twins with Crohn's disease. ISME J. 2008; 2(7): 716–27. https://doi.org/10.1038/ismej.2008.37
- Frank D.N., Amand A.L., Feldman R.A., Boedeker E.C., Harpaz N., Pace N.R. Molecular-phylogenetic characterization of microbial community imbalances in human inflammatory bowel diseases. Proc. Nat. Acad. Sci. 2007; 104(34): 13780–5. https://doi.org/10.1073/pnas.0706625104
- Danilova N.A., Abdulkhakov S.R., Grigor'eva T.V., Markelova M.I., Vasil'ev I.Yu., Bulygina E.A., et al. Markers of dysbiosis in patients with ulcerative colitis and Crohn's disease. Terapevticheskiy arkhiv. 2019; (4): 13–20. https://doi.org/10.26442/00403660.2019.04.000211 (in Russian)
- Hewitt J.H., Rigby J. Effect of various milk feeds on numbers of Escherichia coli and Bifidobacterium in the stools of new-born infants. J. Hyg. 1976; 77(1): 129–39. https://doi.org/10.1017/S0022172400055601
- Bel'skiy V.V., Moskovtseva A.I. Colonization of Escherichia coli of the intestine and the frequency of detection of plasmid factors of multidrug resistance and colicinogenicity in children during the first year of life. Pediatriya. Zhurnal im. G.N. Speranskogo. 2007; 86(1): 20–4. (in Russian)
- Kamada N., Seo S.U., Chen G.Y., Nunez G. Role of the gut microbiota in immunity and inflammatory disease Nat. Rev. Immunol. 2013; 13(5): 321–35. https://doi.org/10.1038/nri3430
- Leimbach A., Hacker J., Dobrindt U. Escherichia coli as an all-rounder: the thin line between commensalism and pathogenicity. Curr. Top. Microbiol. Immunol. 2013; 358: 332. https://doi.org/10.1007/82_2012_303
- Lapaquette P., Glasser A.L., Huett A., Xavier R.J., Darfeuille‐Michaud A. Crohn's disease‐associated adherent‐invasive E. coli are selectively favoured by impaired autophagy to replicate intracellularly. Cell. Microbiol. 2010; 12(1): 99–113. https://doi.org/10.1111/j.1462-5822.2009.01381.x
- Siniagina M.N., Markelova M.I., Boulygina E.A., Laikov A.V., Khusnutdinova D.R., Abdulkhakov S.R., et al. Diversity and adaptations of Escherichia coli strains: exploring the intestinal community in Crohn’s disease patients and healthy individuals. Microorganisms. 2021; 9(6): 1299. https://doi.org/10.3390/microorganisms9061299
- Siniagina M., Markelova M., Laikov A., Boulygina E., Khusnutdinova D., Kharchenko A., et al. Cultivated Escherichia coli diversity in intestinal microbiota of Crohn's disease patients and healthy individuals: whole genome data. Data Brief. 2020; 28: 104948. https://doi.org/10.1016/j.dib.2019.104948
- Ohno M., Hasegawa M., Hayashi A., Caballero-Flores G., Alteri C.J., Lawley T.D., et al. Lipopolysaccharide O structure of adherent and invasive Escherichia coli regulates intestinal inflammation via complement C3. PLoS Pathog. 2020; 16(10): e1008928. https://doi.org/10.1371/journal.ppat.1008928
- Meier-Kolthoff J.P., Göker M. TYGS is an automated high-throughput platform for state-of-the-art genome-based taxonomy. Nat. Commun. 2019; 10(1): 2182. https://doi.org/10.1038/s41467-019-10210-3
- Meier-Kolthoff J.P., Auch A.F., Klenk H-P., Göker M. Genome sequence-based species delimitation with confidence intervals and improved distance functions. BMC Bioinform. 2013; 14: 60. https://doi.org/10.1186/1471-2105-14-60
- Lefort V., Desper R., Gascuel O. FastME 2.0: A comprehensive, accurate, and fast distance-based phylogeny inference program. Mol. Biol. Evol. 2015; 32(10): 2798–800. https://doi.org/10.1093/molbev/msv150
- Kreft L., Botzki A., Coppens F., Vandepoele K., Van Bel M. PhyD3: A phylogenetic tree viewer with extended phyloXML support for functional genomics data visualization. Bioinform. 2017; 33(18): 2946–7. https://doi.org/10.1093/bioinformatics/btx324
- David L.A., Maurice C.F., Carmody R.N., Gootenberg D.B., Button J.E., Wolfe B.E., et al. Diet rapidly and reproducibly alters the human gut microbiome. Nature. 2014; 505(7484): 559–63. https://doi.org/10.1038/nature12820
- De Filippo C., Cavalieri D., Di Paola M., Ramazzotti M., Poullet J.B., Massart S., et al. Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa. Proc. Natl Acad. Sci. USA. 2010; 107(33): 14691. https://doi.org/10.1073/pnas.1005963107
- Tyakht A.V., Alexeev D.G., Popenko A.S., Kostryukova E.S., Govorun V.M. Rural and urban microbiota: to be or not to be? Gut Microbes. 2014; 5(3): 351–6. https://doi.org/10.4161/gmic.28685
- Donaldson G.P., Lee S.M., Mazmanian S.K. Gut biogeography of the bacterial microbiota. Nat. Rev. Microbiol. 2016; 14(1): 20–32. https://doi.org/10.1038/nrmicro3552
- Pereira F.C., Berry D. Microbial nutrient niches in the gut. Environ. Microbiol. 2017; 19(4): 1366–78. https://doi.org/10.1111/1462-2920.13659
- Garrity G. Bergey's Manual of Systematic Bacteriology: Volu- me 2: The Proteobacteria, Part B: The Gammaproteobacteria. Springer Science & Business Media; 2007.
- Bingle L.E., Bailey C.M., Pallen M.J. Type VI secretion: a beginner’s guide. Curr. Opin. Microbiol. 2008; 11(1): 3–8. https://doi.org/10.1016/j.mib.2008.01.006
- Pomares M.F., Delgado M.A., Corbalán N.S., Farías R.N., Vincent P.A. Sensitization of microcin J25-resistant strains by a membrane-permeabilizing peptide. App. Environ. Microbiol. 2010; 76(20): 6837–42. https://doi.org/10.1128/AEM.00307-10
- Laviña M.A., Gaggero C., Moreno F. Microcin H47, a chromosome-encoded microcin antibiotic of Escherichia coli. J. Bacteriol. 1990; 172(11): 6585–8. https://doi.org/10.1128/jb.172.11.6585-6588.1990
- Chiuchiolo M.J., Delgado M.A., Farı́as R.N., Salomón R.A. Growth-phase-dependent expression of the cyclopeptide antibiotic microcin J25. J. Bacteriol. 2001; 183(5): 1755–64. https://doi.org/10.1128/JB.183.5.1755-1764.2001
- Connell N., Han Z., Moreno F., Kolter R. An E. coli promoter induced by the cessation of growth. Mol. Microbiol. 1987; 1(2): 195–201. https://doi.org/10.1111/j.1365-2958.1987.tb00512.x
- Kurepina N.E., Basyuk E.I., Metlitskaya A.Z., Zaitsev D.A., Khmel I.A. Cloning and mapping of the genetic determinants for microcin C51 production and immunity. Mol. Gen. Genet. 1993; 241(5-6): 700–6. https://doi.org/10.1007/BF00279914
- Schneider T., Hahn-Löbmann S., Stephan A., Schulz S., Giritch A., Naumann M., et al. Plant-made Salmonella bacteriocins salmocins for control of Salmonella pathovars. Sci. Rep. 2018; 8(1): 1–0. https://doi.org/10.1038/s41598-018-22465-9
- Mushtaq H., Bakht J., Khan I., Ahmad B. Antimicrobial efficacy and prevalence of colicinogenic E. coli in faecal matter of human, cow and sheep. Int. J. Antimicrob. Agents. 2021; 57(1): 106221. https://doi.org/10.1016/j.ijantimicag.2020.106221
- Sassone-Corsi M., Nuccio S.P., Liu H., Hernandez D., Vu C.T., Takahashi A.A., et al. Microcins mediate competition among Enterobacteriaceae in the inflamed gut. Nature. 2016; 540(7632): 280–3. https://doi.org/10.1038/nature20557
- Johnson J.R., Johnston B., Kuskowski M.A., Nougayrede J.P., Oswald E. Molecular epidemiology and phylogenetic distribution of the Escherichia coli pks genomic island. J. Clin. Microbiol. 2008; 46(12): 3906–11. https://doi.org/10.1128/JCM.00949-08
- Putze J., Hennequin C., Nougayrède J.P., Zhang W., Homburg S., Karch H., et al. Genetic structure and distribution of the colibactin genomic island among members of the family Enterobacteriaceae. Infect. Immun. 2009; 77(11): 4696–703. https://doi.org/10.1128/IAI.00522-09
- Suresh A., Ranjan A., Jadhav S., Hussain A., Shaik S., Alam M., et al. Molecular genetic and functional analysis of pks-harboring, extra-intestinal pathogenic Escherichia coli from India. Front. Microbiol. 2018; 9: 2631. https://doi.org/10.3389/fmicb.2018.02631
- Nougayrède J.P., Homburg S., Taieb F., Boury M., Brzuszkiewicz E., Gottschalk G., et al. Escherichia coli induces DNA double-strand breaks in eukaryotic cells. Science. 2006; 313(5788): 848–51. https://doi.org/10.1126/science.1127059
- Olier M., Marcq I., Salvador-Cartier C., Secher T., Dobrindt U., Boury M., et al. Genotoxicity of Escherichia coli Nissle 1917 strain cannot be dissociated from its probiotic activity. Gut Microb. 2012; 3(6): 501–9. https://doi.org/10.4161/gmic.21737
- Dreux N., Denizot J., Martinez-Medina M., Mellmann A., Billig M., Kisiela D., et al. Point mutations in FimH adhesin of Crohn's disease-associated adherent-invasive Escherichia coli enhance intestinal inflammatory response. PLoS Pathog. 2013; 9(1): e1003141. https://doi.org/10.1371/journal.ppat.1003141
- Drider D. Gut microbiota is an important source of bacteriocins and their in situ expression can be explored for treatment of bacterial infections. Prob. Antimicrob. Proteins. 2021; 13(6): 1759–65. https://doi.org/10.1007/s12602-021-09843-y
- Šmajs D., Micenková L., Šmarda J., Vrba M., Ševčíková A., Vališová Z., et al. Bacteriocin synthesis in uropathogenic and commensal Escherichia coli: colicin E1 is a potential virulence factor. BMC Microbiol. 2010; 10: 288. https://doi.org/10.1186/1471-2180-10-288
- Micenková L., Frankovičová L., Jaborníková I., Bosák J., Dítě P., Šmarda J., et al. Escherichia coli isolates from patients with inflammatory bowel disease: ExPEC virulence-and colicin-determinants are more frequent compared to healthy controls. Int. J. Med. Microbiol. 2018; 308(5): 498–504. https://doi.org/10.1016/j.ijmm.2018.04.008
Supplementary files