DNA-protein COVID-19 combination vaccine containing multiepitope T-cell immunogen and receptor binding domain of the SARS-CoV-2 S protein
- Authors: Borgoyakova M.B.1, Rudometov A.P.1, Starostina E.V.1, Yakovlev V.A.1, Tigeeva E.V.1, Zaykovskaya A.V.1, Volosnikova E.A.1, Merkuleva I.A.1, Isaeva A.A.1, Nesmeyanova V.S.1, Shanshin D.V.1, Zaitsev B.N.1, Kisakov D.N.1, Kisakova L.A.1, Shcherbakov D.N.1, Karpenko L.I.1, Ilyichev A.A.1
-
Affiliations:
- State Research Center of Virology and Biotechnology «Vector»
- Issue: Vol 102, No 5 (2025)
- Pages: 571-582
- Section: ORIGINAL RESEARCHES
- URL: https://microbiol.crie.ru/jour/article/view/18834
- DOI: https://doi.org/10.36233/0372-9311-691
- EDN: https://elibrary.ru/WKWCLK
- ID: 18834
Cite item
Full Text
Abstract
Introduction. During the COVID-19 pandemic, the development of preventive vaccines, including those based on new platforms, became extremely relevant. One such platform is vaccines, which combine, for example, DNA and protein components into a single vaccine.
The aim of this study was to investigate the immunogenicity of a DNA vaccine encoding a polyepitopic T-cell immunogen of the SARS-CoV-2 virus, combined with the recombinant RBD protein (the receptor-binding domain of the SARS-CoV-2 virus S protein, Wuhan-Hu-1 strain) conjugated to a polycationic carrier – polyglucin-spermidine (PGS), and to assess the contribution of individual components to the development of an immune response in BALB/c mice.
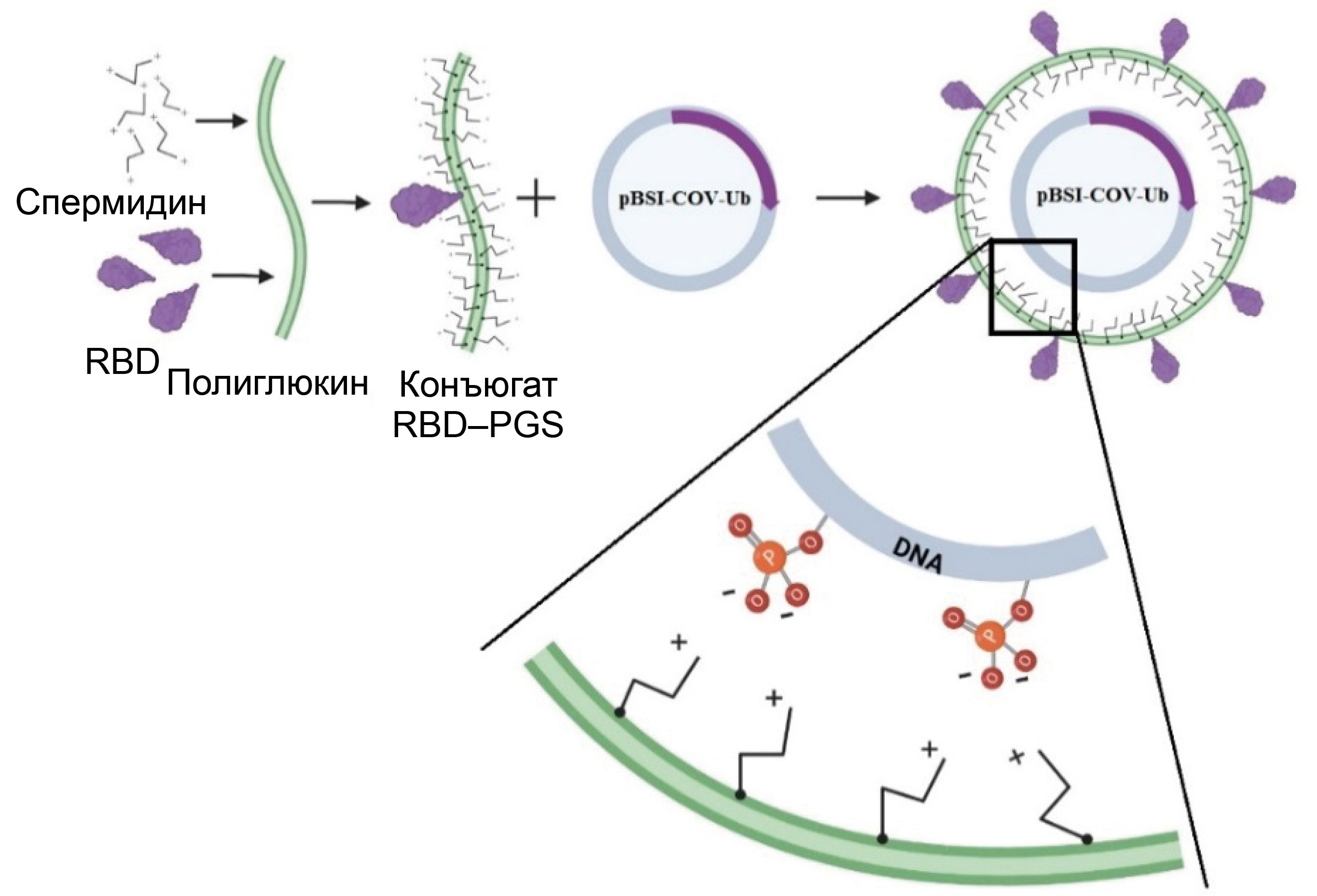
Materials and methods. To create the DNA vaccine (pBSI-COV-Ub), we used a strategy of designing an artificial polyepitope immunogen consisting of conserved immunodominant fragments of various structural proteins of the SARS-CoV-2 virus, containing a large number of T-lymphocyte epitopes: helper and cytotoxic. The recombinant RBD protein was conjugated with the polycation PGS, and upon mixing it with DNA, it formed the vaccine complex CCV–BSI, whose immunogenic properties were investigated in this work.
Results. Immunization of BALB/c mice with the CCV–BSI combined construct resulted in the induction of high antibody titers with neutralizing activity against live SARS-CoV-2 virus, as well as the formation of a virus-specific T-cell response, as demonstrated by ELISA, neutralization assay and ELISpot. It has been shown that the protein component contributes to the humoral immune response, while DNA contributes to the cellular immune response. Administration of the recombinant RBD protein led to the induction of only antibodies, administration of the DNA vaccine led to the induction of only a T-cell response, and administration of the combined preparation led to the induction of both a humoral immune response and specific T cells.
Conclusion. The unique combination of DNA and protein within a single vaccine construct allows for overcoming the limitations of each of these vaccine types and leads to the induction of both arms of immunity. The protein component can be replaced according to the current viral strain, and a universal T-cell immunogen can provide a response to a wide range of circulating variants. This platform can be further used to develop vaccines against various highly variable viruses.
Full Text
Введение
Пандемия COVID-19 привела к принятию беспрецедентных мер в области мирового здравоохранения. В частности, для массовой вакцинопрофилактики в условиях чрезвычайной ситуации были одобрены вакцины, основанные на инновационных подходах: мРНК-вакцины [1], ДНК-вакцины [2], вакцины на основе вирусных векторов [3, 4]. Установлено, что вакцины данных типов способны приводить к индукции Т-клеточных ответов, важных для противостояния вирусной инфекции.
Для родственных SARS-CoV-2 вирусов SARS-CoV (коронавирус, вызывающий острый респираторный синдром) и MERS-CoV (коронавирус, вызывающий ближневосточный респираторный синдром) было показано, что вирус-специфический Т-клеточный ответ сохраняется спустя десятилетия, тогда как нейтрализующие титры сывороток значительно падают уже через полгода после перенесённого заболевания [5, 6]. Снижение нейтрализующей активности сывороток показано и при появлении новых штаммов вируса SARS-CoV-2 [7]. Стоит отметить, что долго сохраняющийся Т-клеточный ответ является специфичным не только к основному антигену — гликопротеину S, главной мишени гуморального иммунного ответа, но и к другим белкам, обычно не используемым в качестве иммуногенов в субъединичных вакцинах [8]. Поскольку белки N, M, E и другие меньше подвержены накоплению мутаций [9], то их последовательности более консервативны [10], и Т-клеточный иммунный ответ, направленный на их эпитопы, может быть специфичным в отношении вновь возникающих штаммов [11]. Актуальна стратегия создания искусственных Т-клеточных полиэпитопных иммуногенов, содержащих иммунодоминантные эпитопы вирусных/бактериальных/опухолевых белков, узнаваемые CD4+- и CD8+-Т-клетками. Платформа ДНК-вакцин способна обеспечить стимуляцию цитотоксических и Т-хелперных лимфоцитов за счёт внутриклеточного синтеза и процессинга иммуногена с последующей презентацией его антигенпрезентирующими клетками в комплексе с молекулами главного комплекса гистосовместимости (MHC) I и II классов.
Однако в случае SARS-CoV-2 не стоит забывать о важности гуморального иммунного ответа, т. к. высокие титры нейтрализующих антител коррелируют с эффективностью вакцины [12]. Наиболее подходящими иммуногенами, индуцирующими наработку антител, являются рекомбинантные белки [13]. Они ограничены в стимуляции специфического цитотоксического ответа, но активируют B-клетки и Т-хелперы, особенно хорошо в присутствии адъювантов [14]. Такими адъювантами могут быть соли алюминия, различные эмульсии, полисахариды. Один из наиболее изученных α-глюканов, который используется для доставки лекарств, — полиглюкин (высокомолекулярный декстран) [15]. Полиглюкин обеспечивает пролонгированное высвобождение активного вещества, а также является иммуномодулятором, в результате чего работает в качестве адъюванта. Благодаря наличию гидроксильных групп полиглюкин легко подвергается модификации, в частности присоединению веществ, содержащих аминогруппы, таких как белки [16].
Объединение разных типов иммуногенов (ДНК и белок) в одной конструкции представляется перспективным подходом, позволяющим преодолеть ограничения каждого из данных типов вакцин и привести к индукции обоих звеньев иммунитета. В большинстве работ, в которых исследуется возможность комбинирования разных иммуногенов, иммунизация проводится с применением прайм-буст-стратегий, при которых чаще всего первую иммунизацию проводят ДНК-вакциной, а бустерную — белком в комплексе с адъювантом [17]. В некоторых работах показано, что совместное введение ДНК и белка приводит к увеличению иммуногенности как по сравнению с группами, получающими компоненты в отдельности, так и с группами, получающими вакцины в системе прайм-буст [18–21]. Так, введение смеси ДНК-вакцины, кодирующей S-белок вируса SARS-CoV-2, и рекомбинантного белка S в сочетании с адъювантом в виде алюминиевых квасцов привело к формированию выраженного защитного иммунного ответа у сирийских хомяков, значительно превышающего значения в группах животных, иммунизированных отдельными компонентами [21]. Также было показано, что одновременное введение ДНК и белка в одно и то же место является более эффективным, чем введение их одновременно, но в разные места (например, в разные лапы) [19].
Для других вакцин, индуцирующих Т-клеточный ответ, таких как мРНК или векторные вакцины, совместное введение с белком в работах не встречается, однако есть исследования, посвящённые положительному эффекту бустирования иммунного ответа на данные типы вакцин субъединичными препаратами [22–25].
Целью данной работы являлось исследование иммуногенности ДНК-вакцины, кодирующей полиэпитопный Т-клеточный иммуноген вируса SARS-CoV-2, в сочетании с рекомбинантным белком RBD (рецептор-связывающий домен белка S вируса SARS-CoV-2), конъюгированным с поликатионным носителем — полиглюкин-спермидином (PGS), а также оценка вклада отдельных компонентов в развитие иммунного ответа у мышей линии BALB/c.
Материалы и методы
Конструирование ДНК-вакцины pBSI-COV
В качестве ДНК-компонента использовали полученную нами ранее плазмиду pBSI-CoV-Ub [26]. Предсказание Т-клеточных эпитопов было выполнено с использованием программы NetMHCpan-4.1 и базы данных Immune Epitope Database 2.22, IEDB 2.22. В качестве анализируемых последовательностей из GenBank были взяты последовательности белков S, N, M и E (GenBank MN908947), полученные в результате секвенирования вируса SARS-CoV-2 штамма Wuhan-Hu-1. Фрагменты, выбранные для исследования, анализировали на консервативность, используя базу данных GISAID, затем их объединили последовательно в одну конструкцию, к N-концу добавили убиквитин и эпитоп PADRE (Pan DR Epitope, универсальный Т-хелперный эпитоп, усиливающий индукцию CD4+-Т-клеток, регулирующих B-клетки и цитотоксические Т-лимфоциты), к C-концу — маркерный эпитоп EPFRDYVDRFYKTLR. Кодирующую генетическую последовательность оптимизировали для эффективной трансляции в клетках млекопитающих с помощью программы Jcat (http://www.jcat.de). Синтез гена был проведён фирмой «ДНК-синтез». Клонирование в вектор pVAX1 провели по сайтам PspLI и ApaI.
Экспрессию целевого гена оценивали с помощью полимеразной цепной реакции с обратной транскрипцией (ОТ-ПЦР) и иммуноблотинга [26].
Получение комбинированного ДНК-белкового препарата
Белок RBD и его конъюгат с PGS были получены, как описано ранее [27]. В качестве источника последовательности RBD использовали геномные данные изолята Wuhan-Hu-1 (GenBank: MN908947.3). Комбинированный ДНК-белковый препарат получали путём смешивания 2 частей плазмидной ДНК с 1 частью белка RBD, конъюгированного с PGS (в массовом соотношении ДНК и белка), а затем добавляли избыток PGS в массовом соотношении 1 : 10 относительно ДНК. Формирование комплексов оценивали по смещению электрофоретической подвижности в 1% агарозном геле, а также с помощью электронной микроскопии: окрашивание 2% водным раствором уранилацетата, микроскоп «JEM-1400» («Jeol»).
Иммунизация животных, подготовка образцов
Работу с животными проводили согласно «Руководству по уходу и использованию лабораторных животных» и принципам гуманности, изложенным в директивах Европейского сообщества (86/609/EEC) и Хельсинкской декларации. Протоколы были одобрены Биоэтической комиссией ГНЦ ВБ «Вектор» (протокол № 1 от 21.03.2023).
Для оценки иммуногенности созданных конструкций использовали самок мышей BALB/c массой 16–18 г, полученных из питомника ГНЦ ВБ «Вектор». Животных разделили на группы по 6 мышей и иммунизировали внутримышечно дважды с интервалом 3 нед. Каждая мышь получала 200 мкл инъекционного препарата в обе бедренные мышцы задних лап (по 100 мкл в каждую):
- группа CCV–BSI — комбинированный препарат, содержащий 100 мкг ДНК и 50 мкг белка;
- группа pBSI-COV-Ub — 100 мкг плазмиды;
- группа RBD–PGS — 50 мкг белка RBD, конъюгированного с PGS;
- группа интактные — неиммунизированные животные.
Спустя 2 нед после 2-й иммунизации у мышей брали кровь для анализа гуморального иммунного ответа и селезёнки для анализа клеточного ответа.
Сыворотки отделяли от клеточных элементов центрифугированием (9000g, 15 мин), прогревали 30 мин при 56°С и исследовали на наличие антител, специфически связывающихся с белком RBD в иммуноферментном анализе (ИФА), а также анализировали их вируснейтрализующую активность.
Селезёнки последовательно измельчали на нейлоновых фильтрах для клеток с диаметром пор 70 и 40 мкм («JET Biofil»). Подготовку спленоцитов проводили, как описано ранее [27].
Иммуноферментный анализ
Рекомбинантный белок RBD сорбировали на 96-луночные планшеты («Nunc») в 2М мочевине при 4°С в течение ночи (1 мкг/мл). Постановку ИФА проводили, как описано ранее [27]. Результаты анализировали при длине волны 450 нм с помощью спектрофотометра «Feyond A-300» («Allsheng»).
Реакция вируснейтрализации
Нейтрализующие свойства антител сывороток крови анализировали в реакции ингибирования цитопатического действия вируса на культуре клеток in vitro, как описано ранее [27]. В работе использовали штамм коронавируса SARS-CoV-2 nCoV/Victoria/1/2020, полученный из Государственной коллекции возбудителей вирусных инфекций и риккетсиозов ГНЦ ВБ «Вектор». Нейтрализующую активность сывороток иммунизированных животных оценивали по разведению сывороток, при котором регистрировали защиту клеток от цитопатического действия вируса в 50% лунок.
ELISpot
ELISpot проводили с использованием набора ELISpot Plus: Mouse IFN-γ (ALP) («Mabtech») по инструкции производителя. Спленоциты пассировали в количестве 2,5 × 105 клеток/лунка и добавляли к ним среду RPMI с 10% эмбриональной телячьей сыворотки (для отрицательного контроля), или смесь вирус-специфических пептидов в концентрации 20 мкг/мл для каждого пептида, или конканавалин A (для положительного контроля). Подсчёт спотов производили визуально с использованием ELISpot-ридера («Carl Zeiss»). Единицу образования пятен (SFU) на 106 клеток рассчитывали путём вычитания значений в лунках с отрицательным контролем.
Пептиды для стимуляции определяли с помощью инструментов IEDB Analysis Resourse и синтезировали в компании «AtaGenix Laboratories». Более 80% составила чистота пептидов: VYAWNRKRI, FERDISTEI, CGPKKSTNL, RFASVYAWNRKRISN, VGGNYNYLYRLFRKS, GGNYNYLYRLFRKSN, YNYKLPDDFTGCVIA, KNKCVNFNFNGLTGT, QPTESIVRF, VSPTKLNDL, LLHAPATVCGPKKST, ASVYAWNRKRISN, YNYLYRLFRKSNL, AYSNNSIAI, QYIKWPWYI, LPPLLTDEM, SAPHGVVFL, WPWYIWLGF, YYRRATRRIRGGDGK, GTWLTYTGAIKLDDK, DDQIGYYRRATRRIR, VKPSFYVYSRVKNLN, CFVLAAVYRINWITG, YYRRATRRI, TPSGTWLTY, KHIDAYKTF, SPDDQIGYY.
Статистическая обработка
Данные были проанализированы с помощью программного обеспечения «GraphPadPrism 6.0». Различия между группами определяли с использованием непараметрического метода Манна–Уитни, при p < 0,05 различия считали значимыми.
Результаты
Дизайн искусственного полиэпитопного Т-клеточного иммуногена
Дизайн Т-клеточного иммуногена осуществляли с использованием подхода, позволяющего выбрать иммунодоминантные эпитопы, а затем объединить их в одну последовательность. По итогам анализа были выбраны 5 фрагментов из белка S, по 2 фрагмента из белков M и N, 1 фрагмент из белка E, которые содержат наибольшее количество CD4+- и CD8+-эпитопов, рестриктируемых молекулами MHC I и II классов человека и мыши. Анализ гомологичности данных фрагментов для разных штаммов вируса, включая актуальные для 2024 г. варианты, показал высокую степень их консервативности (85,4–100%). К конструкции для увеличения эффективности внутриклеточного процессинга синтезируемого белка была добавлена последовательность, кодирующая убиквитин. Аминокислотные последовательности выбранных фрагментов и общая структура иммуногена представлены на рис. 1. Сконструированный иммуноген назван BSI-COV-Ub.
Рис. 1. Схематическое изображение искусственного полиэпитопного Т-клеточного иммуногена BSI-COV-Ub, составленного из фрагментов белков вируса SARS-CoV-2.
Плазмида pBSI-COV-Ub (рис. 2, а) была наработана в препаративных количествах и охарактеризована ранее [26]. С помощью ОТ-ПЦР и вестерн-блоттинга определили экспрессию целевого гена, кодирующего искусственный иммуноген BSI-COV-Ub, на уровне РНК и белка. По данным ОТ-ПЦР, размеры амплифицированного фрагмента составили ~ 1470 п. о., что соответствует теоретически рассчитанному фрагменту при использовании специфических праймеров (рис. 2, б). С помощью иммуноблоттинга выявили дискретные белки, из которых продукт с наибольшей массой соответствовал теоретически рассчитанному продукту гена bsi-cov-ub (54,5 кДа; рис. 2, в). Наличие «лесенки» из дискретных белков свидетельствует об эффективном процессинге Т-клеточного иммуногена в клетке.
Рис. 2. Структура плазмиды pBSI-CoV-Ub (а) и анализ экспрессии целевого гена после трансфекции клеток HEK293T.
б — продукты ОТ-ПЦР с использованием тотальной РНК клеток HEK293T, трансфицированных плазмидой pBSI-CoV-Ub (дорожка 1) (электрофорез в 1% агарозном геле). На дорожке 2 — продукты ПЦР, поставленных на препарате тотальной РНК без обратной транскрипции.
в — белковые продукты, выявленные в трансфицированных плазмидой pBSI-CoV-Ub клетках HEK293T методом иммуноблоттинга с использованием моноклонального 29F2 к маркерному эпитопу.
Рекомбинантный белок RBD и его конъюгат с PGS были наработаны, очищены и охарактеризованы ранее [27].
Получение комплекса ДНК–белок
Получение комбинированного комплекса из ДНК и конъюгата RBD–PGS проводили путём смешивания компонентов, схема их взаимодействия в смеси представлена на рис. 3, а. Формирование комплекса определяли по изменению электрофоретической подвижности плазмидной ДНК в агарозном геле: инкапсулированная плазмида демонстрировала значительно меньшую подвижность в электрическом поле по сравнению с «голой плазмидой» (рис. 3, б).
Рис. 3. Комплекс ДНК–белок.
а — схематическое представление комплекса: отрицательные группы остатков фосфорной кислоты в составе плазмидной ДНК электростатически связываются с положительными аминогруппами в составе спермидина, конъюгированного с полиглюкином.
б — подтверждение инкапсуляции ДНК в оболочке RBD–PGS с помощью электрофореза в 1% агарозном геле: 1 — CCV–BSI; 2 — pBSI-COV-Ub.
в — электронные микрофотографии частиц CCV–BSI.
С помощью электронной микроскопии (рис. 3, в) было показано, что размеры частиц находятся в диапазоне 50–200 нм. Ряд исследователей предполагают, что наночастицы подобных размеров оптимальны для создания вакцин, поскольку они накапливаются в В-клеточных фолликулах и вызывают сильный иммунный ответ [28].
Анализ гуморального иммунного ответа
Для оценки иммуногенности созданных вакцинных конструкций мышей BALB/c иммунизировали дважды в дни 0, 21 и в конечной точке (день 35), производили забор сывороток для анализа. Сыворотки тестировали на наличие RBD-специфических антител с помощью ИФА, а также на способность нейтрализовать живой вирус. В качестве контролей использовали сыворотки мышей, иммунизированных плазмидной ДНК pBSI-COV-Ub и белком RBD, конъюгированным с PGS, а также сыворотки интактных мышей (рис. 4, а).
Рис. 4. Гуморальный иммунный ответ у мышей BALB/c, иммунизированных конструкциями CCV–BSI, pBSI-COV-Ub и RBD–PGS. Интактные, неиммунизированные мыши — отрицательный контроль.
а — схематическое изображение эксперимента по исследованию иммуногенности: мышей иммунизировали дважды с интервалом 3 нед, через 2 нед после 2-й иммунизации у мышей забирали образцы для анализа.
б — титры специфических IgG к RBD SARS-CoV-2, определённые в ИФА.
в — титры вируснейтрализующих антител, определённые с использованием штамма SARS-CoV-2 nCoV/Victoria/1/2020.
Данные фрагментов б и в представлены как геометрические средние обратных титров со стандартным геометрическим отклонением, индивидуальные значения отмечены точками. Значимость различий между группами рассчитывали с использованием непараметрического метода Манна–Уитни (*p < 0,01, **p < 0,05, n.s. — статистическая значимость отсутствует).
По результатам RBD-специфического ИФА через 2 нед после 2-й иммунизации средние титры специфических антител у животных, иммунизированных комбинированной вакциной CCV–BSI, сочетающей в себе белок и ДНК, составили 1 : 1 557 215. В группе, иммунизированной белком RBD, конъюгированным с PGS, средний титр составил 1 : 391 951, что достоверно не отличается от первой группы (p = 0,2739). В сыворотках мышей, получавших только плазмиду pBSI-COV-Ub, и в сыворотках интактных животных антитела, специфически связывающиеся с белком RBD, были обнаружены на уровне фона (рис. 4, б).
Результаты анализа нейтрализующих свойств сывороток с использованием штамма nCoV/Victoria/1/2020 вируса SARS-CoV-2 на культуре клеток in vitro также показали отсутствие значимых различий между группами, иммунизированными комбинированной конструкцией CCV–BSI и белком RBD, конъюгированным с PGS: средние нейтрализующие титры составили 1 : 238 и 1 : 263 соответственно (p = 0,7780). Cыворотки животных, получавших только плазмиду pBSI-COV-Ub, и сыворотки интактных животных не проявили вируснейтрализующей активности (рис. 4, в).
Анализ клеточного иммунного ответа
Для оценки способности созданных конструкций индуцировать клеточный иммунитет у мышей в конечной точке эксперимента брали селезёнки, которые были гомогенизированы и исследованы с помощью метода ELISpot. В качестве контролей использовали спленоциты мышей, иммунизированных плазмидной ДНК pBSI-COV-Ub и белком RBD, конъюгированным с PGS, а также спленоциты интактных мышей. Ответ оценивали по способности спленоцитов отвечать выделением интерферона-γ (IFN-γ) на специфическую стимуляцию, которую осуществляли пулом пептидов, входящих в состав белка RBD и иммуногена BSI-COV-Ub (рис. 5, а).
Рис. 5. Клеточный иммунный ответ у мышей BALB/c.
а — изображения репрезентативных лунок в ELISpot.
б — количество спленоцитов, высвобождающих интерферон-γ в ответ на специфическую стимуляцию пептидами из белка RBD и искусственного иммуногена BSI-COV-Ub, подсчитанное с помощью ELISpot. Данные представлены как геометрические средние со стандартным геометрическим отклонением, индивидуальные значения отмечены точками. Значимость различий между группами рассчитывали с использованием непараметрического метода Манна–Уитни (*p < 0,01, **p < 0,05, n.s. — статистическая значимость отсутствует).
С помощью ELISpot было показано, что наиболее высокие показатели клеточного иммунитета достигнуты в группах, иммунизированных ДНК-вакциной или комбинированным комплексом (рис. 5, б). Так, среднее количество спленоцитов, ответивших на стимуляцию, в обеих группах составило 46 и 54 SFU на 106 клеток соответственно (p > 0,9999 между группами и p < 0,01 по сравнению с двумя другими группами). В группе, получавшей конъюгат RBD–PGS, был зарегистрирован низкий ответ на уровне отрицательного контроля (2 SFU).
Обсуждение
Распространение SARS-CoV-2 в масштабах планеты потребовало быстрой разработки вакцины. Глобальная кампания по вакцинации населения предоставила уникальную возможность сравнить различные стратегии и платформы разработанных вакцин. Практически все имеющиеся у исследователей подходы были задействованы в разработке, и в результате на рынок вышли не только классические типы вакцин (инактивированные, субъединичные), но и вакцины нового поколения (мРНК, векторные, ДНК-вакцины).
Критически важным является не только индукция гуморального иммунного ответа и вируснейтрализующих антител, в частности, но и активация вирус-специфического Т-клеточного ответа [29]. В данной работе для создания вакцинной конструкции мы объединили две платформы: ДНК-вакцину и рекомбинантный белок. Полученная смесь представляет собой ДНК, окружённую положительно заряженными молекулами PGS, конъюгированного с белком RBD. В качестве антигена выбрали RBD, потому что эта область белка S является доминирующей мишенью для нейтрализующего ответа при инфекции COVID-19.
Ранее мы разработали полиэпитопный искусственный иммуноген, содержащий CTL- и Th-эпитопы из белков S, N, M и E вируса SARS-CoV-2 (рис. 1), и получили ДНК-вакцину pBSI-COV-Ub, кодирующую данный иммуноген (рис. 2) [26]. Фрагменты, выбранные для включения в итоговую конструкцию, содержат эпитопы, рестриктируемые широким спектром MHC I и II классов человека и мыши, подтвердившие иммунодоминантность в исследовании SARS-CoV-2-специфичного клеточного иммунитета и являющиеся консервативными для разных вариантов вируса [30–32]. После объединения фрагментов в одну последовательность к её N-концу добавили убиквитин. Присоединение убиквитина к белку способствует нацеливанию его на протеасому, что приводит к эффективному процессингу, высвобождению пептидов-эпитопов, которые презентируются MHC I класса на поверхности антигенпрезентирующей клетки и способствуют активации цитотоксических Т-лимфоцитов [33], а также в результате кросс-презентации активируют Т-хелперы.
Используемый в работе конъюгат PGS ранее был использован нами для доставки кандидатных ДНК-вакцин против ВИЧ-1, Эбола и COVID-19 [27, 34, 35]. В исследованиях данных конструкций были доказаны их безопасность и эффективное усиление иммуногенности ДНК-вакцин за счёт использования PGS. PGS защищает ДНК от воздействия нуклеаз, а также способствует пролонгированному высвобождению иммуногена и оказывает иммуномодулирующее воздействие [15, 16]. Компоненты PGS биоразлагаемы и позволяют лиофилизировать препарат вакцины. Конъюгат RBD–PGS обладает положительным зарядом и при добавлении ДНК начинает взаимодействовать с её отрицательно заряженными остатками фосфорной кислоты, образуя комплекс (рис. 3, а). Формирование комплексов было подтверждено снижением подвижности ДНК в агарозном геле (рис. 3, б).
Исследование гуморального иммунного ответа у мышей линии BALB/c на двукратное внутримышечное введение комплекса CCV–BSI показало, что комбинированная конструкция наравне с белком RBD, конъюгированным с PGS, индуцирует наработку высоких титров специфических антител, обладающих нейтрализующей активностью против штамма nCoV/Victoria/1/2020 вируса SARS-CoV-2 на культуре клеток in vitro (рис. 4, а, б). Рекомбинантный белок RBD без адъюванта обладает менее выраженной иммуногенностью по сравнению с конъюгатом [36]. Более высокий RBD-специфический ответ в группе, иммунизированной CCV–BSI, по сравнению с группой, иммунизированной RBD–PGS (средний титр 1 : 1,5 млн против 1 : 0,3 млн), может говорить о вкладе мультимеризации антигена, возникающем из-за взаимодействия ДНК с несколькими молекулами конъюгата RBD–PGS, а также о стимуляции специфического Т-хелперного ответа за счёт ДНК-вакцины. Подобный синергический эффект в отношении индукции гуморального иммунного ответа был отмечен большинством исследователей, изучавших совместное введение ДНК- и субъединичной вакцины [18–20, 34].
При оценке клеточного ответа с помощью определения количества спленоцитов, продуцирующих IFN-γ в ответ на стимуляцию вирусными пептидами, методом ELISpot наиболее высокий уровень специфического клеточного иммунитета был обнаружен в группах, иммунизированных препаратами, содержащими ДНК-конструкцию pBSI-COV-Ub. Это говорит о том, что ДНК-вакцина и в свободном состоянии, и в состоянии инкапсуляции способна индуцировать специфический Т-клеточный иммунный ответ против широкого спектра вирусных штаммов, тогда как при иммунизации белком RBD, конъюгированным с PGS, индукции Т-клеточного ответа не происходит (рис. 5). Во многих работах использование совместно с субъединичными вакцинами ДНК-вакцин приводит к индукции Т-клеточного ответа. Причём его развитие незначительно зависит от режима введения, т. е. ДНК-вакцина как в качестве прайм-иммунизации, так и при введении совместно с белком стимулирует Т-клетки на уровне, сопоставимом с введением только ДНК-вакцины [20, 21, 34].
Выводы
Уникальная комбинация ДНК и белка в составе одной вакцинной конструкции позволяет преодолеть ограничения каждого из данных типов вакцин и приводит к индукции обоих звеньев иммунитета. Белковый компонент может быть заменён в соответствии с актуальным вирусным штаммом, а универсальный Т-клеточный иммуноген может обеспечить ответ на широкий спектр циркулирующих вариантов. Данная платформа в дальнейшем может быть использована для разработки вакцин против различных вирусов с высокой изменчивостью.
About the authors
Mariya B. Borgoyakova
State Research Center of Virology and Biotechnology «Vector»
Author for correspondence.
Email: borgoyakova_mb@vector.nsc.ru
ORCID iD: 0000-0002-0768-1561
Cand. Sci. (Biol.), senior researcher, Department of bioengineering
Russian Federation, Koltsovo, Novosibirsk regionAndrey P. Rudometov
State Research Center of Virology and Biotechnology «Vector»
Email: rudometov_ap@vector.nsc.ru
ORCID iD: 0000-0003-2808-4309
Cand. Sci. (Biol.), leading researcher, Acting Head, Department of bioengineering
Russian Federation, Koltsovo, Novosibirsk regionEkaterina V. Starostina
State Research Center of Virology and Biotechnology «Vector»
Email: starostina_ev@vector.nsc.ru
ORCID iD: 0000-0002-1733-9524
Cand. Sci. (Biol.), senior researcher, Department of bioengineering
Russian Federation, Koltsovo, Novosibirsk regionVladimir A. Yakovlev
State Research Center of Virology and Biotechnology «Vector»
Email: yakovlev_va@vector.nsc.ru
ORCID iD: 0009-0009-4563-2099
junior researcher, Department of bioengineering
Russian Federation, Koltsovo, Novosibirsk regionElena V. Tigeeva
State Research Center of Virology and Biotechnology «Vector»
Email: tigeeva_ev@vector.nsc.ru
ORCID iD: 0009-0001-5203-2130
junior researcher, Department of bioengineering
Russian Federation, Koltsovo, Novosibirsk regionAnna V. Zaykovskaya
State Research Center of Virology and Biotechnology «Vector»
Email: zaykovskaya_av@vector.nsc.ru
ORCID iD: 0000-0002-0450-5212
Cand. Sci. (Biol.), senior researcher, Department “Collection of microorganisms”
Russian Federation, Koltsovo, Novosibirsk regionEkaterina A. Volosnikova
State Research Center of Virology and Biotechnology «Vector»
Email: volosnikova_av@vector.nsc.ru
ORCID iD: 0000-0001-5028-5647
Cand. Sci. (Biol.), leading researcher, Head, Laboratory of the production and analysis of biosubstances
Russian Federation, Koltsovo, Novosibirsk regionIuliia A. Merkuleva
State Research Center of Virology and Biotechnology «Vector»
Email: merkuleva_iua@vector.nsc.ru
ORCID iD: 0000-0002-6974-0686
Cand. Sci. (Biol.), senior researcher, Department of biophysics and environmental research
Russian Federation, Koltsovo, Novosibirsk regionAnastasya A. Isaeva
State Research Center of Virology and Biotechnology «Vector»
Email: isaeva_aa@vector.nsc.ru
ORCID iD: 0000-0002-4085-2887
Cand. Sci. (Chem.), researcher, Department of biophysics and environmental research
Russian Federation, Koltsovo, Novosibirsk regionValentina S. Nesmeyanova
State Research Center of Virology and Biotechnology «Vector»
Email: nesmeyanova_vs@vector.nsc.ru
ORCID iD: 0000-0003-1091-3586
junior researcher, Department of biophysics and environmental research
Russian Federation, Koltsovo, Novosibirsk regionDaniil V. Shanshin
State Research Center of Virology and Biotechnology «Vector»
Email: shanshin_dv@vector.nsc.ru
ORCID iD: 0000-0001-9985-1582
Cand. Sci. (Biol.), junior researcher, Department of biophysics and environmental research
Russian Federation, Koltsovo, Novosibirsk regionBoris N. Zaitsev
State Research Center of Virology and Biotechnology «Vector»
Email: zaitsev@vector.nsc.ru
ORCID iD: 0000-0001-6359-465X
Cand. Sci. (Phys.-Math.), senior researcher, Department of microscopic research
Russian Federation, Koltsovo, Novosibirsk regionDenis N. Kisakov
State Research Center of Virology and Biotechnology «Vector»
Email: kisakov_dn@vector.nsc.ru
ORCID iD: 0000-0003-4889-6865
junior researcher, Department of bioengineering
Russian Federation, Koltsovo, Novosibirsk regionLyubov A. Kisakova
State Research Center of Virology and Biotechnology «Vector»
Email: orlova_la@vector.nsc.ru
ORCID iD: 0000-0001-7214-1855
junior researcher, Department of bioengineering
Russian Federation, Koltsovo, Novosibirsk regionDmitry N. Shcherbakov
State Research Center of Virology and Biotechnology «Vector»
Email: scherbakov_dn@vector.nsc.ru
ORCID iD: 0000-0001-8023-4453
Cand. Sci. (Biol.), leading researcher, Department of biophysics and environmental research
Russian Federation, Koltsovo, Novosibirsk regionLarisa I. Karpenko
State Research Center of Virology and Biotechnology «Vector»
Email: lkarpenko1@ya.ru
ORCID iD: 0000-0003-4365-8809
Dr. Sci. (Biol.), leading researcher, Head, Laboratory of recombinant vaccines, Department of bioengineering
Russian Federation, Koltsovo, Novosibirsk regionAlexander A. Ilyichev
State Research Center of Virology and Biotechnology «Vector»
Email: ilyichev@vector.nsc.ru
ORCID iD: 0000-0001-5356-0843
Dr. Sci. (Biol.), chief researcher, Department of bioengineering
Russian Federation, Koltsovo, Novosibirsk regionReferences
- Pawlowski C., Lenehan P., Puranik A., et al. FDA-authorized mRNA COVID-19 vaccines are effective per real-world evidence synthesized across a multi-state health system. Med. 2021;2(8):979–92.e8. DOI: https://doi.org/10.1016/j.medj.2021.06.007
- Khobragade A., Bhate S., Ramaiah V., et al. Efficacy, safety, and immunogenicity of the DNA SARS-CoV-2 vaccine (ZyCoV-D): the interim efficacy results of a phase 3, randomised, double-blind, placebo-controlled study in India. Lancet. 2022;399(10332):1313–21. DOI: https://doi.org/10.1016/S0140-6736(22)00151-9
- Logunov D.Y., Dolzhikova I.V., Shcheblyakov D.V., et al. Safety and efficacy of an rAd26 and rAd5 vector-based heterologous prime-boost COVID-19 vaccine: an interim analysis of a randomised controlled phase 3 trial in Russia. Lancet. 2021;397(10275):671–81. DOI: https://doi.org/10.1016/s0140-6736(21)00234-8
- Ramasamy M.N., Minassian A.M., Ewer K.J., et al. Safety and immunogenicity of ChAdOx1 nCoV-19 vaccine administered in a prime-boost regimen in young and old adults (COV002): a single-blind, randomised, controlled, phase 2/3 trial. Lancet. 2021;396(10267):1979–93. DOI: https://doi.org/10.1016/S0140-6736(20)32466-1
- Ng O.W., Chia A., Tan A.T., et al. Memory T cell responses targeting the SARS coronavirus persist up to 11 years post-infection. Vaccine. 2016;34(17):2008–14. DOI: https://doi.org/10.1016/j.vaccine.2016.02.063
- Alhabbab R.Y., Algaissi A., Mahmoud A.B., et al. Middle East respiratory syndrome coronavirus infection elicits long-lasting specific antibody, T and B cell immune responses in recovered individuals. Clin. Infect. Dis. 2023;76(3):e308–18. DOI: https://doi.org/10.1093/cid/ciac456
- Zhang Y., Ndzouboukou J.B., Lin X., et al. SARS-CoV-2 evolves to reduce but not abolish neutralizing action. J. Med. Virol. 2023;95(1):e28207. DOI: https://doi.org/10.1002/jmv.28207
- Le Bert N., Tan A.T., Kunasegaran K., et al. SARS-CoV-2-specific T cell immunity in cases of COVID-19 and SARS, and uninfected controls. Nature. 2020;584(7821):457–62. DOI: https://doi.org/10.1038/s41586-020-2550-z
- Arya R., Tripathi P., Nayak K., et al. Insights into the evolution of mutations in SARS-CoV-2 non-spike proteins. Microb. Pathog. 2023;185:106460. DOI: https://doi.org/10.1016/j.micpath.2023.106460
- Srinivasan S., Cui H., Gao Z., et al. Structural genomics of SARS-CoV-2 indicates evolutionary conserved functional regions of viral proteins. Viruses. 2020;12(4):360. DOI: https://doi.org/10.3390/v12040360
- Choi S.J., Kim D.U., Noh J.Y., et al. T cell epitopes in SARS-CoV-2 proteins are substantially conserved in the Omicron variant. Cell. Mol. Immunol. 2022;19(3):447–8. DOI: https://doi.org/10.1038/s41423-022-00838-5
- Cromer D., Steain M., Reynaldi A., et al. Neutralising antibody titres as predictors of protection against SARS-CoV-2 variants and the impact of boosting: a meta-analysis. Lancet Microbe. 2022;3(1):e52–61. DOI: https://doi.org/10.1016/S2666-5247(21)00267-6
- Chavda V.P., Ghali E.N.H.K., Balar P.C., et al. Protein subunit vaccines: Promising frontiers against COVID-19. J. Control. Release. 2024;366:761–82. DOI: https://doi.org/10.1016/j.jconrel.2024.01.017
- Mekonnen D., Mengist H.M., Jin T. SARS-CoV-2 subunit vaccine adjuvants and their signaling pathways. Expert. Rev. Vaccines. 2022;21(1):69-81. DOI: https://doi.org/10.1080/14760584.2021.1991794
- Lin H., Han R., Wu W. Glucans and applications in drug delivery. Carbohydr. Polym. 2024;332:121904. DOI: https://doi.org/10.1016/j.carbpol.2024.121904
- Huang S., Huang G. The dextrans as vehicles for gene and drug delivery. Future Med. Chem. 2019;11(13):1659–67. DOI: https://doi.org/10.4155/fmc-2018-0586
- An Y., Zhao G., Duan H., et al. Robust and protective immune responses induced by heterologous prime-boost vaccination with DNA-protein dimeric RBD vaccines for COVID-19. J. Med. Virol. 2023;95(7):e28948. DOI: https://doi.org/10.1002/jmv.28948
- Jalah R., Kulkarni V., Patel V., et al. DNA and protein co-immunization improves the magnitude and longevity of humoral immune responses in macaques. PLoS One. 2014;9(3):e91550. DOI: https://doi.org/10.1371/journal.pone.0091550
- Felber B.K., Lu Z., Hu X., et al. Co-immunization of DNA and protein in the same anatomical sites induces superior protective immune responses against SHIV challenge. Cell Rep. 2020;31(6):107624. DOI: https://doi.org/10.1016/j.celrep.2020.107624
- Li Y., Bi Y., Xiao H., et al. A novel DNA and protein combination COVID-19 vaccine formulation provides full protection against SARS-CoV-2 in rhesus macaques. Emerg. Microbes Infect. 2021;10(1):342–55. DOI: https://doi.org/10.1080/22221751.2021.1887767
- Liao H.C., Huang M.S., Chiu F.F., et al. Co-delivery of a trimeric spike DNA and protein vaccine with aluminum hydroxide enhanced Th1-dominant humoral and cellular immunity against SARS-CoV-2. J. Med. Virol. 2023;95(8):e29040. DOI: https://doi.org/10.1002/jmv.29040
- He Q., Mao Q., An C., et al. Heterologous prime-boost: breaking the protective immune response bottleneck of COVID-19 vaccine candidates. Emerg. Microbes Infect. 2021;10(1):629–37. DOI: https://doi.org/10.1080/22221751.2021.1902245
- Peng D., Zhao T., Hong W., et al. Heterologous vaccination with subunit protein vaccine induces a superior neutralizing capacity against BA.4/5-included SARS-CoV-2 variants than homologous vaccination of mRNA vaccine. MedComm (2020). 2023;4(2):e238. DOI: https://doi.org/10.1002/mco2.238
- Kim E., Khan M.S., Ferrari A., et al. SARS-CoV-2 S1 subunit booster vaccination elicits robust humoral immune responses in aged mice. Microbiol. Spectr. 2023;11(3):e0436322. DOI: https://doi.org/10.1128/spectrum.04363-22
- Park H.J., Bang Y.J., Kwon S.P., et al. Analyzing immune responses to varied mRNA and protein vaccine sequences. NPJ Vaccines. 2023;8(1):84. DOI: https://doi.org/10.1038/s41541-023-00684-0
- Borgoyakova M.B., Karpenko L.I., Rudometov A.P., et al. Artificial COVID-19 T-cell immunogen. Bull. Exp. Biol. Med. 2023;175(6):804–9. DOI: https://doi.org/10.1007/s10517-023-05951-7
- Borgoyakova M.B., Karpenko L.I., Rudometov A.P., et al. Self-assembled particles combining SARS-CoV-2 RBD protein and RBD DNA vaccine induce synergistic enhancement of the humoral response in mice. Int. J. Mol. Sci. 2022;23(4):2188. DOI: https://doi.org/10.3390/ijms23042188
- Singh A. Eliciting B cell immunity against infectious diseases using nanovaccines. Nat. Nanotechnol. 2021;16(1):16–24. DOI: https://doi.org/10.1038/s41565-020-00790-3
- Toor S.M., Saleh R., Sasidharan Nair V., et al. T-cell responses and therapies against SARS-CoV-2 infection. Immunology. 2021;162(1):30–43. DOI: https://doi.org/10.1111/imm.13262
- Safavi A., Kefayat A., Mahdevar E., et al. Exploring the out of sight antigens of SARS-CoV-2 to design a candidate multi-epitope vaccine by utilizing immunoinformatics approaches. Vaccine. 2020;38(48):7612–28. DOI: https://doi.org/10.1016/j.vaccine.2020.10.016
- Mahajan S., Kode V., Bhojak K., et al. Immunodominant T-cell epitopes from the SARS-CoV-2 spike antigen reveal robust pre-existing T-cell immunity in unexposed individuals. Sci. Rep. 2021;11(1):13164. DOI: https://doi.org/10.1038/s41598-021-92521-4
- Bhattacharya M., Sharma A.R., Ghosh P., et al. A next-generation vaccine candidate using alternative epitopes to protect against wuhan and all significant mutant variants of SARS-CoV-2: An immunoinformatics approach. Aging Dis. 2021;12(8):2173–95. DOI: https://doi.org/10.14336/AD.2021.0518
- de Oliveira L.M., Morale M.G., Chaves A.A., et al. Expression, polyubiquitination, and therapeutic potential of recombinant E6E7 from HPV16 antigens fused to ubiquitin. Mol. Biotechnol. 2017;59(1):46–56. DOI: https://doi.org/10.1007/s12033-016-9990-6
- Bazhan S.I., Karpenko L.I., Lebedev L.R., et al. A synergistic effect of a combined bivalent DNA-protein anti-HIV-1 vaccine containing multiple T- and B-cell epitopes of HIV-1 proteins. Mol. Immunol. 2008;45(3):661–9. DOI: https://doi.org/10.1016/j.molimm.2007.07.016
- Karpenko L.I., Apartsin E.K., Dudko S.G., et al. Cationic polymers for the delivery of the Ebola DNA vaccine encoding artificial T-cell immunogen. Vaccines (Basel). 2020;8(4):718. DOI: https://doi.org/10.3390/vaccines8040718
- Volosnikova E.A., Merkuleva I.A., Esina T.I., et al. SARS-CoV-2 RBD conjugated to polyglucin, spermidine, and dsRNA elicits a strong immune response in mice. Vaccines (Basel). 2023;11(4):808. DOI: https://doi.org/10.3390/vaccines11040808
Supplementary files