Genetic characterization and phylogenetic analysis of human norovirus infection in individual municipalities of the Sverdlovsk region in 2022
- Authors: Bykov R.O.1, Scriabina S.V.2, Kilyachina A.S.3, Itani T.M.1, Chalapa V.I.1, Starikova P.K.1, Koltunov S.S.3, Ponomareva A.V.2, Semenov A.V.1,4
-
Affiliations:
- Federal Scientific Research Institute of Viral Infections «Viromе»
- Federal Service for Surveillance on Consumer Rights Protection and Human Wellbeing in the Sverdlovsk Region
- Center for Hygiene and Epidemiology in the Sverdlovsk Region
- Ural Federal University named after the First President of Russia B.N. Yeltsin
- Issue: Vol 100, No 4 (2023)
- Pages: 306-313
- Section: ORIGINAL RESEARCHES
- URL: https://microbiol.crie.ru/jour/article/view/9992
- DOI: https://doi.org/10.36233/0372-9311-402
- EDN: https://elibrary.ru/qiehre
- ID: 9992
Cite item
Full Text
Abstract
Introduction. Human Noroviruses (HuNoV) are highly contagious pathogens responsible of acute human norovirus infection (HuNoVI). GII.4 is the prevailing norovirus genotype in the incidence of gastroenteritis worldwide. Currently there are no studies on the molecular monitoring and phylogenetic analysis of HuNoV in the territory of the Sverdlovsk region, therefore it is not possible to objectively assess their genetic diversity.
The aim of the study is to carry out genotyping and phylogenetic analysis of HNoVI in municipalities of the Sverdlovsk region: Ekaterinburg, Nizhny Tagil, Kamensk-Uralsky in 2022.
Materials and methods. Fecal samples were collected from children suffering from HuNoVI in the municipalities of the Sverdlovsk region. Viral RNA was extracted from stool samples followed by reverse transcription polymerase chain reaction with degenerate primers flanking the ORF1/ORF2 junction. The amplified samples were genotyped by Sanger sequencing. Multiple sequence alignments were carried out and phylogenetic trees were constructed.
Results. The total number of studied samples was 220. According to sequencing results, 73 positive sequences of HuNoV were typed, which represents 33% of the total volume of the studied samples. The largest share of genotypes is occupied by noroviruses GII (58%), noroviruses GI (42%). Noroviruses GII.17, GI.3 were the dominant genotypes. Phylogenetic analysis demonstrates that the identified sequences on the territory of the Sverdlovsk region have the smallest genetic distance, which gives grounds for their unification into a common cluster.
Conclusion. For the first time, a genetic analysis of HuNoV was carried out in the territory of the Sverdlovsk region. The norovirus genotyping system based on the amplification of the ORF1/ORF2 region makes it possible to successfully identify various genotypes of noroviruses from the Sverdlovsk region. It has been confirmed that GII noroviruses remain the dominant genetic group. The results of phylogenetic analysis demonstrate the greatest homology in the formed clusters between isolates from Ekaterinburg, Nizhny Tagil, Kamensk-Uralsky.
Keywords
Full Text
Введение
В глобальном масштабе норовирусы являются причиной каждого пятого случая острого небактериального гастроэнтерита и ежегодно вызывают порядка 699 млн случаев заболевания и более 200 тыс. случаев смерти [1, 2]. Норовирусы реализуют своё заразное начало с помощью фекально- орального механизма передачи. Для инфицирования реактивного организма человека требуется около 18 вирусных частиц [2]. Норовирусная инфекция (НВИ) — убиквитарная, в эпидемический процесс вовлекаются все возрастные группы населения, бóльшая часть заболеваемости и смертности приходится на детей и людей пожилого возраста [3, 4].
Норовирусы — безоболочечные РНК-содержащие вирусы, относящиеся к роду Norwalk, семейству Caliciviridae. Норовирус входит в генетически разнообразную группу вирусов, поражающих широкий спектр видов млекопитающих-хозяев. Геном норовируса представлен тремя открытыми рамками считывания (ORF), которые контролируют синтез вирусных белков (ORF1 — РНК-зависимая РНК-полимераза, ORF2 — мажорный капсидный белок VP1, ORF3 — минорный капсидный белок VP2) [5, 6]. Белок VP1 отвечает за связывание вирусов с клеточными рецепторами — полисахаридными антигенами групп крови человека. Белок VP2 стабилизирует вирусную частицу и участвует в сборке вирионов [7]. В последние годы было зарегистрировано несколько новых генотипов норовирусов, что потребовало обновления существующей схемы классификации [8].
Классификация генотипов норовируса основана на определении типовой принадлежности двух участков РНК-генома: участка, кодирующего основной капсидный белок, — VP1-типирование; участка, кодирующего РНК-зависимую РНК-полимеразу, — P-типирование. В результате типирования дифференцируют Р-группы и P-типы. Современная классификация, основанная на типировании участка VP1, включает 10 сформированных геногрупп и до 49 генотипов [8]. Формированию новых геновариантов внутри геногрупп способствуют гомологичные, межгенные, субгенные рекомбинации. Выделяют несколько основных «горячих точек мутационных замен» в геноме, где чаще всего образуются рекомбинанты: регион ORF1/ORF2 и ORF2, кодирующие структурные и неструктурные белки; область ORF2 — главный капсидный белок VP1 (гипервариабельные домены и субдомены главного капсидного белка S, P1, P2) [9–12].
Вторая генетическая группа GII вызывает более 50% случаев вспышечной заболеваемости по всему миру. Превалирующим генотипом в генотипической структуре НВИ человека является GII.4 [13, 14]. На фоне ограничительных мероприятий в отношении коронавирусной инфекции в 2020 г. произошло снижение заболеваемости НВИ в 2 раза. Однако в 2021 г. показатель заболеваемости НВИ в России увеличился на 40%1. Наибольший удельный вес в генотипической структуре возбудителей НВИ на территории России занимает GII.4.
Из-за высокой частоты рекомбинационной изменчивости каждые 2–3 года новые варианты GII.4 последовательно заменяют друг друга, быстро распространяясь среди населения всего мира [3]. На территории Свердловской области ранее не проводился молекулярно-генетический мониторинг циркуляции возбудителей НВИ вместе с филогенетическим анализом. Использование унифицированной системы генотипирования норовирусов и последующий филогенетический анализ позволят в полной мере оценить генетическое разнообразие норовирусов, а также отслеживать события эволюционной дивергенции внутри вирусной популяции.
Цель исследования — генотипирование и филогенетический анализ возбудителей НВИ человека на территории Екатеринбурга, Нижнего Тагила, Каменск-Уральского за 2022 г.
Материалы и методы
С февраля 2022 г. по декабрь 2022 г. проводился сбор образцов фекального материала от больных детей с НВИ на территории Свердловской области в муниципалитетах с повышенной заболеваемостью: Екатеринбург, Нижний Тагил, Каменск-Уральский. НВИ в биологических образцах была лабораторно подтверждена методами иммуноферментного анализа (ИФА) и/или полимеразной цепной реакции (ПЦР) в реальном времени. Общее количество исследованных образцов составляет 220.
Исследование проводилось при добровольном информированном согласии законных представителей пациентов. Протокол исследования одобрен Этическим комитетом ФБУН ФНИИВИ «Виром» Роспотребнадзора (протокол № 1 от 17.03.2023).
Выделение РНК, ПЦР и секвенирование
Из нативных образцов фекалий готовилась 10% суспензия в физиологическом растворе, из которой выделялись нуклеиновые кислоты методом переосаждения с использованием набора реагентов «РИБО-преп» (ЦНИИ Эпидемиологии). Затем производилась обратная транскрипция для получения кДНК на матрице РНК при помощи комплекта реагентов «РЕВЕРТА-L» (ЦНИИ Эпидемиологии). Для амплификации возбудителя НВИ был выбран специфический участок генома, соответствующий региону ORF1/ORF2 (участки генома, контролирующие экспрессию РНК-зависимой РНК-полимеразы и главного капсидного белка VP1). Для постановки ПЦР был использован протокол с вырожденными праймерами, фланкирующими регион ORF1/ORF2 [6]. Для приготовления реакционной смеси использовали «5х ScreenMix» («Евроген») и последовательности вырожденных праймеров для амплификации участка генома первой и второй геногрупп норовируса: G1SKF, G1SKR, G2SKF, G2SKR. Температурный профиль протокола амплификации проходил при 94оС в течение 3 мин, 40 циклов при 94оС, 50оС и 72оС в течение 2 мин, 72оС в течение 7 мин. Экстракция кДНК из геля проводилась при помощи набора реагентов «PureLink™ Quick Gel Extraction and PCR Purification Combo Kit» («Invitrogen», «Lifetechnologies»). Последующее определение нуклеотидных последовательностей генома норовирусов производили методом секвенирования по Сэнгеру при помощи набора реагентов «BigDye Terminator v3.1 Cycle Sequencing Kit» («Applied Biosystems»).
Филогенетический анализ
Идентификацию полученной генетической последовательности проводили при помощи сервиса BLAST. Для создания консенсусной последовательности выбирали референсные последовательности норовируса, представленные в GenBank, демонстрирующие наибольшую гомологию с типированным образцом. Анализ и применение алгоритма множественного выравнивания (ClustalW) типированных генетических последовательностей норовируса человека проводили в программе «MEGA v. 11». Для создания филогенетических деревьев и определения попарных расстояний между таксонами в двух геногруппах норовирусов использовали метод ближайших соседей вместе с параметрической моделью Kimura-2. Достоверность топологии аддитивных филограмм оценивали методом повторных выборок на основании анализа 1000 псевдорепликаций. На филограммах указываются статистические индексы поддержки более 60. Генетическое разнообразие полученных кластеров анализировали в программе «MEGA v. 11». Консенсусные последовательности были депонированы в GenBank: ON681575.1–ON681586.1, OP862432.1, OP862428.1–OP862431.1, OP862363.1–OP862368.1.
Результаты
Распределение и распространение генотипов норовируса
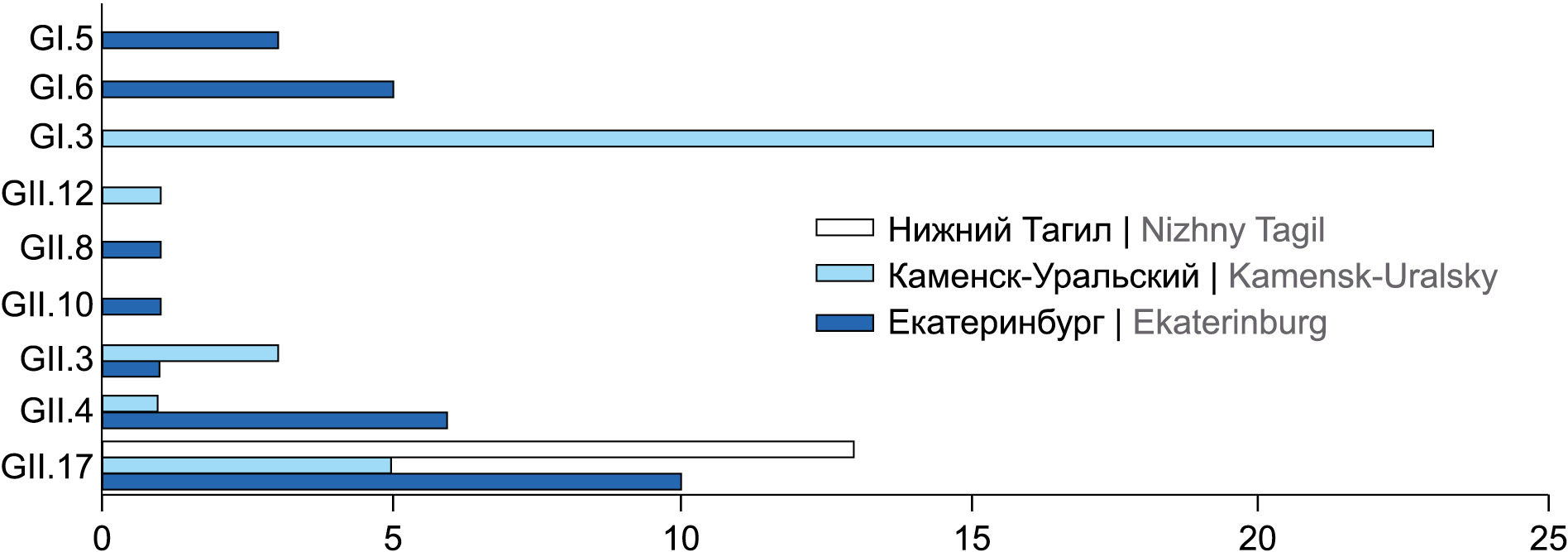
Всего за анализируемый период было исследовано 220 образцов клинического материала. По результатам секвенирования удалось типировать 73 возбудителя НВИ, что составляет 33% общего объёма исследованных образцов. В результате генотипирования по Сэнгеру установлено, что наибольший удельный вес в генотипическом профиле норовирусов приходится на вторую геногруппу GII (n = 42 (58%)), норовирусы GI (n = 31 (42%)). Выявлены различные генотипы возбудителей НВИ на территории отдельных муниципалитетов (рис. 1). Генотипическое распределение полученных последовательностей имеет следующий вид: GII.17 (n = 28; 40%); GI.3 (n = 23; 33%); GII.4 (n = 7; 10%,); GI.6 (n = 5; 7%); GII.3 (n = 4; 6%); G1.7 (n = 3; 4%). Норовирусы, относящиеся к генотипам GII.17, GI.3, являются доминирующими вирусами, выявленными на территории отдельных муниципалитетов в Свердловской области за 2022 г. (рис. 2).
Рис. 1. Распределение выявленных генотипов в отдельных муниципалитетах Свердловской области за 2022 г.
Fig. 1. Distribution of identified genotypes in selected municipalities of the Sverdlovsk region for 2022.
Рис. 2. Удельный вес возбудителей НВИ человека в Свердловской области за 2022 г.
Fig. 2. Distribution of different norovirus genogroups detected in the Sverdlovsk region in 2022.
Филогенетический анализ норовирусов GI и GII
Последовательности GI.5 из Екатеринбурга группируются в общий кластер. Изоляты из данного кластера генетически неоднородны. Последовательность GI.5_197 имеет меньшую гомологию и наибольшую генетическую дистанцию между другими генотипами, входящими в кластер. Последовательности GI.5 из Екатеринбурга образуют парафилетическую группу с близкородственными генотипами из Таиланда и Южной Африки.
Последовательности GI.6 из Екатеринбурга входят в общий кластер. Последовательности из данного кластера образуют парафилетическую группу с близкородственными генотипами из Японии.
При построении филогенетического дерева консенсусные последовательности GI.3 из Каменск-Уральского образуют общий кластер. Последовательности из кластера GI.3 образуют парафилетическую группу с близкородственными генотипами из Бразилии, Японии и США.
Последовательности GII.17 из Нижнего Тагила, Екатеринбурга, Каменск-Уральского группируются в единый кластер. Последовательности из данного кластера образуют парафилетическую группу с близкородственными генотипами из Китая (рис. 3).
Рис. 3. Филогенетическое древо, основанное на фрагменте главного капсидного белка, построенное на основе нуклеотидных последовательностей норовирусных генотипов GI.5, GI.6, GI.3, GII.17.
Fig. 3. Phylogenetic tree based on the HuNoV viral protein 1 major capsid gene fragment, constructed based on nucleotide sequences of HuNoV genotypes GI.5, GI.6, GI.3, GII.17.
Обсуждение
Из 220 исследованных образцов 197 были положительны на антиген норовируса в ИФА, 23 образца — в ПЦР. Полученный результат согласуется с данными предшествующего исследования, в котором была продемонстрирована низкая специфичность ИФА на антиген норовируса. Низкая специфичность может приводить к ложноположительному результату при определении генетической группы у норовирусов. Низкий процент типированных норовирусов может быть обусловлен высоким процентом ложноположительных результатов в биологических образцах, обнаруженных методом ИФА [15]. Из-за высокой частоты мутационных событий вторая генетическая группа занимает доминирующее положение в популяции норовирусов. В генотипической структуре возбудителей НВИ GII.4 является доминирующим генотипом с середины 1990-х гг. в США, Европе и Океании, вызывая до 70–80% вспышек норовирусных гастроэнтеритов [16]. Молекулярно-генетический мониторинг возбудителей НВИ, а также обнаружение эволюционных событий на уровне вирусной популяции позволили подтвердить основную парадигму, в которой наиболее часто регистрируемой геногруппой является GII. Наибольший удельный вес в генотипической структуре норовирусов занимает группа GII (58%), норовирусы группы GI — 42%. Так, впервые за 2022 г. в Екатеринбурге, Нижнем Тагиле, Каменск-Уральском генотипический профиль и наибольший удельный вес представлены норовирусными генотипами GII.17 и GI.3. Наблюдается рост заболеваемости в отдельных регионах России рекомбинантным GII.3. Дальнейший мониторинг циркуляции норовирусов позволит отследить возможный рост рекомбинантного GII.3 на территории отдельных муниципалитетов в Свердловской области [18].
Идентифицированы редкие норовирусные генотипы, относящиеся к геногруппе GI (GI.3, GI.5, GI.6, GI.7), которые были депонированы в GenBank. Филогенетический анализ продемонстрировал, что консенсусные последовательности норовирусов GI и GII из Екатеринбурга часто образуют парафилетические группы с изолятами из Японии, Китая, Таиланда, США, что может свидетельствовать о наименьшей генетической дистанции между ними [9, 14, 16, 19–21]. Множество референсных изолятов норовирусов не кластеризуются и не образуют парафилетические группы с последовательностями из Свердловской области, что, возможно, указывает на дивергентную эволюцию идентифицированных генотипов [22]. Организация молекулярно-генетического мониторинга и проведение филогенетического анализа предоставляют возможность объективно определять генетическую дистанцию между норовирусами и циркуляцию различных геновариантов на эндемичных территориях, что способствует повышению эффективности работы основных подсистем эпидемиологического надзора.
Заключение
Впервые на территории отдельных муниципалитетов Свердловской области проведён генетический анализ возбудителей НВИ. Cистема генотипирования норовирусов, основанная на амплификации региона ORF1/ORF2, позволяет успешно идентифицировать различные генотипы норовирусов. Генотипический профиль представлен норовирусами, которые относятся к генотипам GII.17 и GI.3. Консенсусные последовательности выявленных норовирусов демонстрируют наименьшую генетическую дистанцию с изолятами из Китая, Японии, США, Вьетнама, Таиланда, Бразилии, Новой Зеландии, что говорит об убиквитарности идентифицированных генотипов. Отсутствие данных о структуре генотипического профиля возбудителей НВИ на территории Екатеринбурга, Нижнего Тагила, Каменск-Уральского в прошлом позволяет предположить, что выявленные норовирусы могли циркулировать ранее. Дальнейшее применение представленной структуры генотипирования позволит с большей доступностью определять генетическое разнообразие возбудителей НВИ в Свердловской области. Организация надзорной деятельности за циркуляцией норовирусной популяции, секвенирование и филогенетический анализ последовательностей генома предоставят возможность анализировать возникшие гипотезы об общности источника возбудителя инфекции, а также определения возможных факторов передачи в очагах заболеваемости острым норовирусным гастроэнтеритом.
Этическое утверждение. Исследование проводилось при добровольном информированном согласии законных представителей пациентов. Протокол исследования одобрен Этическим комитетом ФНИИВИ «Виром» (протокол № 1 от 17.03.2023).
Источник финансирования. Исследование выполнено в рамках государственного задания ФНИИВИ «Виром» Роспотребнадзора (рег. № НИР 123051100045-0).
Конфликт интересов. Авторы декларируют отсутствие явных и потенциальных конфликтов интересов, связанных с публикацией настоящей статьи.
Участие авторов. Все авторы внесли существенный вклад в проведение поисково-аналитической работы и подготовку статьи, прочли и одобрили финальную версию до публикации.
Ethics approval. The study was conducted with the voluntary informed consent of the legal representatives of the patients. The research protocol was approved by the Ethics Committee of the Federal Scientific Research Institute of Viral Infections «Virom» (protocol No. 1, March 17, 2023).
Funding source. The study was carried out within the framework of the state task of the FNIIVI "Virom" of Rospotrebnadzor (registration No. NIR 123051100045-0).
Conflict of interest. The authors declare no apparent or potential conflicts of interest related to the publication of this article.
Author contribution. Аll authors made a substantial contribution to the conception of the work, acquisition, analysis, interpretation of data for the work, drafting and revising the work, final approval of the version to be published.
1 Государственный доклад «О состоянии санитарно- эпидемиологического благополучия населения в Российской Федерации в 2021 году». URL: https://www.rospotrebnadzor.ru/upload/iblock/594/sqywwl4tg5arqff6xvl5dss0l7vvuank/Gosudarstvennyy-doklad.-O-sostoyanii-sanitarno_epidemiologicheskogo-blagopoluchiya-naseleniya-v-Rossiyskoy-Federatsii-v-2021-godu.pdf
About the authors
Roman O. Bykov
Federal Scientific Research Institute of Viral Infections «Viromе»
Author for correspondence.
Email: bykov_ro@eniivi.ru
ORCID iD: 0000-0003-3868-3225
ResearcherId: ИКР-5821-2023
Postgraduate Student, Researcher, Laboratory of Enteral Viral Infections
Russian Federation, Ekaterinburg
Svetlana V. Scriabina
Federal Service for Surveillance on Consumer Rights Protection and Human Wellbeing in the Sverdlovsk Region
Email: scriabina_sv@66.rospotrebnadzor.ru
ORCID iD: 0000-0001-7182-6262
Head, Epidemiological Surveillance Department
Russian Federation, EkaterinburgAnastasia S. Kilyachina
Center for Hygiene and Epidemiology in the Sverdlovsk Region
Email: kilyachina_sv@66.rospotrebnadzor.ru
ORCID iD: 0000-0003-1751-3462
Head, Laboratory of Biological Factors Control, Virologist
Russian Federation, EkaterinburgTarek M. Itani
Federal Scientific Research Institute of Viral Infections «Viromе»
Email: itani_tm@eniivi.ru
ORCID iD: 0000-0002-2113-6543
Cand. Sci. (Biol.), Head, Laboratory of Enteral Viral Infections
Russian Federation, EkaterinburgVladislav I. Chalapa
Federal Scientific Research Institute of Viral Infections «Viromе»
Email: chalapa_vi@eniivi.ru
ORCID iD: 0000-0001-5823-5257
Researcher, Laboratory of Enteral Viral Infections
Russian Federation, EkaterinburgPolina K. Starikova
Federal Scientific Research Institute of Viral Infections «Viromе»
Email: starikova_pk@eniivi.ru
ORCID iD: 0000-0002-7664-3059
Epidemiologist, Laboratory of Enteral Viral Infections
Russian Federation, EkaterinburgStanislav S. Koltunov
Center for Hygiene and Epidemiology in the Sverdlovsk Region
Email: koltunov_sv@66.rospotrebnadzor.ru
ORCID iD: 0009-0008-0881-7644
Head, Department of Epidemiological Examinations, Epidemiologist
Russian Federation, EkaterinburgAngelika V. Ponomareva
Federal Service for Surveillance on Consumer Rights Protection and Human Wellbeing in the Sverdlovsk Region
Email: ponomareva_sv@66.rospotrebnadzor.ru
ORCID iD: 0000-0002-8237-8335
Deputy Head
Russian Federation, EkaterinburgAlexander V. Semenov
Federal Scientific Research Institute of Viral Infections «Viromе»; Ural Federal University named after the First President of Russia B.N. Yeltsin
Email: semenov_av@eniivi.ru
ORCID iD: 0000-0003-3223-8219
D. Sci. (Biol.), Director, Federal Scientific Research Institute of Viral Infections «Viromе»; Professor, Department of Experimental Biology and Biotechnology
Russian Federation, Ekaterinburg; EkaterinburgReferences
- Епифанова Н.В., Луковникова Л.Б., Новикова Н.А. и др. Эпидемические варианты норовирусов генотипа GII.4 в Нижнем Новгороде в 2006–2012 гг. Журнал микробиологии, эпидемиологии и иммунобиологии. 2014;(2):64–72. Epifanova N.V., Lukovnikova L.B., Novikova N.A., et al. Epidemic variants of norovirus genotype GII.4 in Nizhny Novgorod in 2006–2012. Journal of Microbiology Epidemiology and Immunobiology. 2014;(2):64–72. EDN: https://elibrary.ru/ttzjwp
- Patel M.M., Widdowson M.A., Glass R.I., et al. Systematic literature review of role of noroviruses in sporadic gastroenteritis. Emerg. Infect. Dis. 2008;14(8):1224–31. DOI: https://doi.org/10.3201/eid1408.071114
- Lopman B.A., Steele D., Kirkwood C.D., Parashar U.D. The vast and varied global burden of norovirus: prospects for prevention and control. PLoS Med. 2016;13(4):e1001999. DOI: https://doi.org/10.1371/journal.pmed.1001999
- Bartsch S.M., Lopman B.A., Ozawa S., et al. Global economic burden of norovirus gastroenteritis. PLoS One. 2016;11(4):e0151219. DOI: https://doi.org/10.1371/journal.pone.0151219
- Prasad B.V., Shanker S., Muhaxhiri Z., et al. Chapter 3.1 – Structural biology of noroviruses. In: Viral Gastroenteritis. Molecular Epidemiology and Pathogenesis. Elsevier;2016:329–54. DOI: https://doi.org/10.1016/B978-0-12-802241-2.00016-X
- Katayama H., Shirato-Horikoshi H., Kojima S., et al. Phylogenetic analysis of the complete genome of 18 Norwalk-like viruses. Virology. 2002;299(2):225–39. DOI: https://doi.org/10.1006/viro.2002.1568
- Graziano V.R., Wei J., Wilen C.B. Norovirus attachment and entry. Viruses. 2019;11(6):495. DOI: https://doi.org/10.3390/v11060495
- Chhabra P., de Graaf M., Parra G.I., et al. Updated classification of norovirus genogroups and genotypes. J. Gen. Virol. 2019;100(10):1393–406. DOI: https://doi.org/10.1099/jgv.0.001318
- Bull R.A., Hansman G.S., Clancy L.E., et al. Norovirus recombination in ORF1/ORF2 overlap. Emerg. Infect. Dis. 2005;11(7):1079–85. DOI: https://doi.org/10.3201/eid1107.041273
- Phan T.G., Kaneshi K., Ueda Y., et al. Genetic heterogeneity, evolution, and recombination in noroviruses. J. Med. Virol. 2007;79(9):1388–400. DOI: https://doi.org/10.1002/jmv.20924
- de Graaf M., van Beek J., Koopmans M.P. Human norovirus transmission and evolution in a changing world. Nat. Rev. Microbiol. 2016;14(7):421–33. DOI: https://doi.org/10.1038/nrmicro.2016.48
- Pérez-Losada M., Arenas M., Galán J.C., et al. Recombination in viruses: Mechanisms, methods of study, and evolutionary consequences. Infect. Genet. Evol. 2015;30:296–307. DOI: https://doi.org/10.1016/j.meegid.2014.12.022
- Chen S.Y., Chiu C.H. Worldwide molecular epidemiology of norovirus infection. Paediatr. Int. Child Health. 2012;32(3): 128–31. DOI: https://doi.org/10.1179/2046905512Y.0000000031
- Siebenga J.J., Vennema H., Zheng D.P., et al. Norovirus illness is a global problem: emergence and spread of norovirus GII.4 variants, 2001–2007. J. Infect. Dis. 2009;200(5):802–12. DOI: https://doi.org/10.1086/605127
- Schmid M., Oehme R., Schalasta G., et al. Fast detection of noroviruses using a real-time PCR assay and automated sample preparation. BMC Infect. Dis. 2004;4:15. DOI: https://doi.org/10.1186/1471-2334-4-15
- Huhti L., Szakal E.D., Puustinen L., et al. Norovirus GII-4 causes a more severe gastroenteritis than other noroviruses in young children. J. Infect. Dis. 2011;203(10):1442–4. DOI: https://doi.org/10.1093/infdis/jir039
- Epifanova N.V., Sashina T.A., Morozova O.V., et al. An increase in prevalence of recombinant GII.3[P12] norovirus in sporadic acute diarrhea in children in Nizhny Novgorod, Russia, 2018–2021. Virus Genes. 2022;58(5):467–72. DOI: https://doi.org/10.1007/s11262-022-01919-3
- Pires S.M., Fischer-Walker C.L., Lanata C.F., et al. Aetiology-specific estimates of the global and regional incidence and mortality of diarrhoeal diseases commonly transmitted through food. PLoS One. 2015;10(12):e0142927. DOI: https://doi.org/10.1371/journal.pone.0142927
- Boon D., Mahar J.E., Abente E.J., et al. Comparative evolution of GII.3 and GII.4 norovirus over a 31-year period. J. Virol. 2011;85(17):8656–66. DOI: https://doi.org/10.1128/jvi.00472-11
- Khamrin P., Kumthip K., Thongprachum A., et al. Genetic diversity of norovirus genogroup I, II, IV and sapovirus in environmental water in Thailand. J. Infect. Public Health. 2020;13(10):1481–9. DOI: https://doi.org/10.1016/j.jiph.2020.05.004
- Moyo S., Hanevik K., Blomberg B., et al. Genetic diversity of norovirus in hospitalised diarrhoeic children and asymptomatic controls in Dar es Salaam, Tanzania. Infect. Genet. Evol. 2014;26:340–7. DOI: https://doi.org/10.1016/j.meegid.2014.06.013
- Shen W., Sheng Y., Weng J., et al. Molecular epidemiology of norovirus associated with acute gastroenteritis in Taizhou, China: a retrospective study. J. Infect. Public Health. 2020;13(1):34–9. DOI: https://doi.org/10.1016/j.jiph.2019.06.006
Supplementary files