Antimicrobial resistance and virulence of carbapenem-resistant Klebsiella pneumoniae strains isolated from children in intensive care and surgical units
- Authors: Novikova I.E.1, Sadeeva Z.Z.1, Alyabyeva N.M.1, Samoylova E.A.1, Karaseva O.V.1,2, Yanyushkina O.G.2, Lazareva A.V.1
-
Affiliations:
- National Medical Research Center of Children's Health
- Clinical and Research Institute of Emergency Pediatric Surgery and Trauma
- Issue: Vol 100, No 4 (2023)
- Pages: 321-332
- Section: ORIGINAL RESEARCHES
- URL: https://microbiol.crie.ru/jour/article/view/3138
- DOI: https://doi.org/10.36233/0372-9311-373
- EDN: https://elibrary.ru/rmjxsl
- ID: 3138
Cite item
Full Text
Abstract
Introduction. Infections associated with carbapenem-resistant (Carba-R) Klebsiella pneumoniae (KP) are a serious public health problem because they lead to an increase in hospital stays, treatment costs, and an increase in patient morbidity and mortality.
Aim — to characterize antimicrobial resistance, virulence factors and genotypes of Carba-R KP strains isolated from patients in intensive care and surgical units.
Materials and methods. In total, 455 KP strains that were resistant to meropenem and/or imipenem, or susceptible to meropenem and/or imipenem at increased exposure were collected in 2018–2020. For further analysis, total 90 KP strains isolated from clinically significant sites — blood/сerebrospinal fluid/urine/lower respiratory tract/wounds/abdominal cavity of patients in intensive care and surgical units were selected. Antibiotic susceptibility was determined using the broth microdilution method. Carbapenemase genes were detected by real-time PCR. The virulence genes and K1/K2 capsular serotypes were determined by multiplex PCR. Sequence types (ST) were determined using multilocus sequence typing.
Results. Most of the selected isolates (97%) were recognized as extensively drug-resistant pathogens, three isolates were classified as multidrug-resistant pathogens. The major determinant of carbapenem resistance was blaOXA-48-like (53%), blaNDM-group was detected in 13% strains, a combination of blaNDM-group and blaOXA-48-like genes was found in 31% isolates. Two isolates harbored blaKPC-group. Most of the isolates had the virulence genes entB (100%), mrkD (99%), and ybtS (78%). The iutA gene was found in 16% strains. Four isolates had kfu gene and four strains carried rmpA gene. The K2 capsule type was determined in 13% isolates. Four ST dominated in the studied population: ST395 (26%), ST2975 (19%), ST198 (12%) and ST307 (11%).
Conclusion. Carba-R KP isolates had a high level of resistance not only to carbapenems, but also to antibiotics of other classes. Carbapenem resistance was mainly associated with OXA-48 carbapenemase. The entB, mrkD and ybtS genes were the main determinants of virulence. The epidemically successful clones CG395 and CG307 were the predominant genetic lines. Resistance to antimicrobials was combined with the presence of various virulence factors. The data obtained are important for the epidemiological surveillance of the spread of KP and have important clinical implications.
Full Text
Введение
Представители порядка Enterobacterales играют важную роль в структуре нозокомиальных инфекций. Среди них особое место занимает Klebsiella pneumoniae (KP), входящая в группу ESKAPE (Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa, Enterobacter species), представители которой являются ведущей причиной внутрибольничных инфекций во всём мире [1]. KP занимает различные ниши в природе, а также колонизирует желудочно-кишечный тракт человека и животных. Зачастую KP может быть инфекционным агентом при различных тяжёлых состояниях: пневмонии, бактериемии, менингите [2].
Глобальной проблемой здравоохранения является растущая антибиотикорезистетность KP. Штаммы KP могут сочетать различные механизмы резистентности, что ведёт к появлению мультирезистентных патогенов, устойчивых практически ко всем классам антибактериальных препаратов (АМП), что значительно усложняет лечение [3]. Основными препаратами для лечения инфекций, вызванных KP, являются бета-лактамные антибиотики. Продукция ферментов бета-лактамаз ведёт к разрушению бета-лактамного кольца, обеспечивающего антимикробную активность препарата [4]. Бета-лактамазы разделяют на четыре молекулярных класса. Сериновые бета-лактамазы относятся к классам А, C, D. Металло-бета-лактамазы (МБЛ), содержащие 1 или 2 атома цинка в активном центре, относятся к классу B [4].
В 1980-х гг. появились первые сообщения о внебольничных инфекциях у здоровых людей, вызванных гипервирулентными штаммами KP, приводящими к эндофальмиту, гнойному абсцессу печени и другим заболеваниям [5]. В настоящее время вызывает беспокойство появление гипервирулентных штаммов, резистентных к бета-лактамным антибиотикам, в том числе карбапенемам [6].
Важную роль в развитии инфекционного процесса играют факторы вирулентности KP: капсула, липополисахарид, системы утилизации ионов трёхвалентного железа и адгезивные структуры [7]. Полисахаридная капсула обеспечивает защиту бактериальной клетки от воздействия антител и факторов комплемента. Гипервирулентные штаммы ассоцированы с мукоидным фенотипом за счёт гиперпродукции капсульных полисахаридов. Гены-регуляторы мукоидного фенотипа rmpA и rmpA2 ответственны за регуляцию синтеза внеклеточных полисахаридов. Наличие слизистого фенотипа также связано с геном magA, который располагается в кластере генов cps и кодирует капсульную полимеразу Wzy, ассоциированную с серотипом K1 [8]. По данным нескольких исследований, вирулентные штаммы KP, обладающие гипермукоидным фенотипом, чаще всего принадлежали к капсульным серотипам К1 и К2 [9, 10]. Во время инфекционного процесса клеткам KP, как и другим бактериальным патогенам, требуется железо.
Сидерофоры — молекулы, секретирующиеся клетками KP для получения ионов железа из организма хозяина. У KP встречаются несколько видов сидерофоров: энтеробактин, иерсиниабактин, сальмохелин и аэробактин. Наиболее часто KP продуцируют энтеробактин, он широко распространён среди как классических, так и гипервирулетных изолятов. Экспрессию энтеробактина способен нейтрализовать липокалин-2 — белок, синтезируемый нейтрофилами и другими клетками во время инфекций. Сальмохелин — это глюкозилированная форма энтеробактина, не позволяющая липокалину-2 нейтрализовать сальмохелин [7]. Йерсиниобактин, впервые обнаруженный как фактор вирулентности у бактерий рода Yersinia, чаще экспрессируется гипервирулентными клебсиеллами (90%), чем классическими (18%) [7]. Аэробактин наиболее распространён среди гипервирулентных изолятов (93–100%), у классических штаммов встречается редко [7]. Считается, что система утилизации трёхвалентного железа Kfu также ассоциируется с гипервирулентными KP. Штаммы, обладающие этой системой поглощения железа, главным образом, выделялись от пациентов с инвазивными инфекциями [7].
Адгезивные структуры — ещё один важный фактор патогенности KP. Фимбрии 1-го и 3-го типов обеспечивают прикрепление KP к клеткам человека и абиотическим поверхностям, участвуют в формировании биоплёнок. Фимбрии 1-го типа кодируются кластером генов fim, фимбрии 3-го типа кодирует кластер генов mrkABCD [11]. Метаболизм аллантоина помогает бактериям получать углерод и азот из окружающей среды. По данным исследования, проведённого в Тайване, ген, ассоцированный с усвоением аллантоина, присутствовал в 100% гипервирулентных КР [12].
Высокий уровень устойчивости к АМП в сочетании с повышенной вирулентностью КР способствует её длительной персистенции в организме пациента. При этом происходит диссеминация микроорганизма из локусов мониторинга (зев, анус) в клинически значимые стерильные локусы (раны, нижние дыхательные пути, кровоток, мочевыводящие пути и др.) Это неблагоприятно сказывается на течении основного заболевания, приводя к увеличению сроков пребывания в лечебном учреждении и повышению расходов на лечение, поэтому мониторинг факторов вирулентности и антибиотикорезистентности крайне важен.
Цель исследования — охарактеризовать антибиотикорезистентность, вирулентные свойства и генотипы карбапенемрезистентных (Карба-Р) штаммов KP, выделенных от пациентов, находившихся в реанимационных и хирургических отделениях.
Материалы и методы
Бактериальные культуры
В 2018–2020 гг. собраны 455 штаммов KP, не чувствительных к меропенему и/или имипенему (минимальная подавляющая концентрация (МПК) меропенема и имипенема более 8 и 4 мг/л соответственно) либо чувствительных к меропенему и/или имипенему при увеличенной экспозиции (МПК меропенема и имипенема 2–8 и 2–4 мг/л соответственно). Штаммы были получены от пациентов, находившихся в реанимационных и хирургических отделениях НМИЦ здоровья детей и НИИ неотложной детской хирургии и травматологии.
Для дальнейшего исследования были отобраны 90 изолятов KP, выделенных из клинически значимых локусов: крови и ликвора (n = 10), мочи (n = 41), нижних дыхательных путей (n = 22), ран (n = 10), брюшной полости (n = 7). Отобранные изоляты KP были получены от 79 пациентов через 48 ч и более после госпитализации. Штаммы от одного пациента включали в исследование, если между датами выделения прошло не менее 1 мес.
Исследование проводилось при добровольном информированном согласии пациентов и их законных представителей. Протокол исследования одобрен Локальным независимым этическим комитетом Национального медицинского исследовательского центра здоровья детей (протокол № 5 от 02.06.2022).
Посевы биологического материала производили на питательную среду Uri-select агар («BioRad») и инкубировали при 37ºС в течение 24–48 ч. Видовую идентификацию проводили на масс-спектрометре «MALDI-ToF-MS» («Bruker Daltonics»). Рекомендуемые значения Score ≥ 2,0 использовали в качестве критерия надёжной видовой идентификации.
Чувствительность к антибактериальным препаратам
Для определения чувствительности к АМП использовали метод микроразведений в бульоне Мюллера–Хинтона («bioMerieux»). Клинические категории чувствительности изолятов к АМП определяли на основании пограничных МПК в соответствии с рекомендациями EUCAST, версия 10.0 [13]. Определена чувствительность к 15 АМП: пиперациллину-тазобактаму, тикарциллину-клавуланату, цефтазидиму, цефепиму, азтреонаму, меропенему, имипенему, тобрамицину, амикацину, гентамицину, ципрофлоксацину, левофлоксацину, колистину, триметоприму-сульфаметоксазолу и фосфомицину.
Все изоляты были разделены на штаммы с множественной лекарственной устойчивостью (multidrug-resistant, MDR) и на штаммы с широкой лекарственной устойчивостью (extensively drug-resistant, XDR) в соответствии с рекомендациями G.J. German и соавт. [14]. Классификация основана на чувствительности микроорганизмов к 6 группам АМП: аминогликозидам (тобрамицин или гентамицин), пенициллинам (пиперациллин-тазобактам), карбапенемам (имипенем или меропенем), цефалоспоринам (цефотаксим, цефтриаксон или цефтазидим), фторхинолонам (ципрофлоксацин), сульфаниламидам (триметоприм-сульфаметоксазол). Изоляты, не чувствительные к 3 или 4 перечисленным группам, классифицировали как MDR, штаммы, не чувствительные к 5 или 6 группам, — как XDR.
Определение генов карбапенемаз
Бактериальную ДНК выделяли из суточной культуры с помощью коммерческих наборов «ГК-экспресс» (ЦНИИ Эпидемиологии). Карбапенемазы групп IMP, NDM, VIM, KPC, OXA-48 определяли с использованием наборов с гибридизационно-флуоресцентной детекцией «АмплиСенс MDR MBL-FL», «АмплиСенс MDR KPC/OXA-48-FL» (ЦНИИ Эпидемиологии). В качестве положительного и отрицательного контролей использовали образцы, входящие в состав набора. Реакцию амплификации проводили в соответствии с инструкцией производителя.
Определение факторов вирулентности и капсульных серотипов
Гены вирулентности и принадлежность к капсульным серотипам К1/К2 определяли методом мультиплексной ПЦР [15].
Изоляты были протестированы на наличие следующих генов:
- magA, специфичный для серотипа К1;
- rmpA, регулирующий синтез мукоидного фенотипа;
- entB, ассоциированный с синтезом энтеробактина;
- ybtS, связанный с синтезом йерсиниобактина;
- kfu, ответственный за связывание железа;
- iutA, кодирующий транспортер аэробактина;
- mrkD, ассоциированный с фимбриальными адгезинами 3-го типа;
- allS, связанный с метаболизмом аллантоина;
- wzi, специфичный для серотипа К2.
Ген entB был включён в дизайн исследования в качестве положительного контроля, т.к. широко распространён среди KP. Результаты реакции оценивали путём проведения электрофореза в 2% агарозном геле. Использованные праймеры представлены в табл. 1.
Таблица 1. Праймеры, использованные для определения генов вирулентности и серотипов K1/K2
Table 1. Primers used for identification of the virulence genes and K1/K2 serotypes
Праймер Primer | Последовательность 5’–3’ Sequence 5’–3’ | Размер продукта, п.о. Amplicon size, bp | Источник Source |
ybtS_f | GACGGAAACAGCACGGTAAA | 242 | [15] |
ybtS_r | GAGCATAATAAGGCGAAAGA | ||
mrkD_f | AAGCTATCGCTGTACTTCCGGCA | 340 | [15] |
mrkD_r | GGCGTTGGCGCTCAGATAGG | ||
entB_f | GTCAACTGGGCCTTTGAGCCGTC | 400 | [15] |
entB_r | TATGGGCGTAAACGCCGGTGAT | ||
rmpA_f | CATAAGAGTATTGGTTGACAG | 461 | [15] |
rmpA_r | CTTGCATGAGCCATCTTTCA | ||
K2_f | CAACCATGGTGGTCGATTAG | 531 | [16] |
K2_r | TGGTAGCCATATCCCTTTGG | ||
kfu_f | GGCCTTTGTCCAGAGCTACG | 638 | [15] |
kfu_r | GGGTCTGGCGCAGAGTATGC | ||
allS_f | CATTACGCACCTTTGTCAGC | 764 | [15] |
allS_r | GAATGTGTCGGCGATCAGCTT | ||
iutA_f | GGGAAAGGCTTCTCTGCCAT | 920 | [15] |
iutA_r | TTATTCGCCACCACGCTCTT | ||
magA_f | GGTGCTCTTTACATCATTGC | 1,283 | [17] |
magA_r | GCAATGGCCATTTGCGTTAG |
Мультилокусное секвенирование-типирование
Генотипирование штаммов KP проводили согласно схеме, предложенной Институтом Пастера [18], основанной на анализе аллельных профилей 7 «генов домашнего хозяйства»: rpoB (бета-субъединицы РНК-полимеразы), gapA (глицеральдегид-3-фосфат-дегидрогеназы), mdh (малатдегидрогеназы), pgi (фосфоглюкоза-изомеразы), phoE (фосфорина Е), infB (фактора инициации трансляции 2), tonB (периплазматического энергетического трансдуцера). Полученные нуклеотидные последовательности анализировали с помощью программы «SeqMan» и затем сравнивали с аллельными профилями базы данных BIGSdb1. Сиквенс-типы (SТ) идентифицировали на основании комбинации аллелей.
Результаты
Почти все отобранные штаммы (n = 87; 97%) продемонстрировали фенотип XDR, оставшиеся 3 изолята обладали фенотипом MDR. Все изоляты показали высокий уровень устойчивости к карбапенемам. К меропенему были нечувствительны 79 (88%) штаммов (МПК > 8 мг/л), 11 (12%) изолятов относились к категории чувствительных при повышенной экспозиции (МПК 2–8 мг/л). Резистентность к имипенему проявили 72 (80%) штамма (МПК > 4 мг/л), 16 (18%) изолятов были чувствительны к имипенему при повышенной экспозиции (МПК 2–4 мг/л), 2 (2%) штамма были чувствительны к имипенему (МПК < 2 мг/л).
Все исследованные штаммы были устойчивы к пиперациллину-тазобактаму, тикарциллину-клавуланату и тобрамицину. Высокая частота устойчивости была выявлена к цефтазидиму (98%), цефепиму (98%), азтреонаму (94%), фосфомицину (91%), амикацину (89%), гентамицину (83%), ципрофлоксацину (87%), левофлоксацину (87%), триметоприму-сульфаметоксазолу (87%), колистину (43%) (табл. 2).
Таблица 2. Устойчивость к антибиотикам изолятов K. pneumoniae
Table 2. Antibiotic resistance of K. pneumoniae isolates
Антибиотик Antibiotic | МПК, мг/л* MIC, mg/l* | Число резистентных изолятов Number of resistant isolates | ||
чувствительность ≤ susceptibility ≤ | резистентность > resistance > | n | % | |
Меропенем | Meropenem | 2 | 8 | 79 | 88 |
Имипенем | Imipenem | 2 | 4 | 72 | 80 |
Пиперациллин-тазобактам Piperacillin-tazobactam | 8 | 16 | 90 | 100 |
Тикарциллин-клавуланат Ticarcillin-clavulanate | 8 | 16 | 90 | 100 |
Цефтазидим | Ceftazidime | 1 | 4 | 88 | 98 |
Цефепим | Cefepime | 1 | 4 | 88 | 98 |
Амикацин | Amikacin | 8 | 8 | 80 | 89 |
Гентамицин | Gentamicin | 2 | 2 | 75 | 83 |
Тобрамицин | Tobramycin | 2 | 2 | 90 | 100 |
Ципрофлоксацин | Ciprofloxacin | 0,25 | 0,5 | 78 | 87 |
Левофлоксацин | Levofloxacin | 0,5 | 1 | 78 | 87 |
Триметоприм-сульфаметоксазол Trimethoprim-sulfamethoxazole | 2 | 4 | 78 | 87 |
Колистин | Colistin | 2 | 2 | 39 | 43 |
Азтреонам | Aztreonam | 1 | 4 | 85 | 94 |
Фосфомицин | Fosfomycin | 32 | 32 | 82 | 91 |
Примечание. *В соответствии с EUCAST [13].
Note. *According to EUCAST [13].
Продукция карбапенемаз обнаружена у всех исследованных изолятов KP. Главной детерминантой резистентности была карбапенемаза группы OXA-48 (n = 48; 53%), в 12 (13%) штаммах выявлена МБЛ группы NDM, сочетание карбапенемаз NDM и OXA-48 обнаружено у 28 (31%) изолятов. Два штамма обладали карбапенемазой группы KPC. Карбапенемаз VIM и IMP найдено не было.
При анализе факторов вирулентности выявлено, что все штаммы KP обладали геном entB (n = 90; 100%). Ген mrkD найден у всех исследованных штаммов (n = 89; 99%), за исключением 1 изолята. У большинства изолятов был обнаружен ген ybtS (n = 70; 78%). У 14 (16%) штаммов найден ген iutA. Четыре изолята показали наличие генов kfu, и у 4 штаммов был найден ген rmpA. Гена allS не было найдено ни в одном исследованном изоляте. Было обнаружено 7 комбинаций от 2 до 5 генов вирулентности. Комбинация 2 генов (mrkD + entB) и (ybtS + entB) найдена у 15 и 1 изолятов соответственно; 3 генов (ybtS + mrkD + entB) и (mrkD + entB + iutA) — у 56 и 5 штаммов соответственно; 4 генов (ybtS + mrkD + entB + iutA) и (ybtS + mrkD + entB + kfu) — у 5 и 4 изолятов соответственно; 5 генов (ybtS + mrkD + entB + rmpA + iutA) — у 4 изолятов. Двенадцать (13%) изолятов принадлежали к серотипу K2. Штаммов, относящихся к серотипу К1, не обнаружено (табл. 3).
Таблица 3. Распределение изолятов K. pneumoniae в зависимости от ST, носительства детерминант вирулентности и принадлежности к серотипу К2
Table 3. Distribution of K. pneumoniae isolates depending on the ST, carriage of virulence determinants, and belonging to K2 serotype
ST (n) | n | K2 | ybtS | mrkD | entB | rmpA | kfu | iutA |
ST395 (23) | 7 | + | + | + | + | |||
6 | + | + | ||||||
4 | + | + | + | |||||
3 | + | + | + | + | ||||
2 | + | + | + | + | + | + | ||
1 | + | + | ||||||
ST2975 (17) | 17 | + | + | + | ||||
ST198 (11) | 11 | + | + | + | ||||
ST307 (10) | 8 | + | + | + | ||||
1 | + | + | + | + | ||||
1 | + | + | + | + | ||||
ST377 (6) | 3 | + | + | + | ||||
3 | + | + | ||||||
ST29 (4) | 4 | + | + | + | ||||
ST147 (4) | 3 | + | + | + | ||||
1 | + | + | ||||||
ST397 (2) | 2 | + | + | + | + | |||
ST512 (2) | 2 | + | + | |||||
ST584 (2) | 1 | + | + | + | + | |||
1 | + | + | + | |||||
ST5829 (2) | 2 | + | + | + | + | + | + | |
ST 23(1) | 1 | + | + | + | + | |||
ST42 (1) | 1 | + | + | |||||
ST280 (1) | 1 | + | + | |||||
ST405 (1) | 1 | + | + | + | + | |||
ST985 (1) | 1 | + | + | + | ||||
ST3346 (1) | 1 | + | + | |||||
Новый ST (1) | New ST (1) | 1 | + | + | + | ||||
Всего | Total | 90 | 12 (13%) | 70 (78%) | 89 (99%) | 90 (100%) | 4 (4%) | 4 (4%) | 14 (16%) |
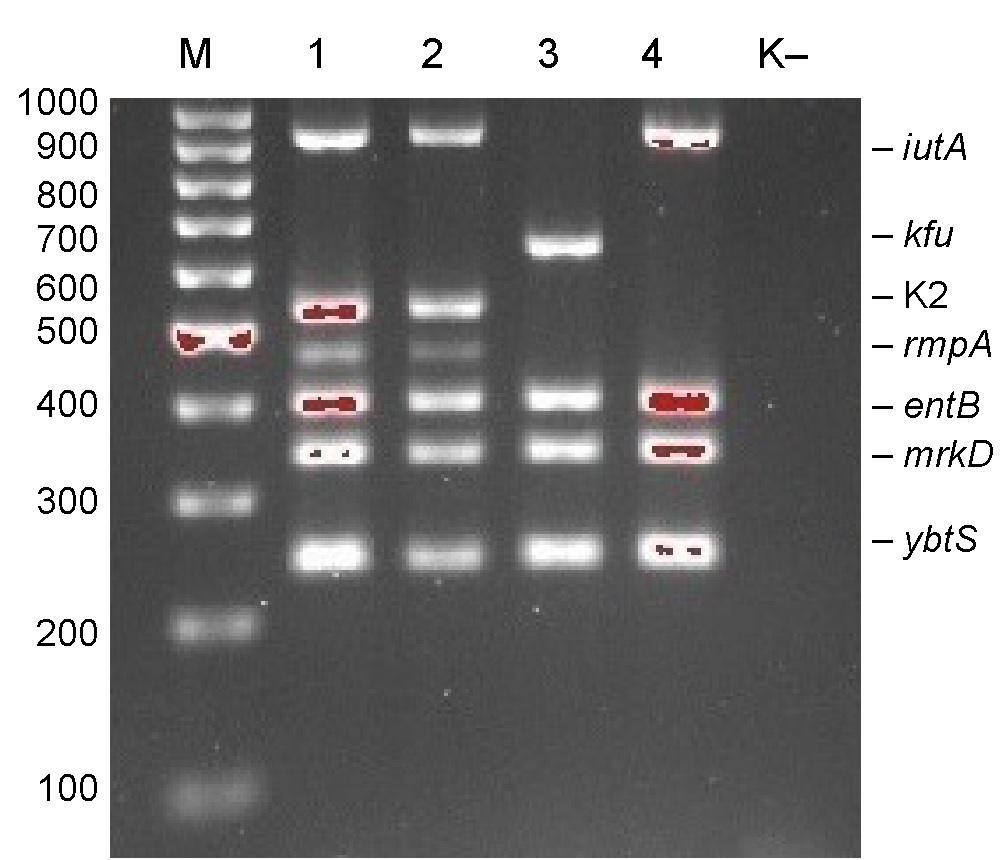
Результаты анализа амплификации генов вирулентности K. pneumoniae представлены на рисунке.
Анализ ПЦР-амплификации генов вирулентности КР при помощи электрофореза в 2% агарозном геле.
М — линейка молекулярных масс; штаммы КР: 1, 2 — ybtS, mrkD, entB, rmpA, серотип К2, iutA; 3 — ybtS, mrkD, entB, kfu; 4 — ybtS, mrkD, entB, iutA; K– — отрицательный контроль реакции.
Analysis of PCR amplification of K. pneumoniae virulence genes by electrophoresis in 2% agarose gel.
М — molecular size marker; K. pneumoniae strains: 1, 2 — ybtS, mrkD, entB, rmpA, K2 serotype, iutA; 3 — ybtS, mrkD, entB, kfu; 4 — ybtS, mrkD, entB, iutA; K– — negative control.
Наиболее вирулентные штаммы, сочетающие 4–5 генов вирулентности, были выделены из нижних дыхательных путей (n = 4/22), мочи (n = 5/41), ран (3/10), брюшной полости (n = 1/7). Из 28 штаммов, сочетающих 2 гена карбапенемаз, 15 (54%) приходилось на изоляты, выделенные из мочи. В штаммах из других локусов 2 гена карбапенемаз встречались у единичных изолятов: нижние дыхательные пути (n = 3), брюшная полость (n = 3), кровь (n = 3), рана (n = 4).
Большинство исследованных штаммов (n = 33; 37%) сочетали 1 ген резистентности к карбапенемам и 3 гена вирулентности: OXA-48 + ybtS + mrkD + entB (n = 22); OXA-48 + mrkD + enB + iutA (n = 3); NDM + ybtS + mrkD + entB (n = 8). Сочетание 2 генов резистентности и 3 генов вирулентности встречалось в 28 (31%) изолятах: OXA-48 + NDM + ybtS + mrkD + entB (n = 26); OXA-48 + NDM + mrkD + entB + iutA (n = 2). Шестнадцать (18%) штаммов обладали одним геном резистентности и 2 генами вирулентности: OXA-48 + mrkD + entB (n = 9); OXA-48 + ybtS + entB (n = 1); NDM + mrkD + entB (n = 4); KPC + mrkD + entB (n = 2). Комбинация 1 гена резистентности и 4–5 генов вирулентности встречалась у 13 (14%) штаммов: OXA-48 + ybtS + mrkD + entB + iutA (n = 5); OXA-48 + ybtS + mrkd + entB + kfu (n = 4); OXA-48 + ybtS + mrkD + entB + rmpA + iutA (n = 4).
При исследовании Карба-Р-штаммов KP методом мультилокусного сиквенс-типирования (МЛСТ) выявлены 18 различных ST, в том числе 3 изолята принадлежали к 2 новым, ранее не описанным ST. Один ST из них зарегистрирован в базе данных МЛСТ под номером ST5829 (n = 2), для него описан новый вариант гена tonB, которому присвоен номер аллеля 809. Второй ST с новым вариантом гена phoE (n = 1) — в процессе регистрации. Большая часть исследованной популяции (68%) принадлежала к 4 ST: ST395 (n = 23; 26%), ST2975 (n = 17; 19%), ST198 (n = 11; 12%) и ST307 (n = 10; 11%). Шесть (7%) штаммов относились к ST377. По 4 изолята принадлежали к ST29 и ST147, остальные ST были представлены 1–2 штаммами (табл. 4).
Таблица 4. Распределение изолятов K. pneumoniae в зависимости от ST и носительства детерминант резистентности, n = 90
Table 4. Distribution of K. pneumoniae isolates depending on ST and carriage of resistance determinants, n = 90
ST | n (%) | n | OXA-48 | NDM | KPC |
ST395 | 23 (26%) | 20 | + | ||
2 | + | + | |||
1 | + | ||||
ST2975 | 17 (19%) | 13 | + | + | |
4 | + | ||||
ST198 | 11 (12%) | 6 | + | + | |
5 | + | ||||
ST307 | 10 (11%) | 10 | + | ||
ST377 | 6 (7%) | 4 | + | ||
2 | + | + | |||
ST29 | 4 (4%) | 4 | + | + | |
ST147 | 4 (4%) | 2 | + | ||
1 | + | ||||
1 | + | + | |||
ST397 | 2 (2%) | 2 | + | ||
ST512 | 2 (2%) | 2 | + | ||
ST584 | 2 (2%) | 2 | + | ||
ST5829 | 2 (2%) | 2 | + | ||
ST23 | 1 (1%) | 1 | + | ||
ST42 | 1 (1%) | 1 | + | ||
ST280 | 1 (1%) | 1 | + | ||
ST405 | 1 (1%) | 1 | + | ||
ST985 | 1 (1%) | 1 | + | ||
ST3346 | 1 (1%) | 1 | + | ||
Новый ST | New ST | 1 (1%) | 1 | + |
Большинство штаммов ST395 (n = 23) ассоциировалось с карбапенемазой OXA-48 (n = 20). Девять из 12 штаммов серотипа К2 принадлежали к ST395. Среди ST2975 (n = 17) преобладали штаммы с сочетанием карбапенемаз OXA-48 и NDM (n = 13). Шесть штаммов ST198 сочетали карбапенемазы OXA-48 и NDM, а в 5 изолятах найдена только NDM. Все штаммы ST307 (n = 10) обладали единственной детерминантой резистентности OXA-48. Во всех изолятах ST29 (n = 4) найдена комбинация OXA-48 и NDM. Среди 4 изолятов ST147 2 показали наличие только NDM, 1 имел OXA-48, 1 обладал сочетанием OXA-48 + NDM. Два штамма, показавшие наличие KPC, относились к ST512. В изолятах, принадлежащих к ST397 (n = 2), ST584 (n = 2), ST5829 (n = 2), ST23 (n = 1), ST405 (n = 1), новому ST (n = 1), была найдена карбапенемаза OXA-48. В изолятах, принадлежащих к ST42 (n = 1), ST280 (n = 1), ST985 (n = 1) и ST3346 (n = 1), найдена МБЛ группы NDM.
Всего с временным интервалом было получено 20 штаммов от 9 пациентов. У 4 пациентов изоляты KP были выделены из разных локусов. Все штаммы имели фенотип XDR. Восемнадцать штаммов обладали одинаковыми детерминантами резистентности и вирулентности (за исключением 2 штаммов от 1 пациента с различием в носительстве гена kfu) и принадлежали к одинаковым ST. Два штамма, выделенные от 1 пациента из нижних дыхательных путей и мочи, относились к разным ST (ST395 и ST2975) и имели различия в носительстве карбапенемаз (OXA-48 и OXA-48 + NDM). Гены вирулентности у этих изолятов были одинаковыми.
Обсуждение
Результаты нашего исследования показали высокий уровень устойчивости к АМП среди Карба-Р-штаммов KP, полученных от пациентов, находившихся в реанимационных и хирургических отделениях НМИЦ здоровья детей и НИИ неотложной детской хирургии и травматологии — стационаров, куда поступают наиболее сложные пациенты из различных регионов России, часто после предшествующих госпитализаций в других учреждениях. Практически все изоляты обладали фенотипом XDR и были нечувствительны к 5–6 группам АМП.
По данным исследования МАРАФОН 2015–2016 гг., среди нозокомиальных изолятов KP высокий уровень устойчивости был выявлен к цефалоспоринам III–IV поколения (цефепиму — 87,6%; цефтазидиму — 87,3%) и азтреонаму (88,6%). Резистентность к имипенему и меропенему проявили 11,9 и 12,2% изолятов соответственно. Оценка чувствительности к аминогликозидам выявила, что резистентными к тобрамицину, гентамицину и амикацину были 81,3, 63,1, 27,1% штаммов KP соответственно. Высоким уровень устойчивости был к защищённым пенициллинам: тикарциллин-клавуланату — 92,5%, пиперациллин-тазобактаму — 66% изолятов. Резистентность к ципрофлоксацину, триметоприму-сульфамексазолу и фосфомицину проявили 83,6, 76,5, 51,2% изолятов соответственно. Высокую активность в отношении штаммов KP показал колистин, к нему были устойчивы только 9,4% изолятов [19].
В исследовании, проведённом нашими коллегами в 2012–2017 гг., был выявлен высокий уровень резистентности нозокомиальных штаммов KP к цефалоспоринам (100%), карбапенемам (93,1–98,7%), фторхинолонам (92,5%), фосфомицину (90,1%), аминогликозидам (49,7–84,3%) и колистину (44,7%) [20]. Исследование 2017–2019 гг. продемонстрировало высокие показатели устойчивости к АМП штаммов KP, выделенных от пациентов в отделении реанимации и интенсивной терапии: большинство штаммов имели фенотип XDR (87,3%). Все изоляты были резистентны к пенициллинам, цефалоспоринам и фторхинолонам. Высокий уровень устойчивости был выявлен к аминогликозидам (98,4%), карбапенемам (84,1%), сульфаниламидам (82,5%). К полимиксинам были нечувствительны 28,6% изолятов [21]. Полученные нами результаты подтверждают тенденцию к повышению уровня резистентности к важнейшим группам АМП нозокомиальных штаммов KP.
Главным механизмом резистентности среди исследованных изолятов KP стало носительство карбапенемазы OXA-48. Впервые карбапенемаза OXA-48 была обнаружена у KP в Турции в 2001 г. [22] и впоследствии распространилась по всему миру [23]. Продуцирующая OXA-48-подобные карбапанемазы KP эндемична для России [19, 24]. В исследованной нами популяции преобладал ST395 — клон высокого эпидемического риска, распространяющий карбапенемазы OXA-48 и KPC в Европе и Азии [4]. В России часто встречаются штаммы ST395, продуцирующие blaOXA-48 [20, 25], что согласуется с нашими данными.
Считается, что ST307 впервые появился в 1990-х гг., за 20 лет до его первого описания, и ответственен за внутрибольничные вспышки по всему миру [26]. Анализ, проведённый K.L. Wyres и соавт., показал, что ST307 является успешным MDR-клоном, источником генов различных карбапенемаз, а также содержит дополнительные факторы резистентности и вирулентности [26]. В исследовании, проведённом в Испании, получены данные о ST307, ассоциированном с карбапенемазой OXA-48 [27]. В работах из России были описаны изоляты ST307, продуцирующие blaOXA-48 [20, 21], что согласуется с нашими данными.
В изученной нами выборке ST2975 однолокусный вариант ST307 был вторым по встречаемости. Первый изолят ST2975, продуцирующий blaKPC-2, был выделен в Великобритании [28] и появился в международной базе данных МЛСТ Института Пастера в 2016 г. Мы не нашли других сообщений о ST2975 в зарубежных и отечественных исследованиях. Бóльшая часть штаммов ST2975 нашей коллекции обладала сочетанием карбапенемаз OXA-48 и NDM. Энтеробактерии, продуцирующие NDM, были выделены по всему миру из клинических образцов, окружающей среды и от животных [29].
Впервые штаммы KP ST198, выделенные в 2014–2016 гг., были описаны в исследованиях из Бразилии [30, 31]. R. Nakamura-Silva и соавт. обнаружили в штаммах ST198 карбапенемазы OXA-1 и KPC [31]. В изолятах ST198, выделенных от белого аиста в Алжире, была найдена МБЛ NDM-5 [32]. Сообщалось о штаммах KP ST198, выделенных из образцов животных и человека в США и Норвегии [33, 34]. В изученных нами изолятах ST198 преобладала комбинация карбапенемаз OXA-48 и NDM.
Мы обнаружили карбапенемазу группы KPC в 2 изолятах генотипа ST512, который относится к CG258. В отечественных исследованиях встречались единичные изоляты KP, продуцирующие KPC [20, 35, 36], что согласуется с нашими данными.
В последние годы появляется всё больше сообщений о появлении одновременно гипервирулентных и мультирезистентных штаммов KP [37, 38], хотя ранее считалось, что эти генетические линии не пересекаются [39]. Такие штаммы являются важной проблемой здравоохранения из-за высокого риска распространения и способности вызывать тяжёлые инфекции [40]. Считается, что KP серотипов К1/К2 более вирулентны, чем KP других серотипов [7]. По данным исследования H. Pan и соавт., 4,5% продуцирующих карбапенемазы штаммов KP относились к серотипу К1, 34,8% — к К2 [41]. Мы не обнаружили штаммов с серотипом К1, а к серотипу К2 принадлежало 13% изолятов. Интересно, что 11 из 12 штаммов серотипа К2 относились к ST395 и его однолокусному варианту ST5829. Похожие данные были получены при исследовании штаммов KP, выделенных в 12 больницах России в 2003–2020 гг. Капсульный серотип К2 был обнаружен у 15,7% штаммов и ассоциировался с ST395 [35].
Основными детерминантами вирулентности в нашем исследовании были гены сидерофоров entB и ybtS и ген mrkD, ассоциированный с фимбриями 3-го типа. Согласно анализу, проведённому D. Hu и соавт., распространённость генов entB и mrkD у KP составила более 90% [42]. В сообщениях из Китая и Индии ген entB встречался у 90,5–90,9% изолятов KP, mrkD — у 98,5%, ybtS — у 44,3–89,4% [41, 43]. В исследовании R. Han и соавт. выявлено, что гены сидерофоров entB и ybtS могут существенно увеличить вирулентность Карба-Р-штаммов KP [44]. Сидерофор аэробактин редко продуцируется классическими нозокомиальными штаммами, поскольку более характерен для гипервирулентных KP [7]. Ген транспортера аэробактина iutA встречался у 56,8% изолятов KP, выделенных от больных менингитом в Китае [45]. В другом исследовании ген iutA был выявлен у 48,5% KP, продуцирующих карбапенемазы [42]. В нашем исследовании iutA встречался у 16% штаммов. Гены rmpA и kfu обнаружены у единичных изолятов, что согласуется с данными других исследований, проведённых среди штаммов KP c фенотипом MDR [21, 41].
Результаты нашего исследования позволяют оценить характеристики Карба-Р-штаммов KP, полученных от пациентов педиатрических стационаров. Наряду с высокой антибиотикорезистентностью, штаммы KP имеют различные вирулентные свойства, что может быть причиной их распространения в стационарах с потенциальным увеличением тяжёлых форм бактериальных инфекций, а также негативно повлиять на успех лечения. Дальнейший мониторинг распространения факторов антибиотикорезистентности и вирулентности является важной задачей.
Заключение
В настоящей работе мы не оценивали распространённость Карба-Р KP в стационарах. Проведённое нами исследование выявило высокий уровень устойчивости Карба-Р-штаммов, выделенных в реанимационных и хирургических отделениях, не только к карбапенемам, но и к антибиотикам других классов. Устойчивость к карбапенемам главным образом была обусловлена карбапенемазой OXA-48 (53%). Преобладающими генетическими линиями были эпидемически успешные клоны CG395 (ST395 — n = 23, ST5829 — n = 2) и CG307 (ST2975 — n = 17, ST307 — n = 10). Главными детерминантами вирулентности были entB (100%), mrkD (99%) и ybtS (78%). Другие гены вирулентности встречались у незначительного числа изолятов, однако вызывает беспокойство сочетание антибиотикорезистентности и важных факторов вирулентности.
Этическое утверждение. Исследование проводилось при добровольном информированном согласии пациентов и их законных представителей. Протокол исследования одобрен Локальным независимым этическим комитетом Национального медицинского исследовательского центра здоровья детей (протокол № 5 от 02.06.2022).
Источник финансирования. Авторы заявляют об отсутствии внешнего финансирования при проведении исследования.
Конфликт интересов. Авторы декларируют отсутствие явных и потенциальных конфликтов интересов, связанных с публикацией настоящей статьи.
Участие авторов. Все авторы внесли существенный вклад в проведение поисково-аналитической работы и подготовку статьи, прочли и одобрили финальную версию до публикации.
Ethics approval. The study was conducted with the informed consent of the patients and their legal representatives. The research protocol was approved by the Local Independent Ethics Committee of the National Medical Research Center of Children's Health (protocol No. 5, June 2, 2022).
Funding source. This study was not supported by any external sources of funding.
Conflict of interest. The authors declare no apparent or potential conflicts of interest related to the publication of this article.
Author contribution. Аll authors made a substantial contribution to the conception of the work, acquisition, analysis, interpretation of data for the work, drafting and revising the work, final approval of the version to be published.
1 URL: https://bigsdb.pasteur.fr/klebsiella
About the authors
Irina E. Novikova
National Medical Research Center of Children's Health
Author for correspondence.
Email: novikovayudina@outlook.com
ORCID iD: 0000-0003-4234-0209
Junior Researcher, Laboratory of Molecular Microbiology
Russian Federation, MoscowZulfirya Z. Sadeeva
National Medical Research Center of Children's Health
Email: zulfiryasadeeva@yandex.ru
ORCID iD: 0000-0002-4587-0902
Junior Researcher, Laboratory of Molecular Microbiology
Russian Federation, MoscowNatalya M. Alyabyeva
National Medical Research Center of Children's Health
Email: bambolinka@hotmail.com
ORCID iD: 0000-0001-9365-9143
Cand. Sci. (Med.), Senior Researcher, Head, Laboratory of Experimental Immunology and Virology
Russian Federation, MoscowEkaterina A. Samoylova
National Medical Research Center of Children's Health
Email: katysamoilova@yandex.ru
ORCID iD: 0000-0002-5993-8285
Junior Researcher, Laboratory of Molecular Microbiology
Russian Federation, MoscowOlga V. Karaseva
National Medical Research Center of Children's Health; Clinical and Research Institute of Emergency Pediatric Surgery and Trauma
Email: karaseva.o@list.ru
ORCID iD: 0000-0001-9418-4418
D. Sci. (Med.), Deputy Director for Scientific Work, Head, Department of Combined Trauma, Anesthesiology and Resuscitation, Clinical and Research Institute of Emergency Pediatric Surgery and Trauma; Head, Department of Emergency Surgery and Pediatric Trauma, National Medical Research Center for Children's Health
Russian Federation, Moscow; MoscowOlga G. Yanyushkina
Clinical and Research Institute of Emergency Pediatric Surgery and Trauma
Email: spartak-lfc@mail.ru
ORCID iD: 0000-0002-6227-466X
Researcher, Department of Combined Trauma
Russian Federation, MoscowAnna V. Lazareva
National Medical Research Center of Children's Health
Email: annalaz71@mail.ru
ORCID iD: 0000-0003-3896-2590
D. Sci. (Med.), Chief Researcher, Laboratory of Molecular Microbiology, Head, Laboratory of Microbiology
Russian Federation, MoscowReferences
- Santajit S., Indrawattana N. Mechanisms of antimicrobial resistance in ESKAPE pathogens. Biomed. Res. Int. 2016; 2016:2475067. DOI: https://doi.org/10.1155/2016/2475067
- Ding L., Yang Z., Lu J., et al. Characterization of phenotypic and genotypic traits of Klebsiella pneumoniae from lung cancer patients with respiratory infection. Infect. Drug. Resist. 2020;13:237–45. DOI: https://doi.org/10.2147/idr.s229085
- Aghapour Z., Gholizadeh P., Ganbarov K., et al. Molecular mechanisms related to colistin resistance in Enterobacteriaceae. Infect. Drug. Resist. 2019;12:965–75. DOI: https://doi.org/10.2147/idr.s199844
- Шамина О.В., Самойлова Е.А., Новикова И.Е., Лазарева А.В. Klebsiella pneumoniae: микробиологическая характеристика, антибиотикорезистентность и вирулентность. Российский педиатрический журнал. 2020;23(3):191–7. Shamina O.V., Samoilova E.A., Novikova I.E., Lazareva A.V. Klebsiella pneumoniae: microbiological characterization, antimicrobial resistance, and virulence. The Russian Journal of Pediatrics. 2020;23(3):191–7. DOI: https://doi.org/10.18821/1560-9561-2020-23-3-191-197 EDN: https://elibrary.ru/fytman
- Liu Y., Cheng D., Lin C. Klebsiella pneumoniae liver abscess associated with septic endophthalmitis. Arch. Intern. Med. 1986;146(10):1913–6. DOI: https://doi.org/10.1001/archinte.1986.00360220057011
- Gu D., Dong N., Zheng Z., et al. A fatal outbreak of ST11 carbapenem-resistant hypervirulent Klebsiella pneumoniae in a Chinese hospital: a molecular epidemiological study. Lancet. Infect. Dis. 2018;18(1):37–46. DOI: https://doi.org/10.1016/s1473-3099(17)30489-9
- Paczosa M.K., Mecsas J. Klebsiella pneumoniae: going on the offense with a strong defense. Microbiol. Mol. Biol. Rev. 2016;80(3):629–61. DOI: https://doi.org/10.1128/mmbr.00078-15
- Fang C.T., Lai S.Y., Yi W.C., et al. The function of wzy_K1 (magA), the serotype K1 polymerase gene in Klebsiella pneumoniae cps gene cluster. J. Infect. Dis. 2010;201(8):1268–9. DOI: https://doi.org/10.1086/652183
- Shon A.S., Bajwa R.P., Russo T.A. Hypervirulent (hypermucoviscous) Klebsiella pneumoniae: a new and dangerous breed. Virulence. 2013;4(2):107–18. DOI: https://doi.org/10.4161/viru.22718
- Cubero M., Grau I., Tubau F., et al. Hypervirulent Klebsiella pneumoniae clones causing bacteraemia in adults in a teaching hospital in Barcelona, Spain (2007–2013). Clin. Microbiol. Infect. 2016;22(2):154–60. DOI: https://doi.org/10.1016/j.cmi.2015.09.025
- Clegg S., Murphy C.N. Epidemiology and virulence of Klebsiella pneumoniae. Microbiol. Spectr. 2016;4(1). DOI: https://doi.org/10.1128/microbiolspec.uti-0005-2012
- Yu W.L., Ko W.C., Cheng K.C., et al. Comparison of prevalence of virulence factors for Klebsiella pneumoniae liver abscesses between isolates with capsular K1/K2 and non-K1/K2 serotypes. Diagn. Microbiol. Infect. Dis. 2008;62(1):1–6. DOI: https://doi.org/10.1016/j.diagmicrobio.2008.04.007
- European Committee on Antimicrobial Susceptibility testing (EUCAST). Breakpoint tables for interpretation of MICs and zone diameters. Ver. 10.0; 2020. URL: https://www.eucast.org/clinical_breakpoints
- German G.J., Gilmour M., Tipples G., et al. Canadian recommendations for laboratory interpretation of multiple or extensive drug resistance in clinical isolates of Enterobacteriaceae, Acinetobacter species and Pseudomonas aeruginosa. Can. Commun. Dis. Rep. 2018;44(1):29–34. DOI: https://doi.org/10.14745/ccdr.v44i01a07
- Compain F., Babosan A., Brisse S., et al. Multiplex PCR for detection of seven virulence factors and K1/K2 capsular serotypes of Klebsiella pneumoniae. J. Clin. Microbiol. 2014;52(12): 4377–80. DOI: https://doi.org/10.1128/jcm.02316-14
- Yu W.L., Fung C.P., Ko W.C., et al. Polymerase chain reaction analysis for detecting capsule serotypes K1 and K2 of Klebsiella pneumoniae causing abscesses of the liver and other sites. J. Infect. Dis. 2007;195(8):1235–6. DOI: https://doi.org/10.1086/512686
- Fang C.T., Chuang Y.P, Shun C.T., et al. A novel virulence gene in Klebsiella pneumoniae strains causing primary liver abscess and septic metastatic complications. J. Exp. Med. 2004;199(5):697–705. DOI: https://doi.org/10.1084/jem.20030857
- Diancourt L., Passet V., Verhoef J., et al. Multilocus sequence typing of Klebsiella pneumoniae nosocomial isolates. J. Clin. Microbiol. 2005;43(8):4178–82. DOI: https://doi.org/10.1128/jcm.43.8.4178-4182.2005
- Сухорукова М.В., Эйдельштейн М.В., Иванчик Н.В. и др. Антибиотикорезистентность нозокомиальных штаммов Enterobacterales в стационарах России: результаты многоцентрового эпидемиологического исследования МАРАФОН 2015-2016. Клиническая микробиология и антимикробная химиотерапия. 2019;21(2):147–59. Sukhorukova M.V., Eidel'shtein M.V., Ivanchik N.V., et al. Antimicrobial resistance of nosocomial Enterobacterales isolates in Russia: results of multicenter epidemiological study «Marathon 2015-2016». Clinical Microbiology and Antimicrobial Chemotherapy. 2019;21(2):147–59. DOI: https://doi.org/10.36488/cmac.2019.2.147-159 EDN: https://elibrary.ru/qdarvm
- Shamina O.V., Kryzhanovskaya O.A., Lazareva A.V., et al. Emergence of a ST307 clone carrying a novel insertion element MITEKpn1 in the mgrB gene among carbapenem-resistant Klebsiella pneumoniae from Moscow, Russia. Int. J. Antimicrob. Agents. 2020;55(2):105850. DOI: https://doi.org/10.1016/j.ijantimicag.2019.11.007
- Fursova N.K., Astashkin E.I., Gabrielyan N.I., et al. Emergence of five genetic lines ST395NDM-1, ST13OXA-48, ST3346OXA-48, ST39CTX-M-14, and novel ST3551OXA-48 of multidrug-resistant clinical Klebsiella pneumoniae in Russia. Microb. Drug. Resist. 2020;26(8):924–33. DOI: https://doi.org/10.1089/mdr.2019.0289
- Poirel L., Héritier C., Tolün V., Nordmann P. Emergence of oxacillinase mediated resistance to imipenem in Klebsiella pneumoniae. Antimicrob. Agents Chemother. 2004;48(1):15–22. DOI: https://doi.org/10.1128/aac.48.1.15-22.2004
- Potron A., Poirel L., Rondinaud E., Nordmann P. Intercontinental spread of OXA48 beta-lactamase-producing Enterobacteriaceae over a 11-year period, 2001 to 2011. Euro Surveill. 2013;18(31):20549. DOI: https://doi.org/10.2807/1560-7917.es2013.18.31.20549
- Новикова И.Е., Садеева З.З., Шакирзянова Р.А. и др. Использование полимеразной цепной реакции для детекции генов резистентности у грамотрицательных бактерий в рутинной практике педиатрического стационара. Клиническая лабораторная диагностика. 2022;67(3):180–5. Novikova I.E., Sadeeva Z.Z., Shakirzyanova R.A., et al. The using of the polymerase chain reaction for the detection of resistance genes in gram-negative bacteria in routine practice in a pediatric hospital. Russian Clinical Laboratory Diagnostics. 2022;67(3):180–5. DOI: https://doi.org/10.51620/0869-2084-2022-67-3-180-185 EDN: https://elibrary.ru/akcthf
- Шайдуллина Э.Р., Эйдельштейн М.В., Склеенова Е.Ю. и др. Антибиотикорезистентность нозокомиальных карбапенемазопродуцирующих штаммов Enterobacterales в России: результаты эпидемиологического исследования 2014–2016 гг. Клиническая микробиология и антимикробная химиотерапия. 2018;20(4):362–9. Shaidullina E.R., Eidel'shtein M.V., Skleenova E.Yu., et al. Antimicrobal resistance of nosocomial carbapenemase-producing Enterobacterales in Russia: results of surveillance, 2014-2016. Russian Clinical Laboratory Diagnostics. 2018;20(4):362–9. DOI: https://doi.org/10.36488/cmac.2018.4.362-369 EDN: https://elibrary.ru/zaghhn
- Wyres K.L., Hawkey J., Hetland M.A.K., et al. Emergence and rapid global dissemination of CTX-M-15-associated Klebsiella pneumoniae strain ST307. J. Antimicrob. Chemother. 2019;74(3):577–81. DOI: https://doi.org/10.1093/jac/dky492
- Cañada-García J.E., Moure Z., Sola-Campoy P.J., et al. CARB-ES-19 multicenter study of carbapenemase-producing Klebsiella pneumoniae and Escherichia coli from all Spanish provinces reveals interregional spread of high-risk clones such as ST307/OXA-48 and ST512/KPC-3. Front. Microbiol. 2022;13:918362. DOI: https://doi.org/10.3389/fmicb.2022.918362
- Villa L., Feudi C., Fortini D., et al. Diversity, virulence, and antimicrobial resistance of the KPC-producing Klebsiella pneumoniae ST307 clone. Microb. Genom. 2017;3(4):e000110. DOI: https://doi.org/10.1099/mgen.0.000110
- Khan A.U., Maryam L., Zarrilli R. Structure, genetics and worldwide spread of New Delhi metallo-β-lactamase (NDM): a threat to public health. BMC Microbiol. 2017;17(1):101. DOI: https://doi.org/10.1186/s12866-017-1012-8
- Lopes R., Cerdeira L.T., Fernandes M.R., et al. Draft genome sequence of a CTX-M-15-producing endophytic Klebsiella pneumoniae ST198 isolate from commercial lettuce. J. Glob. Antimicrob. Resist. 2017;10:19–20. DOI: https://doi.org/10.1016/j.jgar.2017.03.005
- Nakamura-Silva R., Cerdeira L., Oliveira-Silva M., et al. Multidrug-resistant Klebsiella pneumoniae: a retrospective study in Manaus, Brazil. Arch. Microbiol. 2022;204(4):202. DOI: https://doi.org/10.1007/s00203-022-02813-0
- Loucif L., Chelaghma W., Cherak Z., et al. Detection of NDM-5 and MCR-1 antibiotic resistance encoding genes in Enterobacterales in long-distance migratory bird species Ciconia ciconia, Algeria. Sci. Total. Environ. 2022;814:152861. DOI: https://doi.org/10.1016/j.scitotenv.2021.152861
- Holt K.E., Wertheim H., Zadoks R.N., et al. Genomic analysis of diversity, population structure, virulence, and antimicrobial resistance in Klebsiella pneumoniae, an urgent threat to public health. Proc. Natl Acad. Sci. USA. 2015;112(27):E3574–81. DOI: https://doi.org/10.1073/pnas.1501049112
- Raffelsberger N., Hetland M.A.K., Svendsen K., et al. Gastrointestinal carriage of Klebsiella pneumoniae in a general adult population: a cross-sectional study of risk factors and bacterial genomic diversity. Gut Microbes. 2021;13(1):1939599. DOI: https://doi.org/10.1080/19490976.2021.1939599
- Khrulnova S., Fedorova A., Frolova I., et al. Distribution of virulence genes and capsule types in Klebsiella pneumoniae among bloodstream isolates from patients with hematological malignancies. Diagn. Microbiol. Infect. Dis. 2022;104(1):115744. DOI: https://doi.org/10.1016/j.diagmicrobio.2022.115744
- Лазарева И.В., Агеевец В.А., Ершова Т.А. и др. Распространение и антибактериальная резистентность грамотрицательных бактерий, продуцентов карбапенемаз, в Санкт-Петербурге и некоторых других регионах Российской Федерации. Антибиотики и химиотерапия. 2016;61(11–12):28–38. Lazareva I.V., Ageevets V.A., Ershova T.A., et al. The spread and antibacterial resistance of gram-negative bacteria, carbapenemase producers, in St. Petersburg and some other regions of the Russian Federation. Antibiotics and Chemoterapy. 2016;61(11–12):28–38. EDN: https://elibrary.ru/yjtpzr
- Karlsson M., Stanton R.A., Ansari U., et al. Identification of a carbapenemase-producing hypervirulent Klebsiella pneumoniae isolate in the United States. Antimicrob. Agents Chemother. 2019;63(7):e00519–19. DOI: https://doi.org/10.1128/aac.00519-19
- Yonekawa S., Mizuno T., Nakano R., et al. Molecular and epidemiological characteristics of carbapenemase-producing Klebsiella pneumoniae clinical isolates in Japan. mSphere. 2020;5(5):e00490–20. DOI: https://doi.org/10.1128/msphere.00490-20
- Bialek-Davenet S., Criscuolo A., Ailloud F., et al. Genomic definition of hypervirulent and multidrug-resistant Klebsiella pneumoniae clonal groups. Emerg. Infect. Dis. 2014;20(11): 1812–20. DOI: https://doi.org/10.3201/eid2011.140206
- Lan P., Jiang Y., Zhou J., Yu Y. A global perspective on the convergence of hypervirulence and carbapenem resistance in Klebsiella pneumoniae. J. Glob. Antimicrob. Resist. 2021;25:26–34. DOI: https://doi.org/10.1016/j.jgar.2021.02.020
- Pan H., Lou Y., Zeng L., et al. Infections caused by carbapenemase-producing Klebsiella pneumoniae: microbiological characteristics and risk factors. Microb. Drug Resist. 2019; 25(2):287–96. DOI: https://doi.org/10.1089/mdr.2018.0339
- Hu D., Li Y., Ren P., et al. Molecular epidemiology of hypervirulent carbapenemase-producing Klebsiella pneumoniae. Front. Cell. Infect. Microbiol. 2021;11:661218. DOI: https://doi.org/10.3389/fcimb.2021.661218
- Remya P.A., Shanthi M., Sekar U. Characterisation of virulence genes associated with pathogenicity in Klebsiella pneumoniae. Indian J. Med. Microbiol. 2019;37(2):210–8. DOI: https://doi.org/10.4103/ijmm.ijmm_19_157
- Han R., Niu M., Liu S., et al. The effect of siderophore virulence genes entB and ybtS on the virulence of carbapenem-resistant Klebsiella pneumoniae. Microb. Pathog. 2022;171:105746. DOI: https://doi.org/10.1016/j.micpath.2022.105746
- Xu M., Fu Y., Fang Y., et al. High prevalence of KPC-2-producing hypervirulent Klebsiella pneumoniae causing meningitis in Eastern China. Infect. Drug Resist. 2019;12:641–53. DOI: https://doi.org/10.2147/idr.s191892
Supplementary files