Recombinase polymerase amplification: method’s characteristics and applications in diagnostics of infectious diseases
- Authors: Bondareva O.S.1, Baturin A.A.1, Mironova A.V.1
-
Affiliations:
- Volgograd Plague Control Research Institute
- Issue: Vol 101, No 2 (2024)
- Pages: 270-280
- Section: REVIEWS
- URL: https://microbiol.crie.ru/jour/article/view/18478
- DOI: https://doi.org/10.36233/0372-9311-470
- EDN: https://elibrary.ru/zphlvm
- ID: 18478
Cite item
Full Text
Abstract
Isothermal amplification techniques have been actively developed in recent years and are gradually introduced into the range of methods for infectious disease diagnostics. One of the fastest isothermal methods is recombinase polymerase amplification (RPA). This review contains information about the principle of RPA, the role of individual reaction components and primer design considerations. It provides information on characteristics of various methods of RPA results detection, effects of inhibitors, temperature and agitation on the efficiency of reaction. Approaches to quantitative and multiplex RPA are described, as well as some variants of portable devices designed to identify infectious agents. The conclusion summarizes advantages and disadvantages of RPA in comparison with other amplification methods.
Keywords
Full Text
Введение
Методы амплификации нуклеиновых кислот (НК) широко используются в лабораторной диагностике для выявления возбудителей инфекционных заболеваний, определения вирусной нагрузки, генотипирования патогенов. В последние годы интенсивно развиваются методы, основанные на изотермической амплификации. Протекание реакции при одной температуре позволяет проводить анализ в условиях ограниченных ресурсов, а также сокращает время исследования.
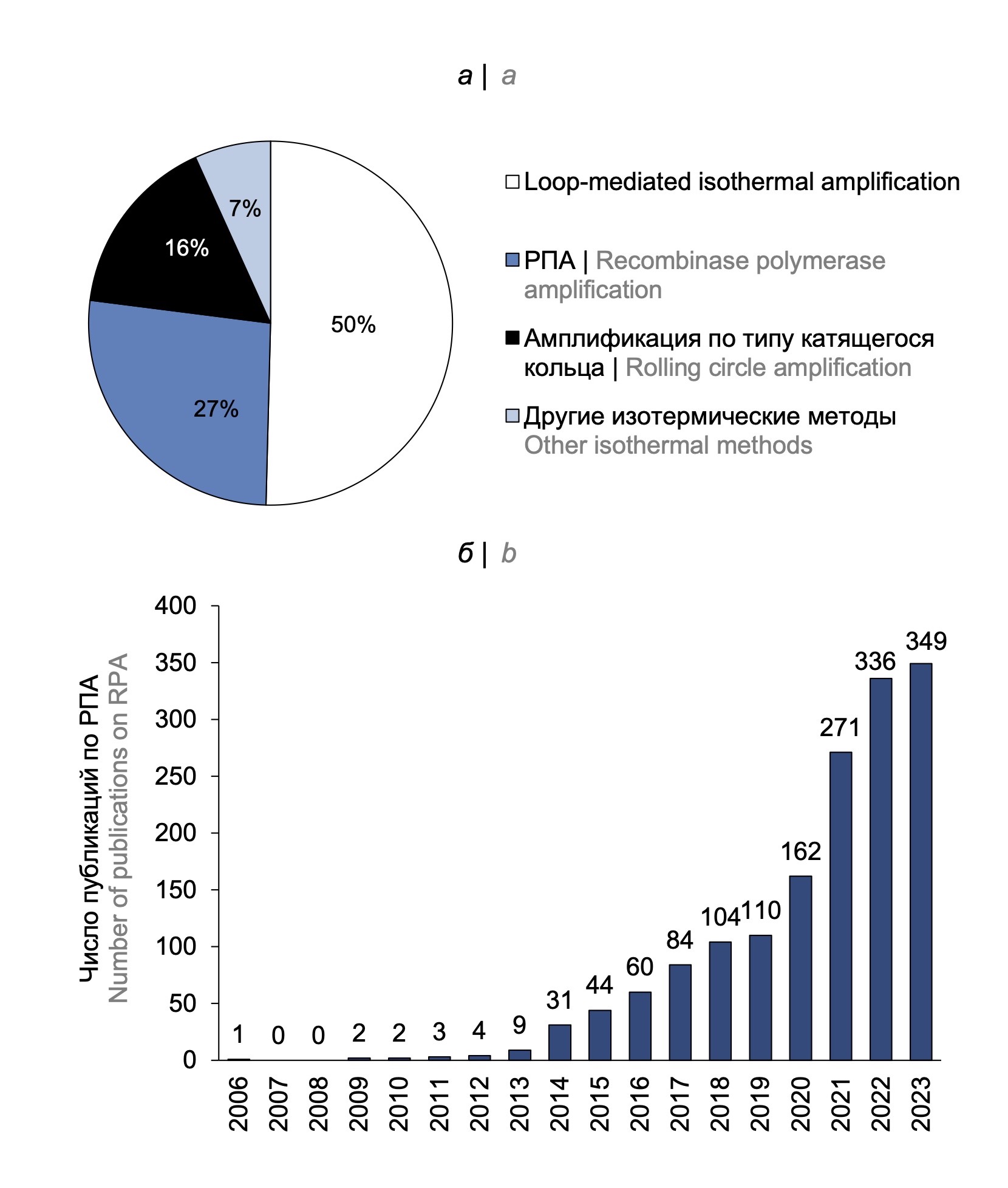
Анализ числа публикаций в базе данных PubMed, посвящённых методам изотермической амплификации, за последние 3 года показал, что наиболее популярным методом является петлевая изотермическая амплификация (Loop-mediated isothermal amplification; LAMP), использованию которой посвящено около половины статей, на втором месте — рекомбиназная полимеразная амплификация (РПА; 27% статей), затем амплификация по типу катящегося кольца — 16% публикаций, остальные изотермические методы в совокупности заняли менее 7% от общего числа публикаций (рис. 1, а).
Рис. 1. Анализ публикационной активности по методам изотермической амплификации согласно базе данных PubMed NCBI (а) за последние 3 года; б — по РПА.
Fig. 1. Analysis of publication activity on isothermal amplification methods according to the PubMed NCBI database (a) over the past 3 years; b — on RPA.
Метод РПА был предложен относительно недавно, в 2006 г., группой учёных под руководством N.A. Armes [1]. В последние годы отмечается ежегодный рост числа публикаций, посвящённых РПА (рис. 1, б), что свидетельствует об активном интересе к данной технологии.
Принцип метода РПА заключается в моделировании in vitro естественного биологического процесса — гомологичной рекомбинации; при этом денатурация двойной цепи ДНК осуществляется благодаря действию нескольких ферментов и реагентов (рис. 2).
Рис. 2. Механизм РПА.
Fig. 2. Schematic representation of recombinase polymerase amplification.
Основной фермент РПА — рекомбиназа — является гомологом белка RecA Escherichia coli. В процессе гомологичной рекомбинации RecA связывается с одноцепочечной ДНК, осуществляет поиск комплементарной последовательности на гомологичной хромосоме, внедряется в неё, образуя гетеродуплекс и вытесняя вторую цепь.
Реакция РПА начинается с присоединения рекомбиназы к праймерам посредством белка T4 UvsY. Сформировавшийся нуклеопротеиновый комплекс при сближении с гомологичной последовательностью двухцепочечной ДНК расплетает её, способствуя отжигу праймера. При этом образуется D-петля. Раскрученные одноцепочечные нити стабилизируют SSB белки T4 gp32. После отсоединения рекомбиназы происходят посадка Bsu-полимеразы (или Sau-полимеразы) и элонгация целевой мишени с вытеснением комплементарной цепи. В результате образуются два дуплекса, которые выступают в качестве матрицы при экспоненциальной амплификации. Фактор краудинга (высокомолекулярный полиэтиленгликоль, PEG 35K) способствует имитации условий in vivo и катализирует работу ферментов. Дитиотреитол стабилизирует работу ферментов; фосфокреатинин, креатинкиназа и аденозинтрифосфат обеспечивают систему энергией. Дезоксирибонуклеозидтрифосфаты используются для синтеза ампликонов. Для поддержания необходимого pH применяют буфер, содержащий трис и другие соли. Ацетат магния выступает в роли кофактора ферментов и инициирует РПА [2].
При добавлении в реакционную смесь обратной транскриптазы в качестве матрицы в РПА может выступать РНК, что используется при выявлении вирусов с РНК-геномом [3–5].
Аналогом РПА является метод Recombinase Aided Amplification (RAA), отличающийся источником происхождения ферментов [6, 7]. Учитывая общий принцип действия, в дальнейшем будут рассмотрены особенности РПА на основе публикаций по методам РПА и RAA.
Требования, предъявляемые к выбору праймеров и мишени для РПА, существенно отличаются от аналогичных в полимеразной цепной реакции (ПЦР). Так, размер мишени не должен превышать 500 п.н. Применяются ампликоны как менее 100 п.н., так и значительно большей длины. Оптимальный размер мишени, при котором обеспечивается эффективная амплификация, составляет 100–250 п.н. [8, 9].
В руководстве по РПА [8] описаны следующие характеристики праймеров: рекомендуемая длина 30–35 нуклеотидов, отсутствие однонуклеотидных и SSR-повторов, палиндромов, GC-состав в диапазоне 40–60%, отсутствие вторичных структур, таких как праймер-димеры, шпильки, которые могут привести к артефактам амплификации. Наличие на 3'-конце гуанозина или цитидина повышает производительность праймера. Олигонуклеотиды меньшей длины также могут использоваться для амплификации. Существует ряд публикаций о применении праймеров для ПЦР в РПА, однако считается, что связывание рекомбиназы с короткими праймерами менее эффективно, что сказывается на чувствительности реакции [10, 11]. Больший размер праймеров повышает вероятность образования вторичных структур и димеров, что также нежелательно. В работе N. Sharma и соавт. [12] продемонстрировано использование модифицированных нуклеотидов, которые не связываются между собой, но образуют водородные связи с нуклеотидами матрицы, что позволяет избежать образования артефактных продуктов амплификации. В отличие от ПЦР, при выборе праймеров для РПА не важна расчётная температура их отжига, в требованиях указан диапазон 50–100ºC. Ведутся работы по созданию приложений для подбора РПА-праймеров с учётом существующих критериев [13]. Но даже в руководстве по РПА указано, что на данный момент невозможно предсказать эффективность амплификации с конкретными праймерами, поэтому целесообразны выбор нескольких вариантов и их проверка эмпирическим путём [8].
Чувствительность и специфичность
Аналитическая чувствительность и специфичность — это ключевые характеристики любого метода, применяемого для обнаружения возбудителей инфекционных заболеваний. Чувствительность РПА зависит от конкретных праймеров. Как показано в ряде работ [14, 15], в зависимости от расположения праймера на целевой ДНК меняется эффективность амплификации. Длина праймера также имеет большое значение. Так, при проверке различных комбинаций праймеров, незначительно варьируемых по длине, месту отжига и не образующих вторичных структур in silico, в ряде случаев ампликоны не образовывались вовсе, либо чувствительность реакции отличалась на несколько порядков [16]. В литературе есть данные о чувствительности РПА, сопоставимой и даже выше, чем в ПЦР [17]. Однако стоит учитывать, что для РПА чувствительность часто считают в числе копий НК, попадающих в реакцию. Это связано с тем, что в стандартном протоколе РПА добавляется всего 1 мкл анализируемой пробы [8], тогда как в ПЦР обычно вносят 5–10 мкл.
Особенностью действия рекомбиназы является её способность распознавать комплементарный участок, даже если в нём есть однонуклеотидные замены. Биологический смысл данного явления — осуществление репарации повреждённой ДНК. Рекомбиназа распознаёт участки целевой последовательности при наличии в них большого числа замен (до 9) [2]. Как и в ПЦР, наибольшее значение имеет комплементарность нуклеотидов на 3'-конце праймеров, поскольку именно этот участок важен для начала работы полимеразы. С одной стороны, это может быть преимуществом при выборе целевой мишени для вирусных высоковариабельных геномов, с другой стороны, это повышает риск неспецифической амплификации и перекрёстного обнаружения близкородственных видов [18].
Методы детекции
Детекция с помощью электрофореза позволяет оценить результат амплификации по наличию ампликонов специфической длины. В реакционной смеси РПА содержится много белков, большая часть ампликонов после реакции связана с белками и агентами краудинга, что влияет на подвижность ампликонов в геле при электрофорезе и приводит к появлению артефактов на электрофореграмме. Существует несколько способов обработки реакционной смеси для денатурации белков и устранения искажений. Удобным, достаточно эффективным и поэтому часто используемым методом является прогрев при 65ºC в течение 10 мин [19]. Иногда для этой цели смесь прогревают при 95ºC, возможны обработка детергентом (SDS), ферментативное расщепление (протеиназой K), высокоскоростное центрифугирование [2, 19, 20].
Применение электрофореза в качестве способа детекции результатов РПА нерационально, поскольку занимает больше времени, чем сама реакция, и требует специального оборудования, поэтому обычно используется только на этапе подбора и проверки праймеров.
Детекция с помощью зондов. Флуоресцентно меченные зонды, применяемые в ПЦР, не подходят для РПА: для молекулярных маяков температура реакции является слишком низкой, а отсутствие экзонуклеазной активности Bsu-полимеразы не позволяет использовать зонды типа TaqMan. Поэтому для флуоресцентной детекции разработаны специальные зонды и нуклеазы (рис. 3).
Рис. 3. Структура зондов и принцип детекции при использовании различных типов нуклеаз. а — экзонуклеаза III; б — экзонуклеаза Fpg; в — эндонуклеаза IV.
Fig. 3. Structure of probes and detection principles with different types of nucleases. a — exonuclease III; b — exonuclease Fpg; c — endonuclease IV).
При использовании экзонуклеазы III выбирают зонд длиной 45–50 нуклеотидов, в котором флуорофор и гаситель должны быть связаны с тимином на расстоянии примерно 30–40 нуклеотидов от начала и на расстоянии 2–5 нуклеотидов друг от друга. Между флуорофором и гасителем вместо одного из нуклеотидов помещают остаток тетрагидрофурана, который в случае гибридизации зонда разрезается экзонуклеазой, и при этом генерируется флуоресцентный сигнал.
Мишенью для экзонуклеазы Fpg служит линкер C-O-C (так называемая dR-группа, например, карбоксифлуоресцеин), с помощью которого флуорофор крепится к зонду. После гибридизации зонда с мишенью Fpg расщепляет dR-остаток, флуорофор освобождается от действия гасителя и испускает флуоресцентный сигнал [2, 8].
Преимуществами флуоресцентной детекции являются высокая скорость получения результата амплификации, точность анализа, отсутствие необходимости дополнительных манипуляций. Однако использование меченых олигонуклеотидов, ферментов и специального оборудования повышает стоимость анализа. Тем не менее данный подход может применяться для одновременной амплификации различных мишеней при проведении РПА в мультиплексном формате [4, 21–23].
Эндонуклеазу IV обычно используют при детекции с помощью иммунохроматографических тест-полосок. Зонд и обратный праймер на 5'-конце содержат различающиеся антигенные метки. Дополнительно в зонд на расстоянии 30–35 нуклеотидов от начала встраивают остаток тетрагидрофурана, который разрезается эндонуклеазой при гибридизации зонда с мишенью, а на 3'-конце — блок, препятствующий неспецифической амплификации. После действия нуклеазы зонд выполняет роль прямого праймера. В результате амплификации образуется фрагмент, меченный с 2 сторон разными антигенами, который может быть обнаружен посредством иммунохроматографических тест-полосок. Дополнительно для инициации амплификации реакционная смесь может содержать немеченый прямой праймер. В отличие от экзонуклеазы III, которая расщепляет большую долю двухцепочечной ДНК, эндонуклеаза IV действует точнее и не расщепляет ампликоны, поэтому после её использования результат РПА можно проверять методом электрофореза.
Детекция на основе иммунохроматографических тест-полосок отличается быстротой получения результата, простотой проведения, отсутствием необходимости специального оборудования, однако открытие пробирок с ампликонами повышает риск контаминации. Существуют устройства для проведения иммунохроматографического анализа, представляющие собой герметичный картридж, в котором изолированно расположены тест-полоски, что позволяет предотвратить риск контаминации продуктами реакции, но их использование значительно повышает себестоимость каждого теста.
SHERLOCK. Одним из наиболее активно развивающихся методов амплификации НК является «Specifc High-Sensitivity Enzymatic Reporter Unlocking» (SHERLOCK), основанный на сочетании РПА и детекции с помощью системы CRISPR–Cas13 [24]. В методике SHERLOCK после выделения НК проводят изотермическую амплификацию мишени методом РПА или LAMP. Далее T7-транскриптазу применяют для синтеза на основе ампликонов комплементарных РНК. Способность распознавать целевую последовательность и проявлять побочную (неспецифическую) нуклеазную активность Cas13 используют для детекции РНК-мишеней. Специфичность обеспечивается проявлением нуклеазной активности Cas13 только после связи crРНК и мишени, которая запускает расщепление флуоресцентно меченных репортёрных РНК (короткие РНК, меченные биотином, флуорофором и гасителем). РНК-репортеры служат для усиления сигнала. После их расщепления в результате побочной нуклеазной активности Cas13 образуются фрагменты, меченные биотином и флуорофором, которые можно обнаружить посредством иммунохроматографических тест-полосок. Сочетание Cas13 с Cas12 в одном тесте позволяет одновременно обнаруживать несколько мишеней в рамках одной реакции.
На основе системы CRISPR–Cas разработаны различные диагностические платформы для многих инфекционных патогенов, предложены разнообразные системы пробоподготовки: от применения термического лизиса в качестве экстракции НК до обработки магнитными частицами с целью накопления искомого материала и повышения чувствительности теста [25–27]. По данным публикаций, метод SHERLOCK обладает более высокой чувствительностью по сравнению с аналогичными подходами, предел обнаружения сочетанного использования технологий РПА и CRISPR–Cas составляет несколько копий мишени в реакции [24, 28].
Альтернативные методы детекции результатов РПА. Обнаружение методом флокуляции основано на использовании особых полимеров, которые способствуют связыванию ампликонов, полученных в результате РПА, и магнитных частиц с образованием агрегатов, выпадающих в виде хлопьев, легко различимых невооруженным глазом. Для образования хлопьев важными условиями являются длина НК более 100 нуклеотидов и определённый pH буфера. РПА в сочетании с полимерной флокуляционной седиментацией была предложена J. Hu и соавт. для выявления Staphylococcus aureus, чувствительность составила 38 КОЕ/мл [29]. Данный способ относится к детекции по конечной точке, не требует специального оборудования, занимает 10 мин. Однако необходимость выбирать ампликоны длиннее, чем обычно, может негативно отразиться на эффективности амплификации.
В качестве методов детекции результатов РПА также применяют хемилюминесценцию, измерение силы тока в растворе, фотонную детекцию на основе кремниевого микрокольцевого резонатора [30, 31]. На основе электрохимической детекции для обнаружения ДНК HPV16 разработан биосенсор с высокой специфичностью и чувствительностью 0,23 копии/мкл [30]. Поверхностно-усиленная Рамановская спектроскопия позволяет при использовании нескольких репортёрных молекул проводить одномоментный анализ до 5 мишеней [32]. Поскольку данные методы детекции требуют специального оборудования, они не находят широкого применения в практике.
Влияние температуры и перемешивания
В руководстве по РПА рекомендуется температура реакции 37–42ºC [8]. Y. Yang и соавт. протестировали широкий диапазон температур — 15–50ºC [33]. Установлено, что для успешной амплификации минимальная температура реакции должна быть выше 30ºC. В некоторых источниках сообщается о положительном результате РПА при 25ºC [34, 35]. Такая относительно низкая температура позволяет отказаться даже от термостата и использовать для поддержания температуры другие источники, например грелку для рук. Z.A. Crannell и соавт. проверяли возможность поддержания постоянной температуры РПА путём инкубирования пробирок на различных участках тела человека [36]. Отмечены высокая чувствительность и стабильность реакции при зажимании пробирок в кулаке.
Развитие диагностики «у постели больного» или возможность применения РПА для самотестирования подразумевает разработку устройства, удобного для применения, с минимальным количеством компонентов и простой техникой проведения. В рамках данного направления несколькими группами учёных [37, 38] были разработаны браслеты на предплечье, в которые вставляется чип или колонки, где проходит реакция. Для детекции предлагают устройство, подключаемое к смартфону. Использование таких браслетов позволяет также с лёгкостью перемешивать реакционную смесь, что значительно влияет на эффективность амплификации.
Буфер, в котором протекает РПА, отличается повышенной вязкостью по сравнению с ПЦР. Многими исследователями показано, что при перемешивании в ходе РПА повышается эффективность амплификации, причем наилучшие результаты получены при постоянном автоматизированном перемешивании, а необходимым минимумом считается дополнительное встряхивание через 4–5 мин после начала реакции. В работе L. Wambua и соавт. без перемешивания положительный флуоресцентный сигнал был получен через 8–14 мин, а при условии перемешивания — в среднем через 5–8 мин [39].
L. Lillis и соавт. продемонстрировали, что уменьшение реакционного объёма с 50 до 5 мкл может компенсировать эффект перемешивания [40], поскольку меньший объём увеличивает взаимодействие между реагентами и олигонуклеотидами, необходимыми для амплификации. Уменьшение объёма реакционной смеси также снижает себестоимость реакции.
Влияние ингибиторов
В ряде исследований была показана высокая устойчивость РПА к присутствию ингибиторов. При проведении термического лизиса реакцию можно проводить без полноценного выделения НК из таких образцов, как сыворотка, кал, моча, плевральная жидкость, молоко, почва [17, 27, 41–44]. Продемонстрирована устойчивость к наличию гемоглобина, этанола и гепарина [45]. Тем не менее РПА может подавляться высокими концентрациями геномной ДНК в образцах цельной крови (20–100 нг/мкл). При анализе нативных образцов мочи замечено также снижение чувствительности РПА при низких концентрациях целевой ДНК и внесении мочи в объёме более 5% от общего объёма реакционной смеси [46].
Надёжность РПА в присутствии традиционных ингибиторов позволяет исключить предварительную обработку образца, упрощает анализ и снижает затраты [19]. Стоит отметить, что в некоторых работах наблюдалось снижение чувствительности РПА при прямом исследовании образца [2, 44].
Количественная РПА
Проведение количественного анализа необходимо для оценки уровня экспрессии генов, определения количества вирусной РНК/ДНК в крови с целью прогноза течения заболевания и эффективности лечения. Способ количественной РПА был предложен для выявления Vibrio mimicus, вирусов иммунодефицита человека и денге [21, 47, 48]. Концентрация ДНК экспоненциально коррелировала с подъёмом кривых флуоресценции в РПА, в экспериментах удалось определить концентрацию опытных образцов с незначительной погрешностью.
В отличие от классической ПЦР в реальном времени, в РПА сложно точно контролировать начальную точку реакции, поскольку РПА инициируется путём добавления ацетата магния, а не повышением температуры реакции. Кроме того, ПЦР синхронизируется в течение каждого цикла, тогда как в РПА отжиг и элонгация происходят параллельно при оптимальной температуре. По этим причинам РПА в реальном времени не считается надёжным средством определения концентрации НК.
Количественный анализ НК возможен с помощью цифровой РПА [49]. Данный метод предполагает фракционирование образца и распределение его в разных разведениях на отсеки объёмом от микро- до пиколитров, в каждом из которых впоследствии отдельно происходит амплификация НК. В качестве метода детекции обычно используют флуоресцентные зонды. В конце амплификации подсчёт положительных и отрицательных результатов разных разведений даёт точную, абсолютную количественную оценку исходного количества матрицы нуклеиновой кислоты на основе статистики Пуассона.
Часто цифровую РПА реализуют в формате микрочипов [50–52]. Для одномоментного запуска реакции во всех камерах (лунках) применяются различные подходы. Так, в работе F. Shen и соавт. применили чип, состоящий из 2 планшетов, скользящих относительно друг друга, содержащих различные реагенты для РПА, реакция запускалась при сопоставлении планшетов в определённом положении [50]. В другом микрочипе, состоящем из массива 27 000 пиколитровых лунок (PWA), загрузка образца осуществлялась скребковым лезвием [51]. J.Q. Cui и соавт. разработали технологию PADLOCK (Picoinjection Aided Digital reaction unLOCKing), в которой пикоинъектором в отдельные капли реакционной смеси вносили ацетат магния [52]. В сочетании с детекцией на основе CRISPR/Cas13a методом цифровой капельной РПА авторам удалось обнаруживать отдельные копии НК за 30 мин.
Мультиплексная РПА
Возможность параллельного тестирования нескольких генетических маркеров одновременно предпочтительнее с точки зрения себестоимости и времени анализа. Мультиплексирование удобно при проведении дифференциальной диагностики заболеваний со сходными клиническими симптомами и эпидемиологическим анамнезом.
J. Zhai и соавт. описали способ обнаружения Chlamydia trachomatis и Neisseria gonorrhoeae методом РПА с иммунохроматографической детекцией на тест-полосках [53]. Аналитическая чувствительность составила 200 копий на реакцию. Мультиплексную РПА в одной пробирке с электрофоретической детекцией результатов применили для выявления разных штаммов вируса папилломы человека. По сравнению с ПЦР метод показал 100% специфичность, 78% диагностическую точность, 75% чувствительность, но был намного быстрее и дешевле, поэтому авторы предложили использовать его в экспресс-диагностике [54].
В тесте для выявления новой коронавирусной инфекции на основе мультиплексной РПА проводится одновременное обнаружение двух консервативных мишеней SARS-CoV-2 — генов E и RdRP, что позволяет получать более точные результаты анализа. При флуоресцентной детекции аналитическая чувствительность теста составила 9,5 и 17 копий РНК на реакцию для генов Е и RdRP соответственно. При использовании тест-полосок аналитическая чувствительность метода соответствовала 130 копиям РНК на реакцию [4].
Мультиплексная РПА также была предложена для обнаружения бактерий [55, 22], вирусов [56], простейших [57]. Наибольший уровень мультиплексности представлен в работе J. Song и соавт., которые предложили объединить реакции РПА и LAMP на микрочипе. Вначале на этапе РПА проходила одновременная амплификация 16 различных ДНК-мишеней, затем реакционная смесь распределялась между 16 камерами, в которых проходили LAMP и флуоресцентная детекция результатов по отдельности для каждого возбудителя. Такая двухэтапная изотермическая амплификация РПА-LAMP в 16-плексном формате позволила обнаружить до 1 БОЕ штамма вируса Зика [58].
Другим подходом к мультиплексированию является проведение параллельного анализа. Например, разработан подход на основе микрочипа, в котором сначала проводится РПА в отдельных пробирках, а затем продукты амплификации переносят в микрочип, лунки которого покрыты частицами с иммобилизованными квантовыми точками. Удалось добиться одновременной детекции ВИЧ и вируса гепатита B с пределом обнаружения 1000 копий вируса на 1 мл [59].
Для диагностики лихорадки Эбола разработано бумажное микрофлюидное устройство (micro-Paper Analytical Devices — µPADs), на котором в каплях воска были лиофилизированы компоненты, необходимые для проведения РПА [5]. Для апробации «в полевых условиях» все реактивы помещали в небольшой чемодан, для детекции использовали портативную фотокамеру и 2 светодиода. Далее авторы модифицировали µPADs, добавив новые точки с лиофилизированными на них праймерами ещё к 2 мишеням. При сопоставлении с ПЦР чувствительность технологии µPADs составила 90%.
В целом проведение мультиплексной РПА в параллельном формате имеет более высокую производительность анализа, позволяет избежать димеров между олигонуклеотидами и анализировать большее число мишеней по сравнению с постановкой РПА в одной пробирке. Степень мультиплексирования ограничена количеством доступных уникальных меток, используемых для детекции разных мишеней, и размерами реакционной поверхности. Однако в данном случае экономится только время реакции при той же себестоимости, а в некоторых случаях возникает риск контаминации.
Автономные портативные устройства
В последние годы значительное внимание уделяется разработке автономных миниатюрных устройств (микрочипов) для проведения молекулярно-генетических исследований. РПА — наиболее удобный для этой цели метод, поскольку реакция протекает быстро и при относительно невысокой температуре. Цель разработки микрочипа — интегрировать процессы подготовки пробы, амплификации и детекции в одном устройстве, обычно за счёт микрофлюидики.
D. Liu и соавт. для диагностики новой коронавирусной инфекции разработали микрочип на основе микрофлюидных технологий [60], который объединил РПА с обратной транскрипцией (ОТ-РПА) и систему детекции на основе иммунохроматографических тест-полосок. Разработанная система показала предел обнаружения, равный 1 копии/мкл или 30 копиям на образец, при проверке клинических образцов диагностическая чувствительность составила 97%, а специфичность — 100%, что сопоставимо с характеристиками ОТ-ПЦР. При решении аналогичной задачи другими авторами было создано устройство в форме ручки, совмещающее модули амплификации, иммунохроматографической детекции и герметизации [61].
Для обнаружения SARS-CoV-2 предложен чип на основе центробежной микрофлюидики, сочетающий ОТ-РПА и флуоресцентную детекцию по конечной точке [62]. Микрофлюидный чип в форме предметного стекла для микроскопа позволял одновременно обнаруживать наличие 3 вирусных мишеней и 1 мишень ДНК человека за 30 мин. Чувствительность составила 40 копий РНК на реакцию для гена E, 20 копий — для гена N, 10 копий — для гена ORF1ab. Альтернативный вариант микрочипа, объединяющий все этапы анализа, основан на очистке образца с помощью магнитных частиц и РПА с флуоресцентной детекцией. Его применение обеспечивало выявление РНК SARS-CoV-2 за 37 мин с чувствительностью 10 копий/мл [63].
В целом разработка интегрированных систем анализа на основе РПА, позволяющих исследовать образец без дополнительных этапов пробоподготовки до этапа получения результата, направлена на обеспечение портативности данного метода, проведения диагностики «у постели больного» и даже для самотестирования. Однако ускоренные методы экстракции НК не подходят для некоторых видов биологического материала, например цельной крови или секционного материала.
Заключение
Идеальный метод выявления возбудителей инфекционных заболеваний должен быть специфичным, чувствительным, быстрым и экономически выгодным. Оценивать чувствительность и специфичность РПА в целом довольно трудно, поскольку данные параметры сильно зависят от выбора конкретных праймеров и способа детекции результатов. Не существует надежных критериев и программного обеспечения, гарантирующих высокую эффективность РПА, что затрудняет разработку новых тест-систем. Специфичность РПА теоретически может быть ниже, чем у ПЦР, за счёт устойчивости рекомбиназы к однонуклеотидным заменам, но большая длина праймеров в РПА частично нивелирует этот недостаток. Чувствительность РПА сопоставима с ПЦР и достигает нескольких копий ДНК/РНК на реакцию, однако может превышать 100 копий на реакцию.
РПА отличается самой высокой скоростью получения результата. При условии перемешивания реакционной смеси и флуоресцентной детекции положительные результаты регистрируют через 5–10 мин. Изотермичность реакции в сочетании с температурным оптимумом в 37–40ºC позволяют не только отказаться от термоциклеров, но и использовать для инкубации тепло человеческого тела, что может быть актуальным для диагностики в «полевых» условиях. Несомненным достоинством метода является устойчивость РПА к ряду ингибиторов, которая дает возможность исключить этап выделения НК при исследовании большинства типов образцов, что снижает время проведения и общую стоимость анализа. С другой стороны, себестоимость реакционной смеси РПА выше, чем LAMP и ПЦР, за счёт большего числа ферментов и компонентов реакции.
На сегодняшний день технология РПА, в отличие от LAMP, защищена патентом, что препятствует ее широкому внедрению в лабораторную диагностику. Наиболее перспективными направлениями развития данного метода, на наш взгляд, является сочетание РПА с детекцией на основе CRISPR–Cas, обеспечивающее высокую чувствительность, а также разработка портативных устройств, объединяющих все этапы анализа и подходящих для экспресс-диагностики.
About the authors
Olga S. Bondareva
Volgograd Plague Control Research Institute
Author for correspondence.
Email: bondareva0s@mail.ru
ORCID iD: 0000-0001-5690-6686
Cand. Sci. (Med.), senior researcher, Laboratory of gene diagnostics of particularly dangerous infections
Russian Federation, VolgogradArtem A. Baturin
Volgograd Plague Control Research Institute
Email: bondareva0s@mail.ru
ORCID iD: 0000-0001-9510-7246
researcher, Laboratory of gene diagnostics of particularly dangerous infections
Russian Federation, VolgogradAnna V. Mironova
Volgograd Plague Control Research Institute
Email: bondareva0s@mail.ru
ORCID iD: 0000-0002-6958-7861
researcher, Laboratory of gene diagnostics of particularly dangerous infections
Russian Federation, VolgogradReferences
- Piepenburg O., Williams C.H., Stemple D.L., Armes N.A. DNA detection using recombination proteins. PLoS Biol. 2006;4(7):204. doi: https://doi.org/10.1371/journal.pbio.0040204
- Li J., Macdonald J., von Stetten F. Review: a comprehensive summary of a decade development of the recombinase polymerase amplification. Analyst. 2018;144(1):31–67. doi: https://doi.org/10.1039/C8AN01621F
- Juma K.M., Takita T., Ito K., et al. Optimization of reaction condition of recombinase polymerase amplification to detect SARS-CoV-2 DNA and RNA using a statistical method. Biochem. Biophys. Res. Commun. 2021;567:195–200. doi: https://doi.org/10.1016/j.bbrc.2021.06.023
- Cherkaoui D., Huang D., Miller B.S., et al. Harnessing recombinase polymerase amplification for rapid multi-gene detection of SARS-CoV-2 in resource-limited settings. Biosens. Bioelectron. 2021;189:113328. doi: https://doi.org/10.1101/2021.02.17.21251732
- Magro L., Jacquelin B., Escadafal C., et al. Paper-based RNA detection and multiplexed analysis for Ebola virus diagnostics. Sci. Rep. 2017;7(1):1347. doi: https://doi.org/10.1038/s41598-017-00758-9
- Zhang X., Guo L., Ma R., et al. Rapid detection of Salmonella with recombinase aided amplification. J. Microbiol. Methods. 2017;139:202–4. doi: https://doi.org/10.1016/j.mimet.2017.06.011
- Chen W., Fan J., Li Z., et al. Development of recombinase aided amplification combined with disposable nucleic acid test strip for rapid detection of porcine circovirus type 2. Front. Vet. Sci. 2021;8:676294. doi: https://doi.org/10.3389/fvets.2021.676294
- TwistAmp® DNA Amplification Kits: Combined Instruction Manual. URL: https://www.twistdx.co.uk/wp-content/uploads/2021/04/ta01cmanual-combined-manual_revo_v1-3b.pdf
- Salazar A., Ochoa-Corona F.M., Talley J.L., et al. Recombinase polymerase amplification (RPA) with lateral flow detection for three Anaplasma species of importance to livestock health. Sci. Rep. 2021;11:15962. doi: https://doi.org/10.1038/s41598-021-95402-y
- Fuller S.L., Savory E.A., Weisberg A.J., et al. Isothermal amplification and lateral-flow assay for detecting crown-gall-causing Agrobacterium spp. Phytopathology. 2017;107(9):1062–8. doi: https://doi.org/10.1094/PHYTO-04-17-0144-R
- Yamanaka E.S., Tortajada-Genaro L.A., Maquieira Á. Low-cost genotyping method based on allele-specific recombinase polymerase amplification and colorimetric microarray detection. Microchim. Acta. 2017;184:1453–62. doi: https://doi.org/10.1007/s00604-017-2144-0
- Sharma N., Hoshika S., Hutter D., et al. Recombinase-based isothermal amplification of nucleic acids with self-avoiding molecular recognition systems (SAMRS). Chembiochem. 2014;15(15):2268–74. doi: https://doi.org/10.1002/cbic.201402250
- Higgins M., Ravenhall M., Ward D., et al. PrimedRPA: primer design for recombinase polymerase amplification assays. Bioinformatics. 2019;35(4):682–4. doi: https://doi.org/10.1093/bioinformatics/bty701
- Li J.S., Hao Y.Z., Hou M.L., et al. Development of a recombinase-aided amplification combined with lateral flow dipstick assay for the rapid detection of the African swine fever virus. Biomed. Environ. Sci. 2022;35(2):133–40. doi: https://doi.org/10.3967/bes2022.018
- Patchsung M., Jantarug K., Pattama A., et al. Clinical validation of a Cas13-based assay for the detection of SARS-CoV-2 RNA. Nat. Biomed. Eng. 2020;4(12):1140–9. doi: https://doi.org/10.1038/s41551-020-00603-x
- Naveen K.P., Bhat A.I. Development of reverse transcription loop-mediated isothermal amplification (RT-LAMP) and reverse transcription recombinase polymerase amplification (RT-RPA) assays for the detection of two novel viruses infecting ginger. J. Virol. Methods. 2020;282:113884. doi: https://doi.org/10.1016/j.jviromet.2020.113884
- Shen X.X., Qiu F.Z., Shen L.P., et al. A rapid and sensitive recombinase aided amplification assay to detect hepatitis B virus without DNA extraction. BMC Infect. Dis. 2019;19(1):229. DOI: https://doi.org/10.1186/s12879-019-3814-9
- Patel P., Abd El Wahed A., Faye O., et al. A field-deployable reverse transcription recombinase polymerase amplification assay for rapid detection of the Chikungunya virus. PLoS Negl. Trop. Dis. 2016;10(9):e0004953. doi: https://doi.org/10.1371/journal.pntd.0004953
- Londono M.A., Harmon C.L., Polston J.E. Evaluation of recombinase polymerase amplification for detection of begomoviruses by plant diagnostic clinics. Virol. J. 2016;13:48. DOI: https://doi.org/10.1186/s12985-016-0504-8
- Babu B., Washburn B.K., Miller S.H., et al. A rapid assay for detection of Rose rosette virus using reverse transcription-recombinase polymerase amplification using multiple gene targets. J. Virol. Methods. 2017;240:78–84. doi: https://doi.org/10.1016/j.jviromet.2016.11.014
- Crannell Z.A., Rohrman B., Richards-Kortum R. Quantification of HIV-1 DNA using real-time recombinase polymerase amplification. Anal. Chem. 2014;86(12):5615–9. doi: https://doi.org/10.1021/ac5011298
- Conrad C.C., Daher R.K., Stanford K., et al. Sensitive and accurate recombinase polymerase amplification assay for detection of the primary bacterial pathogens causing bovine respiratory disease. Front. Vet. Sci. 2020;7:208. doi: https://doi.org/10.3389/fvets.2020.00208
- Guo M., Feng P., Zhang L., et al. Rapid detection of Clostridium tetani by recombinase polymerase amplification using an exo probe. J. Microbiol. Biotechnol. 2022;32(1):91–8. doi: https://doi.org/10.4014/jmb.2109.09022
- Zahra A., Shahid A., Shamim A., et al. The SHERLOCK platform: an insight into advances in viral disease diagnosis. Mol. Biotechnol. 2023;65(5):699–714. doi: https://doi.org/10.1007/s12033-022-00625-7
- Arizti-Sanz J., Bradley A., Zhang Y.B., et al. Simplified Cas13-based assays for the fast identification of SARS-CoV-2 and its variants. Nat. Biomed. Eng. 2022;6(8):932–43. doi: https://doi.org/10.1038/s41551-022-00889-z
- Joung J., Ladha A., Saito M., et al. Detection of SARS-CoV-2 with SHERLOCK one-pot testing. N. Engl. J. Med. 2020;383(15):1492–4. doi: https://doi.org/10.1056/NEJMc2026172
- Zhang J.X., Xu J.H., Yuan B., et al. Detection of Burkholderia pseudomallei with CRISPR-Cas12a based on specific sequence tags. Front. Public Health. 2023;11:1153352. doi: https://doi.org/10.3389/fpubh.2023.1153352
- Волков А.А., Долгова А.С., Дедков В.Г. Молекулярные диагностические платформы, созданные на базе систем CRISPR/Cas. Инфекция и иммунитет. 2022;12(1):9–20. Volkov A.A., Dolgova A.S., Dedkov V.G. CRISPR/Cas-based diagnostic platforms. Russian Journal of Infection and Immunity. 2022;12(1):9–20. DOI: https://doi.org/10.15789/2220-7619-CCB-1843. EDN: https://elibrary.ru/fdhjgz
- Hu J., Wang Y., Ding H., et al. Recombinase polymerase amplification with polymer flocculation sedimentation for rapid detection of Staphylococcus aureus in food samples. Int. J. Food. Microbiol. 2020;331:108691. doi: https://doi.org/10.1016/j.ijfoodmicro.2020.108691
- Nakowong P., Chatchawal P., Chaibun T. Detection of high-risk HPV 16 genotypes in cervical cancers using isothermal DNA amplification with electrochemical genosensor. Talanta. 2024;269:125495. doi: https://doi.org/10.1016/j.talanta.2023.125495
- Dao T.N.T., Lee E.Y., Koo B., et al. A microfluidic enrichment platform with a recombinase polymerase amplification sensor for pathogen diagnosis. Anal. Biochem. 2018;544:87–92. doi: https://doi.org/10.1016/j.ab.2017.12.030
- Zhuang J., Zhao Z., Lian K., et al. SERS-based CRISPR/Cas assay on microfluidic paper analytical devices for supersensitive detection of pathogenic bacteria in foods. Biosens. Bioelectron. 2022;207:114167. doi: https://doi.org/10.1016/j.bios.2022.114167
- Yang Y., Qin X., Wang G., et al. Development of an isothermoal amplification-based assay for rapid visual detection of an Orf virus. Virol. J. 2016;13:46. doi: https://doi.org/10.1186/s12985-016-0502-x
- Poulton K., Webster B. Development of a lateral flow recombinase polymerase assay for the diagnosis of Schistosoma mansoni infections. Anal. Biochem. 2018;546:65–71. doi: https://doi.org/10.1016/j.ab.2018.01.031
- Wang F., Liang Q., Lv R., et al. Optimization and validation of reverse transcription recombinase-aided amplification (RT-RAA) for Sorghum mosaic virus detection in sugarcane. Pathogens. 2023;12(8):1055. doi: https://doi.org/10.3390/pathogens12081055
- Crannell Z.A., Rohrman B., Richards-Kortum R. Equipment-free incubation of recombinase polymerase amplification reactions using body heat. PLoS One. 2014;9(11):e112146. DOI: https://doi.org/10.1371/journal.pone.0112146
- Kong M., Zihan L.I., Wu J., et al. A wearable microfluidic device for rapid detection of HIV-1 DNA using recombinase polymerase amplification. Talanta. 2019;205:120155. doi: https://doi.org/10.1016/j.talanta.2019.120155
- Trinh K.T.L., Lee N.Y. Fabrication of wearable PDMS device for rapid detection of nucleic acids via recombinase polymerase amplification operated by human body heat. Biosensors (Basel). 2022;12(2):72. DOI: https://doi.org/10.3390/bios12020072
- Wambua L., Schneider B., Okwaro A., et al. Development of field-applicable tests for rapid and sensitive detection of Cadidatus Phytoplasma oryzae. Mol. Cell. Probes. 2017;35:44–56. doi: https://doi.org/10.1016/j.mcp.2017.06.004
- Lillis L., Siverson J., Lee A., et al. Factors influencing Recombinase polymerase amplification (RPA) assay outcomes at point of care. Mol. Cell Probes. 2016;30(2):74–8. doi: https://doi.org/10.1016/j.mcp.2016.01.009
- Liu Y., Xiang J., Gao Y., et al. Rapid detection of Cryptosporidium spp. in diarrheic cattle feces by isothermal recombinase polymerase amplification assays. Heliyon. 2023;9(10):e20794. DOI: https://doi.org/10.1016/j.heliyon.2023.e20794
- Balea R., Pollak N.M., Hobson-Peters J., et al. Development and pre-clinical evaluation of a Zika virus diagnostic for low resource settings. Front. Microbiol. 2023;14:1214148. doi: https://doi.org/10.3389/fmicb.2023.1214148
- Choi G., Jung J.H., Park B.H., et al. A centrifugal direct recombinase polymerase amplification (direct-RPA) microdevice for multiplex and real-time identification of food poisoning bacteria. Lab. Chip. 2016;16(12):2309–16. doi: https://doi.org/10.1039/C6LC00329J
- Wu Y.D., Zhou D.H., Zhang L.X., et al. Recombinase polymerase amplification (RPA) combined with lateral flow (LF) strip for equipment-free detection of Cryptosporidium spp. oocysts in dairy cattle feces. Parasitol. Res. 2016;115(9):3551–5. DOI: https://doi.org/10.1007/s00436-016-5120-4
- Kersting S., Rausch V., Bier F.F., von Nickisch-Rosenegk M. Rapid detection of Plasmodium falciparum with isothermal recombinase polymerase amplification and lateral flow analysis. Malar J. 2014;13:99. doi: https://doi.org/10.1186/1475-2875-13-99
- Rosser A., Rollinson D., Forrest M., Webster B.L. Isothermal Recombinase Polymerase amplification (RPA) of Schistosoma haematobium DNA and oligochromatographic lateral flow detection. Parasit. Vector. 2015;8:446. doi: https://doi.org/10.1186/s13071-015-1055-3
- Zhu P., Huang Z., Xiong Z., et al. Development and evaluation of real-time recombinase polymerase amplification assay for rapid and sensitive detection of Vibro mimicus in human plasma samples. J. Appl. Microbiol. 2022;133(3):1650–9. doi: https://doi.org/10.1111/jam.15666
- Valloly P., Roy R. Nucleic acid quantification with amplicon yield in recombinase polymerase amplification. Anal. Chem. 2022;94(40):13897–905. doi: https://doi.org/10.1021/acs.analchem.2c02810
- Choi J.W., Seo W.H., Kang T., et al. Droplet digital recombinase polymerase amplification for multiplexed detection of human coronavirus. Lab. Chip. 2023;23(10):2389–98. doi: https://doi.org/10.1039/d3lc00025g
- Shen F., Davydova E.K., Du W., et al. Digital isothermal quantification of nucleic acids via simultaneous chemical initiation of recombinase polymerase amplification reactions on SlipChip. Anal. Chem. 2011;83(9):3533–40. doi: https://doi.org/10.1021/ac200247e
- Li Z., Liu Y., Wei Q., et al. Picoliter well array chip-based digital recombinase polymerase amplification for absolute quantification of nucleic acids. PLoS ONE. 2016;11(4):e0153359. DOI: https://doi.org/10.1371/journal.pone.0153359
- Cui J.Q., Liu F.X., Park H., et al. Droplet digital recombinase polymerase amplification (ddRPA) reaction unlocking via picoinjection. Biosens. Bioelectron. 2022;202:114019. doi: https://doi.org/10.1016/j.bios.2022.114019
- Zhai J., Wang L., Qiao X., et al. Detection of Neisseria gonorrhoeae and Chlamydia trachomatis infections in pregnant women by multiplex recombinase polymerase amplification. PLoS One. 2021;16(5):e0251119. doi: https://doi.org/10.1371/journal.pone.0251119
- Wongsamart R., Bhattarakasol P., Chaiwongkot A., et al. Multiplex recombinase polymerase amplification for high-risk and low-risk type HPV detection, as potential local use in single tube. Sci. Rep. 2023;13(1):829. doi: https://doi.org/10.1038/s41598-023-28038-9
- Лапа С.А., Суржиков С.А., Благодатских С.А. и др. Рекомбиназная полимеразная амплификация для быстрого выявления возбудителей бактериальной пневмонии человека. Молекулярная биология. 2023;57(3):539–45. Lapa S.A., Surzhikov S.A., Blagodatskikh S.A., et al. Recombinase polymerase amplification for rapid detection of human bacterial pneumonia pathogens. Molecular Biology. 2023;57(3):539–45. DOI: https://doi.org/10.31857/S0026898423030072 EDN: https://elibrary.ru/chgyqf
- Ivanov A.V., Safenkova I.V., Zherdev A.V., Dzantiev B.B. Multiplex assay of viruses integrating recombinase polymerase amplification, barcode — anti-barcode pairs, blocking anti-primers, and lateral flow assay. Anal. Chem. 2021;93(40):13641–50. DOI: https://doi.org/10.1021/acs.analchem.1c03030
- Crannell Z., Castellanos-Gonzalez A., Nair G., et al. Multiplexed recombinase polymerase amplification assay to detect intestinal protozoa. Anal. Chem. 2016;88(3):1610–6. doi: https://doi.org/10.1021/acs.analchem.5b03267
- Song J., Liu C., Mauk M.G., et al. Two-stage isothermal enzymatic amplification for concurrent multiplex molecular detection. Clin. Chem. 2017;63(3):714–22. doi: https://doi.org/10.1373/clinchem.2016.263665
- Ming K., Kim J., Biondi M.J., et al. Integrated quantum dot barcode smartphone optical device for wireless multiplexed diagnosis of infected patients. ACS Nano. 2015;9(3):3060–74. DOI: https://doi.org/10.1021/nn5072792
- Liu D., Shen H., Zhang Y., et al. A microfluidic-integrated lateral flow recombinase polymerase amplification (MI-IF-RPA) assay for rapid COVID-19 detection. Lab. Chip. 2021;21(10):2019–26. DOI: https://doi.org/10.1039/D0LC01222J
- Fu Q., Tu Y., Cheng L., et al. A fully-enclosed prototype 'pen' for rapid detection of SARS-CoV-2 based on RT-RPA with dipstick assay at point-of-care testing. Sens. Actuators B Chem. 2023;383:133531. doi: https://doi.org/10.1016/j.snb.2023.133531
- Li R., Su N., Ren X., et al. Centrifugal microfluidic-based multiplex recombinase polymerase amplification assay for rapid detection of SARS-CoV-2. iScience. 2023;26(3):106245. DOI: https://doi.org/10.1016/j.isci.2023.106245
- Seder I., Coronel-Tellez R., Helalat S.H., Sun Y. Fully integrated sample-in-answer-out platform for viral detection using digital reverse transcription recombinase polymerase amplification (dRT-RPA). Biosens. Bioelectron. 2023;237:115487. doi: https://doi.org/10.1016/j.bios.2023.115487
Supplementary files