Характеристика молекулярно-генетических свойств эпидемических штаммов Klebsiella pneumoniae и Staphylococcus aureus — возбудителей инфекций, связанных с оказанием медицинской помощи, циркулирующих на территории Нижегородской области
- Авторы: Соловьева И.В.1, Точилина А.Г.1, Белова И.В.1, Зайцева Н.Н.1, Кучеренко Н.С.2, Садыкова Н.А.2, Молодцова С.Б.1, Кропотов В.С.1
-
Учреждения:
- Нижегородский научно-исследовательский институт эпидемиологии и микробиологии имени академика И.Н. Блохиной Роспотребнадзора
- Управление Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека по Нижегородской области
- Выпуск: Том 102, № 5 (2025)
- Страницы: 560-570
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/18975
- DOI: https://doi.org/10.36233/0372-9311-678
- EDN: https://elibrary.ru/LSVTWH
- ID: 18975
Цитировать
Полный текст
Аннотация
Введение. Молекулярно-эпидемиологический мониторинг направлен на получение актуальной информации о генетических вариантах возбудителей инфекций, связанных с оказанием медицинской помощи (ИСМП), циркулирующих в регионе. В настоящее время особое внимание уделяется слежению за представителями группы ESKAPE, т. к. они являются частой причиной ИСМП, осложняют течение основного заболевания и становятся всё более серьёзной угрозой здоровью и жизни пациентов, поскольку обладают комплексом генов патогенности и разнообразными механизмами антибиотикорезистентности.
Цель работы — анализ результатов полногеномного секвенирования эпидемических штаммов возбудителей ИСМП — Klebsiella pneumoniae ssp. pneumoniae и Staphylococcus aureus, циркулирующих на территории Нижегородской области.
Материалы и методы. Использованы классические бактериологические методы, MALDI TOF масс-спектрометрия, полногеномное секвенирование, биоинформатические методы.
Результаты. Углублённый анализ показал циркуляцию в отделении новорождённых популяции классических штаммов K. pneumoniae сиквенс-типа (ST) 3-К-типа (К) 3, содержащих ряд генов патогенности и бета-лактамазу blaSHV-1. В многопрофильном стационаре обнаружена циркуляция популяции штаммов конвергентного патотипа K. pneumoniae ST395-К39, а также выявлены штаммы конвергентного патотипа K. pneumoniae ST395-К2, К47 и штаммы классического патотипа K. pneumoniae ST5209-К35, ST441-К62, ST147-К64, содержащие в геноме спектр генов патогенности и бета-лактамаз, в том числе Нью-Дели металло-бета-лактамазу blaNDM-1. Штаммы S. aureus, связанные с катетер-ассоциированными инфекциями кровотока, обладают выраженным патогенным потенциалом, относятся к 13 различным ST и 19 spa-типам (t). В гемодиализных центрах и отделениях гемодиализа выявлена циркуляция штаммов метициллин-резистентных (SCCmec IV, ST8, t008) и метициллин-чувствительных (ST1, t127) стафилококков.
Заключение. Полученные данные свидетельствуют о циркуляции в лечебно-профилактических медицинских организациях конвергентных и классических штаммов K. pneumoniae и вирулентных штаммов S. aureus, что обосновывает необходимость молекулярно-эпидемиологического мониторинга.
Полный текст
Введение
Молекулярно-эпидемиологический мониторинг становится одной из неотъемлемых задач организации системы эпидемиологического надзора за инфекционными заболеваниями, т. к. позволяет отслеживать циркуляцию условно-патогенных микроорганизмов и своевременно выявлять признаки возможных массовых случаев инфекций, к которым относятся: выделение однородного спектра микроорганизмов у обследуемых лиц; увеличение частоты возникновения случаев инфекционных заболеваний, обусловленных одним видом или группой видов возбудителей, увеличение частоты обнаружения госпитальных штаммов. Его задачами является слежение за популяционной структурой возбудителей инфекций, в том числе инфекций, связанных с оказанием медицинской помощи (ИСМП), MLST-типирование штаммов, анализ генов патогенности, детерминант антибиотикорезистентности, обнаружение новых вариантов госпитальных штаммов, наблюдение за их изменчивостью с целью оценки прогнозирования эпидемиологической ситуации и обоснования своевременного вмешательства в ход эпидемического процесса [1].
ESKAPE-патогены являются частой причиной ИСМП, осложняют течение основного заболевания и представляют собой серьёзную угрозу здоровью и жизни пациентов, т. к. способны быстро адаптироваться и находить новые способы сопротивления действию лекарственных средств, дезинфицирующих средств и антисептиков, а также на генетическом уровне передавать эту способность другим патогенам [2, 3].
Среди бактерий, ставших причиной госпитальных инфекций, Klebsiella pneumoniae и Staphylococcus aureus занимают лидирующее положение, причём наблюдается рост доли устойчивых к карбапенемам K. pneumoniae и широкое распространение S. aureus, принадлежащих к группе метициллин-резистентных стафилококков (MRSA), способных вызывать вспышечную заболеваемость и обусловливать катетер-ассоциированные инфекции кровотока (КАИК) [4, 5].
Цель исследования — анализ результатов полногеномного секвенирования эпидемических штаммов возбудителей ИСМП — K. pneumoniae ssp. pneumoniae и S. aureus, циркулирующих на территории Нижегородской области.
Материалы и методы
Исследуемые штаммы
Изучены 55 эпидемических штаммов возбудителей: 17 штаммов K. pneumoniae ssp. pneumoniae и 38 штаммов S. aureus. По месту выделения штаммы были разделены на три группы:
- 1-ю группу образовали 7 штаммов pneumoniae, выделенных в отделении новорождённых педиатрического стационара от больных детей (желудочное содержимое) (n = 4), с оборудования и предметов ухода (смывы со шланга для отсоса, шприца для кормления) (n = 3);
- 2-ю группу — 10 штаммов pneumoniae, выделенных в отделениях многопрофильного стационара от больных (отделяемое ран) (n = 9) и из внешней среды отделения (смыв с крана раковины отделения реанимации) (n = 1);
- 3-ю группу — 38 штаммов аureus, из них 31 штамм — от пациентов с КАИК, получавших амбулаторное лечение в гемодиализных центрах города и области и проходивших стационарное лечение в лечебно-профилактических медицинских организациях (МО) города (кровь, рана в области катетера, перитонеальная жидкость, назальный мазок); 3 штамма, выделенные от медперсонала (назальный мазок), и 4 — из внешней среды МО (смывы с оборудования).
Исследование проводилось при добровольном информированном согласии пациентов или их законных представителей. Протокол исследования одобрен Этическим комитетом № 1 по проведению научных исследований с участием человека в качестве объекта исследования Приволжского исследовательского медицинского университета (протокол № 7 от 05.07.2018).
Культивирование и идентификация бактерий
Выделение штаммов условно-патогенных микроорганизмов осуществляли с использованием классического бактериологического метода, идентификацию проводили методом MALDI TOF масс-спектрометрии с использованием масс-спектрометра «Autoflex» («Bruker Daltonics»). Чувствительность бактерий к антибиотикам изучали диско-диффузионным методом на «Питательной среде для определения чувствительности микроорганизмов к антибактериальным препаратам — агаре Мюллер–Хинтон II» (ГНЦ прикладной микробиологии и биотехнологии Роспотребнадзора) с использованием расширенных наборов дисков для энтеробактерий (набор № 7) и стафилококков (набор № 14) (НИИ эпидемиологии и микробиологии имени Пастера). Чувствительность штаммов к цефтазидиму-авибактаму изучали с использованием дисков цефтазидим + авибактам 10/4 мкг («Mast Group»), к тигециклину — дисков с тигециклином 15 мкг («Mast Group»). Оценку проводили в соответствии с клиническими рекомендациями «Определение чувствительности микроорганизмов к антимикробным препаратам»1.
Полногеномное секвенирование
Подготовку библиотек осуществляли с помощью набора «TrueSeq» («Illumina Inc.»), секвенирование выполняли на платформе «MiSeq» («Illumina Inc.»). Исходные риды обрабатывали утилитой Trimmomatic, для сборки ридов de novo использовали программы SPAdes v. 3.11.1 и Prokka v. 1.12 [6, 7]. Все нуклеотидные последовательности были задепонированы в международной базе GenBank.
Анализ полногеномных последовательностей проводили с использованием программ VFDB2 [8], ResFinder3 [9], веб-платформы BIGSdb-Pasteur4 [10], ресурса PubMLST5 [11], программ Spa-typer и SCCmecFinder [12, 13]. Дендрограммы строили методом максимального правдоподобия с определением генетической дистанции между штаммами микроорганизмов с помощью программы parsnp v. 1.7.4, для оценки первичной топологии древа использовали алгоритм FastTree 2.1.1 и тест Shimodaira–Hasegawa [14]. В качестве референсных использовали последовательности из базы данных GenBank: GCF_000240185.1, GCA_000013425.1, затем ветку референсного генома удаляли. Визуализацию филогенетических деревьев проводили с помощью сервиса iTol [15].
Результаты
В результате изучения молекулярно-генетических свойств штаммов K. pneumoniae 1-й группы установлено, что у всех штаммов присутствуют кластеры генов колибактина, иерсиниобактина и гены, ответственные за образование фимбрий 3-го типа, у 3 штаммов был обнаружен ген — регулятор гипермукоидного фенотипа rmpA (таблица). В геномах всех штаммов была выявлена детерминанта антибиотикорезистентности (АБР), характерная для данного вида, — бета-лактамаза blaSHV-1, которая обусловливает природную резистентность микроорганизмов к аминопенициллинам — ампициллину и амоксициллину. Фенотипически штаммы были чувствительны ко всем антибиотикам других групп: цефалоспоринам III–V поколений (цефазолин, цефотаксим, цефтриаксон, цефтазидим, цефтазидим/авибактам, цефепим, цефтаролин), аминогликозидам (гентамицин), фторхинолонам (ципрофлоксацин), карбапенемам (эртапенем, имипенем, меропенем), монобактамам (азтреонам), полимиксинам (колистин), тетрациклинам (тигециклин), триметоприм/сульфаметоксазолу. В результате анализа конституционных генов (генов домашнего хозяйства) и аллелей гена wzi была установлена принадлежность штаммов к серотипу (ST) 3 и капсульному типу (К) 3 (таблица).
Молекулярно-генетическая характеристика штаммов K. pneumoniae, выделенных в отделении новорождённых
Штамм | Сиквенс-тип (ST) | Капсульный тип (К) | Гены патогенности | Гены АБР | |||
ген регулятора гипермукоидного фенотипа | гены синтеза колибактина | гены синтеза иерсинио- бактина | гены фимбрий 3-го типа | ||||
n = 3 K. pn 849 JAVGJO000000000 K. pn 852 JAVHUD000000000 K. pn 862 JAVBWS000000000 | 3 | 3 | rmpA | clbABCDEFGHLMNOPQ | fyuA, irp1,2, ybtAEQPSTUX | mrkABCDFHIJ | blaSHV-1 |
n = 4 K. pn 850 JAVCZJ000000000 K. pn 854 JAVGJN000000000 K. pn 863 JAVBWT000000000 K. pn 893 JAVHUE000000000 | 3 | 3 | – | clbABCDEFGHLMNOPQ, | fyuA, irp1,2, ybtAEQPSTUX | mrkABCDFHIJ | blaSHV-1 |
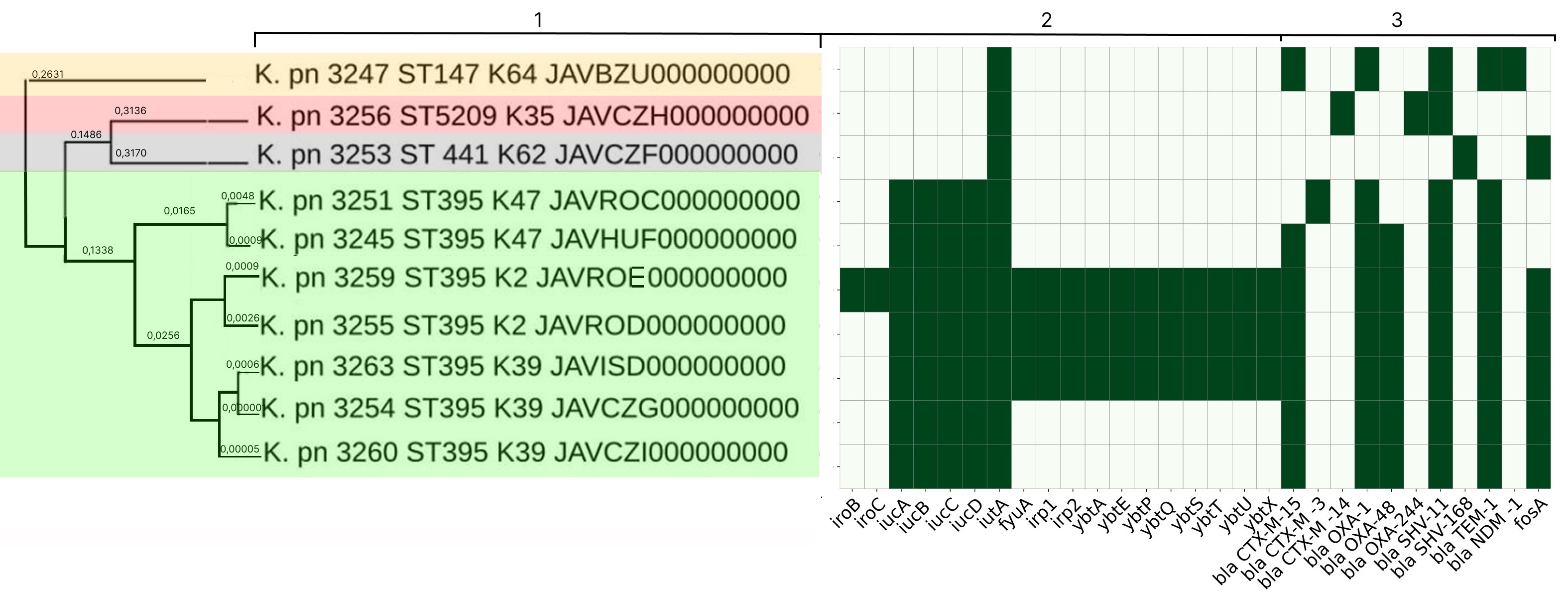
Штаммы K. pneumoniae 2-й группы различались по молекулярно-генетическим свойствам. В геномах штаммов K. pneumoniae 3254, 3260 и 3263 были обнаружены полные кластеры генов сидерофора аэробактина (iucABCD и iutA), штамм K. pneumoniae 3263 отличался наличием кластера генов иерсиниобактина (fyuA, irp1,2, ybtAEPQSTUX) (рис. 1). У всех штаммов был выявлен спектр генов АБР — бета-лактамаз и карбапенемаз blaTEM-1, blaCTX-M-15, blaSHV-11, blaOXA-1, blaOXA-48 и детерминанта устойчивости к фосфомицину — fosA. Это объясняет тот факт, что все 3 штамма имели фенотип MDR и были устойчивы к пенициллинам, цефалоспоринам, аминогликозидам, фторхинолонам, монобактамам и карбапенемам (эртапенему). По анализу конституционных генов и аллелей гена wzi была установлена принадлежность штаммов к ST395 и К39.
Рис. 1. Филогенетическая реконструкция геномов штаммов K. pneumoniae 2-й группы.
1 — вид микроорганизма, номер штамма, ST, К-тип, номер в базе данных GenBank; 2 — детерминанты генов патогенности (зелёный — наличие признака, белый — отсутствие); 3 — детерминанты АБР (зелёный — наличие признака, белый — отсутствие).
Спектр генов патогенности штаммов K. pneumoniae 3245 и 3251, как и у штаммов K. pneumoniae 3254 и 3260, был представлен кластером генов аэробактина (iucABCD и iutA) (рис. 1). У K. pneumoniae 3245 в геноме выявлен идентичный указанным выше штаммам спектр генов АБР (blaTEM-1, blaCTX-M-15, blaSHV-11, blaOXA-1, blaOXA-48), за исключением fosA. Этот штамм имел такие же фенотипы чувствительности и резистентности к антимикробным препаратам, как у штаммов, описанных выше. K. pneumoniae 3251 отличался наличием детерминанты blaCTX-M-3, отсутствием генов blaOXA-48 и blaCTX-M-15 и был чувствителен к карбапенемам. K. pneumoniae 3245 и 3251 также принадлежали к ST395, но отличались от штаммов K. pneumoniae 3254, 3260, 3263 по аллелям гена wzi и принадлежали к К47.
Штаммы K. pneumoniae 3255 и 3259 характеризовались наличием полных кластеров генов аэробактина (iucABCD и iutA) и иерсиниобактина (fyuA, irp1,2, ybtAEPQSTUX), причем у K. pneumoniae 3259 был выявлен ген сальмохелина (iro). Спектр их генов АБР был идентичен спектру детерминант K. pneumoniae 3254, 3260, 3263, 3245 (blaTEM-1, blaCTX-M-15, blaSHV-11, blaOXA-1, blaOXA-48, fosA). K. pneumoniae 3255 и 3259 также имели фенотип MDR и были устойчивы к пенициллинам, цефалоспоринам, аминогликозидам, фторхинолонам, карбапенемам (эртапенему) и тигециклину. Эти штаммы тоже принадлежали к ST395, но к К2.
У штаммов K. pneumoniae 3247, 3253 и 3256 была обнаружена лишь 1 детерминанта кластера аэробактина iutA (рис. 1). Изолят K. pneumoniae 3256 содержал в геноме бета-лактамазы blaCTX-M-14, blaOXA-244 и blaSHV-11, показал фенотипическую резистентность к цефалоспоринам, аминогликозидам и чувствительность к карбапенемам. Была установлена его принадлежность к ST5209 и К35. У K. pneumoniae 3253 выявлены 2 детерминанты АБР: blaSHV-168 и fosA, штамм был чувствителен к антибактериальным препаратам всех групп, за исключением аминопенициллинов, и отнесён к ST441 и К62. В геноме K. pneumoniae 3247 обнаружены гены blaNDM -1, blaTEM-1, blaCTX-M-15, blaSHV-11, blaOXA-1, штамм был фенотипически устойчив к пенициллинам, цефалоспоринам, аминогликозидам, фторхинолонам, всем препаратам из группы карбапенемов (эртапенем, имипенем, меропенем) и чувствителен к колистину, тигециклину и цефтазидим-авибактаму и отнесен к ST147 и K64.
В геномах всех штаммов S. aureus 3-й группы выявлены гены адгезинов, системы секреции VII типа и гамма-гемолизинов.
Штаммы S. aureus 2226, 3092, 3110, 2211, 3196, 3197 и 3198 характеризовались идентичным спектром генов патогенности, который включал, кроме вышеперечисленных, гены сериновых протеаз splABCDE, иммунного уклонения sak, scn, энтеротоксина А sea, эксфолиативного токсина eta и лейкотоксинов lukDE. У них были обнаружены SCCmec кассеты IV типа, у штаммов 3196, 3197, 3110, 3092 выявлен ген бета-лактамазы blaZ, у S. aureus 3198, 2211 и 2226 — гены резистентности к эритромицину и хлорамфениколу ermC и cat (рис. 2). Штаммы показали фенотипическую резистентность к оксациллину и цефокситину, что может указывать на их принадлежность к группе MRSA. В результате MLST-типирования и анализа последовательности повторов гена белка А была установлена их принадлежность к ST8 и spa-типу (t) t008. Также к ST8, но к t024 принадлежал штамм S. aureus 3107, отличающийся отсутствием SCCmec кассеты.
Рис. 2. Филогенетическая реконструкция геномов штаммов S. aureus 3-й группы.
1 — вид микроорганизма, номер штамма, номер в базе данных GenBank, ST, spa-тип; 2 — детерминанты генов патогенности (зелёный — наличие признака, белый — отсутствие); 3 — детерминанты АБР (зелёный — наличие признака, белый — отсутствие).
В геномах штаммов S. aureus 3086, 3087, 3088 выявлены гены патогенности splABCDE, eta, lukDE, а S. aureus 2203 и 2213 характеризовались наличием генов sak и scn (рис. 2). У S. aureus 3086, 3087, 3088 обнаружены гены АБР blaZ и ermC, у S. aureus 2213 — только blaZ. Все 5 изолятов были устойчивы к амоксициллину, тетрациклину и линкозамидам. Установлено, что штаммы 3086, 3087, 3088 принадлежали к ST1 t127, а S. aureus 2203 и 2213 — к ST1 t177.
В результате анализа молекулярно-генетических свойств штаммов S. aureus 3082, 3094, 3111, 3102, 2212, 2204 были выявлены гены эксфолиативного токсина eta и стафилокиназы sak, а также установлено отсутствие детерминант сериновых протеаз (spl) и лейкотоксинов D и E (lukDE). У S. aureus 3082 были обнаружены гены энтеротоксинов sec, sell; у 3094 — sec, sell, selo. У S. aureus 3082, 3111, 3094, 3082 присутствовал ген blaZ; у штамма 3094 — ген blaZ и детерминанта устойчивости к аминогликозидам aph(3´)-III; у S. aureus 2212 — aph(3´)-Ia. Все 5 изолятов были фенотипически резистентны к амоксициллину и амикацину. Установлена принадлежность штаммов к ST45, но разным spa-типам: t102, t362, t8416, t5599, t5132 (рис. 2).
Штаммы S. aureus 3084, 3085, 2219 характеризовались наличием генов патогенности splABCDE, eta, lukDE и blaZ. У 3085 штаммов выявлены гены энтеротоксинов sei, sej, selo, selr. Все штаммы были фенотипически резистентны к амоксициллину, установлена их принадлежность к ST5, t002 и t688 (рис. 2). S. aureus 3096 и 3089 характеризовались наличием генов splABCDE, lukDE, sak, scn, blaZ, у 3096 присутствовал ген tsst, у 3089 — sea. Они также резистентны к амоксициллину и были отнесены к ST707 и ST6, причём используемая в исследовании программа не позволила определить их spa-тип. S. aureus 3100 и 3133 характеризовались отсутствием генов сериновых протеаз (spl), лейкотоксинов D и E (lukDE), штамм 3133 был устойчив к оксациллину и цефокситину, в его геноме обнаружена SCCmec кассета IV типа. Оба штамма принадлежали к ST398, t571 и t011 (рис. 2).
Остальные 12 штаммов относились к разным ST и spa-типам. У штаммов S. aureus 2210, 3104, 3102, 2207 выявлены гены splABCDE, eta, lukDE, sak, у S. aureus 3104 — ген синдрома токсического шока tsst. Штаммы принадлежали к ST97 и разным spa-типам: t267, t3380, t11521 (рис. 2). Штаммы S. aureus 2208, 2209 и 2214 характеризовались наличием генов splABCDE, lukDE, sak и scn и были отнесены к ST12 и t156. У S. aureus 2206, 2217 и 2215 выявлены гены splABCDE и lukDE, установлена их принадлежность к ST49 и ST1027, но используемая в исследовании программа не позволила определить их spa-типы. У штамма S. aureus 2222 выявлены гены splABCDE, lukE, sak, scn, eta, tsst, он был отнесён к ST426 и t764. S. aureus 3295 характеризовался наличием детерминант sak и eta и был отнесён к ST 4. У всех 12 штаммов отсутствовали детерминанты АБР, они проявили фенотипическую чувствительность к антибактериальным препаратам всех групп.
Всего в ходе работы от пациентов с КАИК, медицинского персонала и внешней среды МО города и области были выделены штаммы S. aureus, принадлежащие к 13 различным ST и 19 spa-типам.
Обсуждение
Под циркуляцией возбудителя понимают его постоянную и последовательную передачу от одного восприимчивого организма к другому, обеспечивающую его существование как биологического вида, а также распространение возбудителя ИСМП в пределах МО, характеризующееся колонизацией объектов внешней среды, вовлечением пациентов и персонала.
Одним из важнейших критериев госпитального штамма является его принадлежность к однородной (гомогенной) по составу популяции циркулирующих микроорганизмов [16]. Однородность популяции наиболее достоверно можно оценить с помощью изучения генетических особенностей штаммов, которые подразумевают выявление и анализ генов патогенности, АБР, определение ST путём анализа аллелей конституционных генов. Для генетического типирования K. pneumoniae важное значение имеет определение их К-типа, зависящего от последовательности гена wzi, кодирующего поверхностный белок, участвующий в сборке капсулы на наружной мембране клетки. Также имеет значение установление принадлежности штаммов к одному из известных патотипов [17].
В последние годы признано существование трех патотипов K. pneumoniae: гипервирулентного (hvKp), классического (cKp) и конвергентного (hv-MDRKp). Гипервирулентный патотип связан с развитием серьёзных инвазивных инфекций у здоровых иммунокомпетентных лиц. На данный момент основным признаком, коррелирующим с гипервирулентностью, является секреция сидерофоров аэробактина, сальмохелина, иерсиниобактина и экзотоксина колибактина [18]. Классический патотип глобально распространён, именно эти клебсиеллы являются представителями микробиома человека, вызывают заболевания у ослабленных больных и принадлежат к ведущим возбудителям нозокомиальных инфекций. В их геноме обязательно присутствует комплекс бета-лактамаз и карбапенемаз, а также могут выявляться отдельные гены патогенности (кроме аэробактина и сальмохелина), K. pneumoniae конвергентного патотипа совмещают свойства гипервирулентных и классических клебсиелл, т. е. обладают высоким патогенным потенциалом и множественной АБР, способны вызывать заболевания как у здоровых иммунокомпетентных лиц, так и у больных людей с иммунодефицитами [17, 18].
Штаммы K. pneumoniae 1-й группы, выделенные в отделении новорождённых от больных детей, с оборудования и предметов ухода, были схожи по детерминантам патогенности и не содержали генов аэробактина и сальмохелина. Однако у 3 штаммов выявлен ген rmpA (гипермукоидный фенотип), который ранее ассоциировали с гипервирулентностью. На сегодняшний день признано, что целесообразно оценивать весь комплекс генов патогенности и наличие этой детерминанты не является значимым признаком [5, 18]. Присутствие гена только у 3 штаммов из 7 изученных (таблица) можно объяснить его плазмидным происхождением и высокой мобильностью в популяции микроорганизмов [19]. В геномах всех штаммов была выявлена одна бета-лактамаза — blaSHV-1, при молекулярном типировании установлена принадлежность популяции штаммов к редкому сиквенс-типу — ST3 и капсульному типу К3, ранее не выделявшемуся в России (в базе данных BIGSdb-Pasteur зарегистрировано всего 16 изолятов данного ST).
Однородность молекулярно-генетических и фенотипических свойств штаммов, выделенных от больных и из внешней среды, указывает на циркуляцию в стационаре популяции классических штаммов K. pneumoniae. Выявление данной популяции является неблагоприятным с эпидемиологической точки зрения и подтверждает тот факт, что случаи ИСМП в отделении новорождённых могут быть связаны не только с гипервирулентными штаммами K. pneumoniae, но и с классическими, не обладающими множественной АБР и широким спектром генов патогенности [20]. Это обусловливает необходимость непрерывного молекулярно-эпидемиологического мониторинга штаммов K. pneumoniae, принадлежащих к разным патотипам, в микробиоте любых локусов новорождённых, матерей, медицинского персонала и объектов внешней среды в отделениях новорождённых педиатрических стационаров и перинатальных центров, изучение их свойств и интеграцию данных в единую российскую базу VGARus.
При анализе штаммов K. pneumoniae 2-й группы, выделенных в многопрофильном стационаре, установлено, что штаммы K. pneumoniae 3254, 3260 и 3263 имели идентичный спектр генов патогенности и АБР, а также одинаковый фенотип АБР. Данные штаммы принадлежали к ST395 и К39 и были отнесены к конвергентному патотипу ввиду наличия у них генов аэробактина и спектра бета-лактамаз и карбапенемаз. Штаммы были выделены как от больных, так и из внешней среды, что свидетельствует о циркуляции штаммов K. pneumoniae ST395 и К39 в стационаре. Наличие у K. pneumoniae 3263 кластера генов иерсиниобактина может объясняться процессами горизонтального переноса данного гена, ассоциированного с транспозоном, и обладающего высокой мобильностью [21]. Штаммы K. pneumoniae 3255 и 3259 (ST395-К2) и K. pneumoniae 3245 и 3251 (ST395-К47) выделялись в единичных случаях только у больных, содержали в геномах гены аэробактина и детерминанты АБР и также были отнесены к конвергентному патотипу.
Таким образом, 7 из 10 штаммов, выделенных в многопрофильном стационаре, принадлежали к ST395, что могло бы быть расценено как свидетельство их циркуляции. Это согласуется с данными научной литературы о широком распространении штаммов данного ST, среди которых часто встречаются штаммы с выраженным патогенным потенциалом, способные вызывать тяжёлые системные инфекции [5, 17]. Однако углублённый анализ молекулярно-генетических свойств штаммов позволил установить их неоднородность даже в пределах одного ST и сделать вывод о циркуляции в стационаре только популяции штаммов K. pneumoniae ST395-К39.
Все остальные штаммы K. pneumoniae: 3256 (ST5209-К35), 3253 (ST 441-К62), 3247 (ST147-К64), были отнесены к классическим и выделялись в единичных случаях только у больных. Необходимо отметить, что в геноме штамма K. pneumoniae 3247 обнаружен ген Нью-Дели металло-бета-лактамазы blaNDM-1, что объясняет высокую степень устойчивости этого штамма к антибиотикам. Известно, что данная детерминанта ассоциирована с плазмидами и способна к активному горизонтальному переносу [17], поэтому обнаружение такого штамма является неблагоприятным с эпидемиологической точки зрения, т. к. может привести к быстрому глобальному распространению полирезистентной популяции в стационаре.
В составе филогенетического древа все штаммы 2-й группы кластеризовались согласно ST и К-типам. Штаммы K. pneumoniae ST395 образовали 3 подкластера согласно их К-типам, 3 штамма K. pneumoniae ST395-К39 вошли в состав единого кластера, объединяющего штаммы, выделенные от больных и из внешней среды отделения МО (рис. 1).
Для изучения циркуляции штаммов S. aureus важное значение имеет анализ спектров генов патогенности, АБР, принадлежности к группе MRSA или MSSA (метициллин-чувствительные стафилококки), определения ST и spa-типа [22], который основан на анализе последовательности повторов гена поверхностного белка клеточной стенки стафилококка (белка А) [4]. При расследовании локальных вспышек заболеваемости установление spa-типа является важным этапом, так как с его помощью возможно установить различия между штаммами, принадлежащими к одному ST.
При проведении молекулярно-генетического анализа штаммов S. aureus III группы, ассоциированных с КАИК, было установлено, что в геномах 7 штаммов S. aureus 2226, 3092, 3110, 2211, 3196, 3197 и 3198 присутствовала SCCmec кассета IV типа, они имели идентичные спектры генов патогенности, одинаковые фенотипы резистентности и принадлежали к ST8 t008.
Штаммы были выделены как от медицинского сотрудника, так и от больных с диагнозом КАИК, получавших амбулаторное лечение в гемодиализном центре и проходивших стационарное лечение в лечебно-профилактических МО города, что подтверждает факт их циркуляции. Различие штаммов по спектру детерминант blaZ, ermC и cat (рис. 2) может объясняться тем, что эти гены входят в состав плазмид и обладают высокой мобильностью [23–25]. По данным научной литературы, штаммы S. aureus ST8 t008 SCCmecIV являются распространёнными, часто связаны с ИСМП и выявляются на территории России с 1990-х гг. [4].
Штаммы S. aureus 3086, 3087, 3088 также идентичны по генам патогенности, детерминантам и фенотипу АБР. Они относились к группе MSSA, были типированы как S. aureus ST1 t127 и выделялись от 3 больных (место выхода перитонеального катетера). В то же время у этих же больных из перитонеальной жидкости были изолированы штаммы S. aureus других сиквенс-типов — ST5 (t688), ST97 (t267) и ST45 (t8416). Это свидетельствует о том, что популяция штаммов S. aureus ST1 t127 циркулирует в этой МО.
Все остальные изоляты S. aureus III группы были гетерогенны по спектру детерминант, фенотипу АБР, сиквенс-типам и spa-типам, что не позволяет оценить их эпидемиологическую значимость в данном исследовании.
Филогенетический анализ полногеномных последовательностей штаммов показал наличие пяти кластеров, объединяющих штаммы, принадлежащие к одинаковым сиквенс-типам и клональным комплексам (CC), которые представляют собой группы генетически близких сиквенс-типов. Штаммы S. aureus ST8, ST97, ST12, ST1 образовали самостоятельные группы (А, В, С, D), в состав единого крупного кластера E вошли штаммы S. aureus различных сиквенс-типов: ST6 и ST5, принадлежащие к пятому клональному комплексу (CC5), S. aureus ST4 и ST45, принадлежащие CC45, а также штаммы ST49, ST1027, ST707, ST398 и ST426, не принадлежащие к определенным клональным комплексам, но имеющие между собой филогенетическое родство (рис. 2).
Таким образом, в отделениях гемодиализа МО города и области были выделены штаммы S. aureus 13 различных сиквенс-типов и 19 spa-типов и показана циркуляция популяций эпидемических штаммов S. aureus MRSA (SCCmec IV) молекулярного типа ST8 t008 и S. aureus MSSA молекулярного типа ST1 t127.
Заключение
В результате проведенного исследования в МО Нижегородской области было выявлено большое генетическое разнообразие штаммов K. pneumoniae и S. aureus. Углублённый анализ показал циркуляцию в отделении новорожденных педиатрического стационара популяции классических штаммов K. pneumoniae ST3-К3, выделенных из желудочно-кишечного тракта детей, с оборудования и предметов ухода, содержащих ряд генов патогенности и бета-лактамазу blaSHV-1.
В многопрофильном стационаре обнаружена циркуляция штаммов конвергентного патотипа K. pneumoniae ST395-К39, а также выявлены штаммы K. pneumoniae ST395-К2, ST395-К47, ST5209-К35, ST441-К62, ST147-К64, содержащие в геноме спектр генов патогенности и бета-лактамаз, в том числе Нью-Дели металло-бета-лактамазу blaNDM-1, что может привести к формированию и распространению в стационаре полирезистентного клона, способного сменить циркулирующий возбудитель и привести к возникновению вспышек ИСМП.
В отделениях гемодиализа МО города и области выявлена циркуляция популяций S. aureus MRSA (SCCmec IV) ST8 t008 и S. aureus MSSA ST1 t127, а также обнаружены другие штаммы S. aureus, принадлежащие к 11 различным ST и 17 spa-типам, потенциально способные к формированию госпитальных клонов и широкому распространению в стационаре. В связи с этим с целью недопущения возникновения и распространения случаев ИСМП необходимо проведение в стационарах обязательного непрерывного микробиологического мониторинга, неотъемлемой частью которого должен являться молекулярно-эпидемиологический мониторинг, направленный на получение актуальной информации о генетических вариантах циркулирующих возбудителей, в том числе K. pneumoniae и S. aureus.
1 Рекомендации МАКМАХ версия 2024. URL: https://www.antibiotic.ru/minzdrav/category/clinical-recommendations
2 Virulence factor database. URL: http://www.mgc.ac.cn/VFs
3 ResFinder. URL: http://genepi.food.dtu.dk/resfinder
4 Institut Pasteur Klebsiella pneumoniae species complex.
URL: https://bigsdb.pasteur.fr/klebsiella
5 PubMLST. MLST Database. Staphylococcus aureus.
URL: https://pubmlst.org/organisms/staphylococcus-aureus
Об авторах
Ирина Владленовна Соловьева
Нижегородский научно-исследовательский институт эпидемиологии и микробиологии имени академика И.Н. Блохиной Роспотребнадзора
Email: lab-lb@yandex.ru
ORCID iD: 0000-0002-3136-9500
д-р биол. наук, доцент, в. н. с., зав. лаб. микробиома человека и средств его коррекции
Россия, Нижний НовгородАнна Георгиевна Точилина
Нижегородский научно-исследовательский институт эпидемиологии и микробиологии имени академика И.Н. Блохиной Роспотребнадзора
Автор, ответственный за переписку.
Email: lab-lb@yandex.ru
ORCID iD: 0000-0001-7753-5730
канд. биол. наук, доцент, с. н. с. лаб. микробиома человека и средств его коррекции
Россия, Нижний НовгородИрина Викторовна Белова
Нижегородский научно-исследовательский институт эпидемиологии и микробиологии имени академика И.Н. Блохиной Роспотребнадзора
Email: lab-lb@yandex.ru
ORCID iD: 0000-0003-3402-1160
канд. мед. наук, доцент, в. н. с. лаб. микробиома человека и средств его коррекции
Россия, Нижний НовгородНаталья Николаевна Зайцева
Нижегородский научно-исследовательский институт эпидемиологии и микробиологии имени академика И.Н. Блохиной Роспотребнадзора
Email: nniiem@yandex.ru
ORCID iD: 0000-0001-5370-4026
д-р мед. наук, директор
Россия, Нижний НовгородНаталия Сергеевна Кучеренко
Управление Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека по Нижегородской области
Email: sanepid@sinn.ru
ORCID iD: 0000-0002-0509-3459
руководитель
Россия, Нижний НовгородНаталья Александровна Садыкова
Управление Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека по Нижегородской области
Email: sanepid@sinn.ru
ORCID iD: 0000-0001-9412-8678
заместитель руководителя
Россия, Нижний НовгородСветлана Борисовна Молодцова
Нижегородский научно-исследовательский институт эпидемиологии и микробиологии имени академика И.Н. Блохиной Роспотребнадзора
Email: lab-lb@yandex.ru
ORCID iD: 0000-0002-4750-5925
н. с. лаб. микробиома человека и средств его коррекции
Россия, Нижний НовгородВасилий Сергеевич Кропотов
Нижегородский научно-исследовательский институт эпидемиологии и микробиологии имени академика И.Н. Блохиной Роспотребнадзора
Email: lab-lb@yandex.ru
ORCID iD: 0000-0002-6903-962X
канд. биол. наук, с. н. с. лаб. микробиома человека и средств его коррекции
Россия, Нижний НовгородСписок литературы
- Зубков В.В., Любасовская Л.А., Рюмина И.И. и др. Микробиологический мониторинг в системе инфекционного контроля неонатальных стационаров. Российский вестник перинатологии и педиатрии. 2014;59(1):51–6. Zubkov V.V., Lyubasovskaya L.A., Ryumina I.I., et al. Microbiological monitoring of the infection control system of neonatal hospitals. Russain Bulletin of Perinatology and Pediatrics. 2014;59(1):51–6. EDN: https://elibrary.ru/rwimxh
- Михайловская В.С., Селиванова П.А., Кузнецова М.В. Распространённость генов qacEΔ1, qacE, oqxA, oqxB, acrA, cepA и zitB среди мультирезистентных Klebsiella pneumoniae, выделенных в кардиохирургическом стационаре. Журнал микробиологии, эпидемиологии и иммунобиологии. 2024;101(4):502–11. Mihailovskaya V.S., Selivanova P.A., Kuznetsova M.V. Prevalence of qacEΔ1, qacE, oqxA, oqxB, acrA, cepA and zitB genes among multidrug-resistant Klebsiella pneumoniae isolated in a cardiac hospital. Journal of Microbiology, Epidemiology and Immunobiology. 2024;101(4): 502–11. DOI: https://doi.org/10.36233/0372-9311-548 EDN: https://elibrary.ru/fcoyee
- Новикова И.Е., Садеева З.З., Алябьева Н.М. и др. Антибиотикорезистентность и вирулентность карбапенем-устойчивых штаммов Klebsiella pneumoniae, выделенных у детей в реанимационных и хирургических отделениях. Журнал микробиологии, эпидемиологии и иммунобиологии. 2023;100(4):321–32. Novikova I.E., Sadeeva Z.Z., Alyabyeva N.M., et al. Antimicrobial resistance and virulence of carbapenem-resistant Klebsiella pneumoniae strains isolated from children in intensive care and surgical units. Journal of Microbiology, Epidemiology and Immunobiology. 2023;100(4):321–32. DOI: https://doi.org/10.36233/0372-9311-373 EDN: https://elibrary.ru/rmjxsl
- Романов А.В., Дехнич А.В., Эйдельштейн М.В. Молекулярная эпидемиология штаммов Staphylococcus aureus в детских стационарах России. Клиническая микробиология и антимикробная химиотерапия. 2012;14(3):201–8. Romanov А.V., Dekhnich А.V., Edelstein М.V. Molecular epidemiology of Staphylococcus aureus in Russian pediatric hospitals. Clinical Microbiology and Antimicrobial Chemotherapy. 2012;14(3):201–8. EDN: https://elibrary.ru/pcnnlp
- Воронина О.Л., Кунда М.С., Рыжова Н.Н. и др. Геномные особенности резистентных изолятов Klebsiella pneumoniae, выделенных из кровяного русла и ликвора пациентов детского стационара. Журнал микробиологии, эпидемиологии и иммунобиологии. 2023;100(6):399–409. Voronina O.L., Kunda M.S., Ryzhova N.N., et al. Genomic features of resistans Klebsiella pneumoniae, isolated from the bloodstream and cerebrospinal fluid of pediatric hospital patients. Journal of Microbiology, Epidemiology and Immunobiology. 2023;100(6):399–409. DOI: https://doi.org/10.36233/0372-9311-430 EDN: https://elibrary.ru/ylxbdz
- Bankevich A., Nurk S., Antipov D., et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. J. Comput. Biol. 2012;19(5):455–77. DOI: https://doi.org/10.1089/cmb.2012.0021
- Seemann T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 2014;30(14):2068–9. DOI: https://doi.org/10.1093/bioinformatics/btu153
- Chen L.H., Yang J., Yu J., et al. VFDB: a reference database for bacterial virulence factors. Nucleic Acids Res. 2005;33(1):325–8. DOI: https://doi.org/10.1093/nar/gki008
- Bortolaia V., Kaas R.S., Ruppe E., et al. ResFinder 4.0 for predictions of phenotypes from genotypes. J. Antimicrob. Chemother. 2020;75(12):3491–500. DOI: https://doi.org/10.1093/jac/dkaa345
- Jolley K.A., Bray J.E., Maiden M.C.J. Open-access bacterial population genomics: BIGSdb software, the PubMLST.org website and their applications. Wellcome Open Res. 2018;3:124. DOI: https://doi.org/10.12688/wellcomeopenres.14826.1
- Guo C., Yang X., Wu Y., et al. MLST-based inference of genetic diversity and population structure of clinical Klebsiella pneumoniae, China. Sci. Rep. 2015;5:7612. DOI: https://doi.org/10.1038/srep07612
- Bartels M.D., Petersen A., Worning P., et al. Comparing whole-genome sequencing with Sanger sequencing for spa typing of methicillin-resistant Staphylococcus aureus. J. Clin. Microbiol. 2014;52(12):4305–8. DOI: https://doi.org/10.1128/jcm.01979-14
- International Working Group on the Classification of Staphylococcal Cassette Chromosome Elements (IWG-SCC). Classification of staphylococcal cassette chromosome mec (SCCmec): guidelines for reporting novel SCCmec elements. Antimicrob. Agents Chemother. 2009;53(12):4961–7. DOI: https://doi.org/10.1128/aac.00579-09
- Treangen T.J., Ondov B.D., Koren S., et al. The Harvest suite for rapid core-genome alignment and visualization of thousands of intraspecific microbial genomes. Genome Biol. 2014;15(11):524. DOI: https://doi.org/10.1186/s13059-014-0524-x
- Letunic I., Bork P. Interactive tree of life (iTOL) v5: an online tool for phylogenetic tree display and annotation. Nucleic Acids Res. 2021;49(W1):293–6. DOI: https://doi.org/10.1093/nar/gkab301
- Брико Н.И., Брусина Е.Б., Зуева Л.П. и др. Госпитальный штамм – непознанная реальность. Эпидемиология и вакцинопрофилактика. 2013;(1):30–5. Briko N.I., Brusina E.B., Zueva L.P., et al. Hospital strain — a mysterious reality. Epidemiology and Vaccinal Prevention. 2013;(1):30–5. EDN: https://elibrary.ru/pvsumn
- Агеевец В.А., Агеевец И.В., Сидоренко С.В. Конвергенция множественной резистентности и гипервирулентности у Klebsiella pneumoniae. Инфекция и иммунитет. 2022;12(3):450–60. Ageevets V.A., Ageevets I.V., Sidorenko S.V. Convergence of multiple resistance and hypervirulence in Klebsiella pneumoniae. Russian Journal of Infection and Immunity. 2022;12(3):450–60. DOI: https://doi.org/10.15789/2220-7619-COM-1825 EDN: https://elibrary.ru/ucpmnf
- Комисарова Е.В., Воложанцев Н.В. Гипервирулентная Klebsiella pneumoniae — новая инфекционная угроза. Инфекционные болезни. 2019;17(3):81–9. Komisarova E.V., Volozhantsev N.V. Hypervirulent Klebsiella pneumoniae: a new infectious threat. Infectious diseases. 2019;17(3):81–9. DOI: https://doi.org/10.20953/1729-9225-2019-3-81-89 EDN: https://elibrary.ru/idonjy
- Ali M.R., Yang Y., Dai Y., et al. Prevalence of multidrug-resistant hypervirulent Klebsiella pneumoniae without defined hypervirulent biomarkers in Anhui, China: a new dimension of hypervirulence. Front. Microbiol. 2023;14:1247091. DOI: https://doi.org/10.3389/fmicb.2023.1247091.8
- Устюжанин А.В., Маханёк А.А., Чистякова Г.Н. и др. Сравнительный геномный анализ клинических изолятов Klebsiella pneumoniae, выделенных от новорождённых детей с различными исходами инфекционного процесса в неонатальном периоде. Журнал микробиологии, эпидемиологии и иммунобиологии. 2025;102(1):62–71. Ustyuzhanin A.V., Makhanyok A.A., Chistyakova G.N. et al. Comparative genomic analysis of clinical isolates of Klebsiella pneumoniae isolated from newborns with different outcomes of the infectious process in the neonatal period. Journal of Microbiology, Epidemiology and Immunobiology. 2025;102(1):62–71. DOI: https://doi.org/10.36233/0372-9311-544 EDN: https://elibrary.ru/zxmnbq
- Самойлова А.А., Краева Л.А., Михайлов Н.В. и др. Геномный анализ вирулентности и антибиотикорезистентности штаммов Klebsiella pneumoniae. Инфекция и иммунитет. 2024;14(2):339–50. Samoylova A.A., Kraeva L.A., Mikhailov N.V., et al. Genomic analysis of virulence and antibiotic resistance of Klebsiella pneumoniae strains. Infection and Immunity. 2024;14(2):339–50. DOI: https://doi.org/10.15789/2220-7619-GAO-15645 EDN: https://elibrary.ru/cmtxuz
- Скачкова Т.С., Замятин М.Н., Орлова О.А. и др. Мониторинг метициллинрезистентных штаммов стафилококка в многопрофильном стационаре Москвы с помощью молекулярно-биологических методов. Эпидемиология и вакцинопрофилактика. 2021;20(1):44–50. Skachkova T.S., Zamyatin M.N., Orlova O.A., et al. Monitoring of methicillin-resistant staphylococcal strains in the Moscow medical and surgical center using molecular biological methods. Epidemiology and Vaccinal Prevention. 2021;20(1):44–50. DOI: https://doi.org/10.31631/2073-3046-2021-20-1-44-50 EDN: https://elibrary.ru/fwncis
- Якубцевич Р.Э., Лемеш А.В., Кирячков Ю.Ю. Патогенетические механизмы формирования генетической устойчивости к антибиотикам при лечении тяжелых инфекций в интенсивной терапии. Журнал Гродненского государственного медицинского университета. 2021;19(3):255–62. Yakubtsevich R.E., Lemesh A.V., Kiryachkov Yu.Yu. Pathogenetic mechanisms of formation of genetic resistance to antibiotics in the treatment of severe infections in intensive care. Journal of the Grodno State Medical University. 2021;19(3):255–62. DOI: https://doi.org/10.25298/2221-8785-2021-19-3-255-262 EDN: https://elibrary.ru/rtqhgv
- Malachowa N., DeLeo F.R. Mobile genetic elements of Staphylococcus aureus. Cell Mol. Life Sci. 2010;67(18):3057–71. DOI: https://doi.org/10.1007/s00018-010-0389-4
- Schwarz S., Cardoso M. Nucleotide sequence and phylogeny of a chloramphenicol acetyltransferase encoded by the plasmid pSCS7 from Staphylococcus aureus. Antimicrob. Agents Chemother. 1991;35(8):1551–6. DOI: https://doi.org/10.1128/aac.35.8.1551
Дополнительные файлы