Мультиплексная ПЦР в режиме реального времени для выявления генов qacA/B и smr у грамположительных бактерий
- Авторы: Ковальчук С.Н.1, Архипова А.Л.1, Ковылкова С.Ю.1, Ильина Е.Н.1, Федорова Л.С.1
-
Учреждения:
- Институт системной биологии и медицины
- Выпуск: Том 101, № 5 (2024)
- Страницы: 692-698
- Раздел: НАУКА И ПРАКТИКА
- URL: https://microbiol.crie.ru/jour/article/view/18690
- DOI: https://doi.org/10.36233/0372-9311-574
- EDN: https://elibrary.ru/ubbcnj
- ID: 18690
Цитировать
Аннотация
Актуальность. Дезинфицирующие вещества (ДВ) являются эффективными средствами неспецифической профилактики инфекций, связанных с оказанием медицинской помощи. Нарушение режимов применения ДВ приводит к формированию устойчивости микроорганизмов к ним. Для целей мониторинга распространения клинически значимых микроорганизмов, устойчивых к ДВ, остаётся актуальной разработка методов их выявления, в том числе молекулярно-генетических.
Целью исследования была разработка мультиплексной полимеразной цепной реакции в режиме реального времени (ПЦР-РВ) для выявления у грамположительных бактерий генов qacA/B и smr — детерминант устойчивости к ДВ из группы катионных поверхностно-активных веществ (КПАВ).
Материалы и методы. Поиск консервативных участков генов qacA, qacB и smr и разработку праймеров и зондов проводили с помощью программ BLASTN, GeneRunner и Multiple Primer Analyzer. Для оценки аналитической чувствительности мультиплексной ПЦР-РВ были сконструированы плазмиды pTZ57-qacA/B, pTZ57-smr и pTZ57-16S, содержащие фрагменты генов qacA/B, smr и 16S рРНК длиной 197, 127 и 287 п. н. соответственно. Апробацию метода проводили с использованием клинических изолятов грамположительных бактерий (n = 30).
Результаты. Разработана мультиплексная ПЦР-РВ с использованием зондов TaqMan для выявления генов qacA/B и smr у грамположительных бактерий. В качестве внутреннего контроля амплификации был использован ген 16S рРНК. Чувствительность мультиплексной ПЦР-РВ составила 103 копий для всех генов. Апробация мультиплексной ПЦР-РВ показала, что гены qacA/B присутствовали у 30% исследованных изолятов, smr — у 10%. Воспроизводимость результатов тестирования составила 100%. Специфичность разработанной мультиплексной ПЦР-РВ составила 100%.
Заключение. Разработанная мультиплексная ПЦР-РВ характеризуется высокой специфичностью и быстротой анализа, а также наличием внутреннего контроля амплификации и может быть использована для выявления грамположительных бактерий, потенциально устойчивых к ДВ из группы КПАВ, при проведении молекулярно-генетических исследований.
Ключевые слова
Полный текст
Введение
Распространение в последние десятилетия штаммов патогенных бактерий, устойчивых к антимикробным препаратам и дезинфицирующим веществам (ДВ), — одна из острых проблем современного здравоохранения. ДВ являются одним из наиболее эффективных средств при проведении неспецифической профилактики инфекционных заболеваний и играют ведущую роль в системе мер по профилактике инфекций, связанных с оказанием медицинской помощи, а также широко используются на предприятиях общественного питания, пищевой промышленности, на коммунальных объектах, в образовательных учреждениях и в быту. Однако наблюдаемый с 1950-х гг. феномен устойчивости микроорганизмов к ДВ приводит к резкому снижению эффективности дезинфекционных мероприятий [1], что связано с использованием неэффективных режимов их применения, приводящим к формированию устойчивости микроорганизмов к ДВ, а также к перекрестной устойчивости к антибиотикам в силу наличия общих механизмов действия [2, 3].
Катионные поверхностно-активные вещества (КПАВ), к которым относятся четвертичные аммониевые соединения (ЧАС), производные гуанидина и третичные амины, являются наиболее часто используемыми ДВ [4, 5]. В 2023 г. ЧАС занимали наибольшую долю ДВ на мировом рынке1, и сохранение этой тенденции прогнозируется на ближайшие 10 лет2. Согласно имеющимся данным, на российском рынке их доля составляет 50–70% [6, 7].
Основным механизмом формирования микробной резистентности к ЧАС является снижение их внутриклеточной концентрации за счёт выведения из клетки c помощью эффлюксных насосов [5, 8], которые на основе структурного сходства и особенностей функционирования объединены в 6 суперсемейств:
- RND (Resistance-Nodulation Division);
- SMR (Small Multidrug Resistance);
- MATE (Multidrug And Toxic compound Extrusion);
- MFS (Major Facilitator Superfamily);
- ABC (ATP Binding Cassette);
- PACE (Proteobacterial Antimicrobial Compound Efflux) [9, 10].
Устойчивость к ЧАС грамположительных бактерий в основном связана с эффлюксными насосами QacA, QacB (суперсемейство MFS), а также Smr (QacC), QacG, QacH и QacJ, относящимися к надсемейству SMR [11, 12]. Мониторинг распространённости этих эффлюксных насосов среди грамположительных бактерий с помощью молекулярногенетических методов ведётся во многих странах и показал, что наиболее часто встречаются гены qacA, qacB и smr [13–16]. При этом информация о распространении этих генов среди российских изолятов грамположительных бактерий практически отсутствует. Лишь в одной публикации были представлены данные о наличии генов qacA и qacB у изолятов стафилоккоков, выделенных из смывов с поверхностей в общественных местах Новосибирска [17]. С учётом того, что гены qacA, qacB и smr имеют преимущественно плазмидную локализацию, они могут широко распространяться посредством горизонтального переноса, приводя к формированию приобретённой устойчивости к КПАВ у грамположительных бактерий [12].
Целью исследования была разработка мультиплексной полимеразной цепной реакции в режиме реального времени (ПЦР-РВ) с флуоресцентно-мечеными зондами для выявления генов qacA/B и smr у грамположительных бактерий.
Материалы и методы
Бактериальные изоляты
В исследовании были использованы изоляты грамположительных бактерий видов Staphylococcus aureus (n = 12), S. haemolyticus (n = 6), Enterococcus faecium (n = 6) и E. faecalis (n = 4), Streptococcus parasanguinis (n = 1) и S. epidermidis (n = 1), полученные от медицинских учреждений Москвы. Изоляты выделены из смывов, взятых с объектов внутрибольничной среды — поверхностей аппаратов ИВЛ и УЗИ, прикроватных тумбочек, поручней кроватей. Бактерии культивировали в течение 16 ч при 37ºC в ГРМ-бульоне (ГНЦ ПМБ). Видовую принадлежность изолятов определяли с помощью масс-спектрометра «SMART MS 5020» («Zhuhai DL Biotech Co., Ltd»).
Выделение ДНК
ДНК выделяли из бактериальной культуры с помощью набора для выделения ДНК «ExtractDNA Blood & Cells» (ЗАО «Евроген») согласно рекомендациям производителя и хранили при –20ºC. Концентрацию образцов ДНК определяли с помощью спектрофотометра «NanoDrop 2000C» («ThermoFS»).
Разработка праймеров и зондов
Нуклеотидные последовательности генов эффлюксных насосов qacA, qacB, smr и 16S рРНК грамположительных бактерий были взяты из базы данных GenBank3. Их анализ проводили с помощью программы BLASTN4. Праймеры и зонды были разработаны на основе консервативных участков генов с использованием программ «GeneRunner v. 62.2.55 Beta»5 и «Multiple Primer Analyzer»6.
Проведение мультиплексной ПЦР-РВ
Мультиплексную ПЦР-РВ проводили в объёме 25 мкл в 96-луночных планшетах для ПЦР с использованием амплификатора «CFX96 Real-Time System» («Bio-Rad Laboratories, Inc.»). Реакционная смесь включала в себя 5× буфер и 2,5 ЕД Taq-полимеразы (ООО «НПФ Литех»), праймеры (по 0,5 мкМ каждого), зонды TaqMan (по 0,25 мкМ каждого; таблица) и образец ДНК (5 нг). Зонды TaqMan синтезированы OOO «ДНК-синтез», праймеры — ЗАО «Евроген». Профиль реакции: 95ºC — 2 мин, 95ºC — 15 с, 56ºC — 20 с (36 циклов), 72ºC — 30 с.
Нуклеотидные последовательности праймеров и зондов
Nucleotide sequences of the primers and probes
Гены Genes | Нуклеотидная последовательность (5’–3’) Nucleotide sequence (5’–3’) | Размер, п. н. Size, bp |
qacA/B | qacA/B-D: 5'-CTGGCTTATACCTATTACCTA-3' qacA/B-R: 5'-TCCAACTAAAATTAATGCTAAAG -3' qacA/B-Pb: 5'-HEX- CGATTTGGACCGAAAATAGTGTTAC-BHQ1 | 197 |
smr | smr-F: AGTAAAACAATGCAACACCTAC-3' smr-R: ATACTATAGTTATTAGATTTATTTG-3' smr-Pb: 5'-FAM-TTAGTCTTAACAACCGTAGTCTCAAT-BHQ1 | 127 |
16S рРНК 16S rRNA | 16S-D: 5'-CAGCAGCCGCGGTAATAC-3' Bakt_805R: 5'-GACTACHVGGGTATCTAATCC-3' 16S-Pb: Cy5-5'-TGTAGCGGTGAAATGCG- BHQ2' | 287 |
Определение аналитической чувствительности мультиплексной ПЦР-РВ
Для оценки аналитической чувствительности метода сконструированы плазмиды pTZ57-qacA/B, pTZ57-smr и pTZ57-16S, содержащие фрагменты генов qacA/B, smr и 16S рРНК длиной 197, 127 и 287 п. н. соответственно. Клонирование проводили с использованием набора реактивов «InsTAclone PCR Cloning Kit» («ThermoFS») согласно рекомендациями производителя. Оценку концентраций плазмид проводили с помощью набора «Spectra Q BR» (ООО «Сесана») и флуориметра «Qubit» («ThermoFS»). Секвенирование полученных плазмид проводили по методу Сэнгера. Путём 10-кратных разведений получены образцы плазмид pTZ57-qacA/B, pTZ57-smr и pTZ57-16S с концентрацией 101–106 копий в 1 мкл, которые использовались в качестве матрицы для ПЦР-РВ. Анализ результатов проводили при помощи программного обеспечения к прибору «CFX96 Real-Time System» («Bio-Rad Laboratories, Inc.»).
Апробация и валидация метода
Апробацию мультиплексной ПЦР-РВ проводили с использованием ДНК, выделенной из изолятов грамположительных бактерий (n = 30). Для оценки вариабельности значений порогового цикла ПЦР (Cq) каждый образец тестировали в 3-кратной повторности и рассчитывали средние значения Cq, стандартные отклонения (SD) и коэффициент вариабельности (Cv, %). Полученные ампликоны генов qacA/B и smr проанализированы с помощью электрофорезного разделения в 1,5% агарозном геле и секвенированы по методу Сэнгера. Для валидации мультиплексной ПЦР-РВ использовали описанные ранее методы ПЦР с электрофоретической детекцией фрагментов генов qacA/B [18] и smr [19].
Результаты
Для мультиплексной ПЦР-РВ разработаны праймеры и зонды типа TaqMan, комплементарные высококонсервативным участкам генов qacA, qacB и smr (таблица), которые были выявлены на основе множественного выравнивания всех полноразмерных нуклеотидных последовательностей этих генов, имеющихся в базах данных GenBank, EMBL7 и DDBJ8. Проанализировано 302 нуклеотидных последовательности генов qacA/B и 220 последовательностей гена smr. Анализ генов qacA и qacВ показал, что они различались 8 нуклеотидами, поэтому для них разработаны общие праймеры и зонд.
При проведении тестирования методом ПЦР важным является использование внутреннего контроля амплификации, который позволяет исключить ложноотрицательные результаты вследствие отсутствия или недостаточного для детекции количества ДНК в реакционной смеси. В качестве эндогенного внутреннего контроля амплификации в разработанной мультиплексной ПЦР-РВ использован ген 16S рРНК. Праймер 16S-D и зонд были разработаны на основе анализа 5000 последовательностей 16S рРНК (таблица). В качестве обратного праймера использовали универсальный праймер Bakt_805R, разработанный ранее [20].
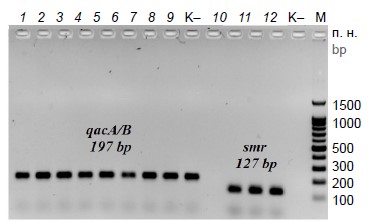
C помощью разработанных нами праймеров проведён поиск изолятов грамположительных бактерий (n = 30), содержащих гены qacA/B и smr. Полученные ампликоны были проанализированы с помощью электрофорезного разделения в 1,5% агарозном геле. Длины ампликонов генов qacA/B и smr соответствовали ожидаемым (таблица; рис. 1). Специфичность праймеров была подтверждена секвенированием ампликонов по методу Сэнгера.
Рис. 1. Результаты амплификации фрагментов генов qacA/B и smr.
1–9 — qacA/B-позитивные изоляты; 10–12 — smr-позитивные изоляты. К– — отрицательный контроль. M — маркер длин ДНК.
Fig. 1. Results of the amplification of qacA/B and smr genes.
1–9 — qacA/B-positive isolates; 10–12 — smr-positive isolates. K– — negative control. M — DNA length marker.
Для оценки аналитической чувствительности мультиплексной ПЦР-РВ сконструированы плазмиды pTZ57-qacA/B, pTZ57-smr и pTZ57-16S, содержащие полученные ампликоны генов qacA/B, smr и 16S рРНК. С помощью 10-кратных разведений получены их образцы с концентрацией от 101 до 106 копий в 1 мкл, которые использовались в качестве матрицы для разработанной мультиплексной ПЦР-РВ. Чувствительность мультиплексной ПЦР-РВ составила 103 копий для всех генов. Эффективность амплификации генов qacA/B, smr и 16S рРНК при составила 95,1, 91,3 и 101,8% соответственно (рис. 2).
Рис. 2. Результаты определения чувствительности мультиплексной ПЦР-РВ для выявления генов qacA/B и smr у грамположительных бактерий.
Fig. 2. Results of multiplex PCR sensitivity evaluation for detection of qacA/B and smr genes in gram-positive bacteria.
Апробацию разработанной мультиплексной ПЦР-РВ проводили с использованием 30 клинических изолятов грамположительных бактерий, полученных от медицинских учреждений Москвы. Тестирование проводили в 3 повторах. Выявлено, что 30% изолятов (6 изолятов S. haemolyticus и по 1 изоляту E. faecium, E. faecalis и S. parasanguinis) имели гены qacA/B и 10% изолятов (2 изолята S. haemolyticus и изолят S. epidermidis) содержали ген smr. У 2 изолятов S. haemolyticus были найдены и qacA/B, и smr. Воспроизводимость результатов тестирования для всех образцов составила 100%, коэффициент вариабельности значений Cq — от 3,4 до 6,2%. Результаты мультиплексной ПЦР-РВ полностью совпали как с данными моноплекных ПЦР с электрофорезной детекцией, так и с результатами секвенирования ампликонов по методу Сэнгера. В качестве альтернативного метода сравнения для разработанной нами мультиплексной ПЦР-РВ использованы методы ПЦР с электрофоретической детекцией генов qacA/B и smr, предложенные ранее K.H. Lin и соавт. [18] и N. Noguchi и соавт. [19] соответственно. Результаты, полученные с помощью этих методов, также полностью совпали. Таким образом, специфичность разработанной нами мультиплексной ПЦР-РВ с флуоресцентно-мечеными зондами составила 100%.
Обсуждение
Выявление генов резистентности методами ПЦР является широко распространенным и доступным методом их мониторинга. Анализ источников литературы показал, что на сегодняшний день было предложено несколько методов детекции генов qacA/B и smr на основе ПЦР с электрофоретической детекцией результатов [9, 14, 21–23] и ПЦР-РВ с использованием интеркалирующих ДНК-красителей [18, 24], однако эти методы имеют ряд недостатков, связанных с длительностью тестирования и надёжностью результатов. Метод ПЦР с электрофоретической детекцией требует проведения гель-электрофореза, что увеличивает продолжительность анализа по сравнению с ПЦР-РВ и делает его трудоёмким и неудобным для проведения тестирования большого количества клинических изолятов. ПЦР с использованием интеркалирующих красителей позволяет проводить детекцию ампликонов в режиме реального времени, что сокращает продолжительность тестирования по сравнению с предыдущим методом, однако может давать ложноположительные результаты, поскольку интеркалирующие красители связываются со всеми двухцепочечными ДНК, включая димеры праймеров и возможные неспецифические продукты ПЦР, что требует тщательного подбора праймеров и соблюдения условий проведения ПЦР, включая используемые реактивы [25]. Нами была разработана мультиплексная ПЦР-РВ, которая лишена недостатков вышеупомянутых методов за счёт использования флуоресцентно-меченых зондов TaqMan, а также наличия внутреннего контроля амплификации.
Заключение
Разработана мультиплексная ПЦР-РВ с флуоресцентно-мечеными зондами для выявления генов qacA/B и smr грамположительных бактерий, которая отличается от существующих методов простотой и быстротой анализа, а также наличием внутреннего контроля амплификации, что позволяет исключить получение ложноотрицательных результатов тестирования. Разработанная мультиплексная ПЦР-РВ может быть использована для проведения мониторинга распространённости генов qacA/B и smr-устойчивости к КПАВ для выявления потенциально устойчивых к ним изолятов грамположительных бактерий. Однако следует учитывать, что все молекулярно-генетические тест-системы являются прогностическим инструментом и не исключают необходимости подтверждения фенотипа устойчивости микробиологическими методами.
1 Global antiseptics and disinfectants market size, share, trends & growth forecast report — segmented by type (alcohol and aldehyde, phenols and derivatives, biguanides and amides, quaternary ammonium compounds, iodine compounds and others), end user (domestic user and institutional user) and region — industry forecast from 2024 to 2029. URL: https://www.marketdataforecast.com/market-reports/antiseptics-and-disinfectants-market (дата обращения 23.10.2024).
2 Global antiseptics and disinfectants market size to exceed USD 79.25 Billion by 2033 | CAGR of 10.67%. URL: https://finance.yahoo.com/news/global-antiseptics-disinfectants-market-size-160000859.html (дата обращения 23.10.2024).
3 URL: https://www.ncbi.nlm.nih.gov/genbank
4 URL: https://blast.ncbi.nlm.nih.gov/Blast.cgi
5 URL: http://www.generunner.net
6 URL: https://www.thermofisher.com
7 URL: https://www.embl.org
8 URL: https://www.ddbj.nig.ac.jp
Об авторах
Светлана Николаевна Ковальчук
Институт системной биологии и медицины
Автор, ответственный за переписку.
Email: s.n.kovalchuk@mail.ru
ORCID iD: 0000-0002-5029-0750
к. б. н., с. н. с. лаб. преодоления микробной резистентности
Россия, МоскваАнна Леонидовна Архипова
Институт системной биологии и медицины
Email: s.n.kovalchuk@mail.ru
ORCID iD: 0000-0002-8835-6671
м. н. с. лаб. преодоления микробной резистентности
Россия, МоскваСветлана Юрьевна Ковылкова
Институт системной биологии и медицины
Email: s.n.kovalchuk@mail.ru
ORCID iD: 0009-0003-5668-7622
специалист лаб. преодоления микробной резистентности
Россия, МоскваЕлена Николаевна Ильина
Институт системной биологии и медицины
Email: s.n.kovalchuk@mail.ru
ORCID iD: 0000-0003-0130-5079
д. б. н., профессор РАН, член-корреспондент РАН, г.н.с., зав. лаб. математической биологии и биоинформатики
Россия, МоскваЛюдмила Самуиловна Федорова
Институт системной биологии и медицины
Email: s.n.kovalchuk@mail.ru
ORCID iD: 0000-0003-2663-0273
д. м. н., профессор, зав. лаб. преодоления микробной резистентности
Россия, МоскваСписок литературы
- Chaplin C.E. Bacterial resistance to quaternary ammonium disinfectants. J. Bacteriol. 1952;63(4):453–8. DOI: https://doi.org/10.1128/jb.63.4.453-458.1952
- Mc Carlie S., Boucher C.E., Bragg R.R. Molecular basis of bacterial disinfectant resistance. Drug. Resist. Updat. 2020;48:100672. DOI: https://doi.org/10.1016/j.drup.2019.100672
- Ковальчук С.Н., Федорова Л.С., Ильина Е.Н. Молекулярные механизмы микробной устойчивости к дезинфицирующим средствам. Антибиотики и химиотерапия. 2023;68(1-2): 45–56. Kovalchuk S.N., Fedorova L.S., Ilina E.N. Molecular mechanisms of microbial resistance to disinfectants. Antibiotics and Chemotherapy. 2023;68(1-2):45–56. DOI: https://doi.org/10.37489/0235-2990-2023-68-1-2-45-56 EDN: https://elibrary.ru/hycybo
- Merchel Piovesan Pereira B., Tagkopoulos I. Benzalkonium chlorides: uses, regulatory status, and microbial resistance. Appl. Environ. Microbiol. 2019;85(13):e00377–19. DOI: https://doi.org/10.1128/aem.00377-19
- Fox L.J., Kelly P.P., Humphreys G.J., et al. Assessing the risk of resistance to cationic biocides incorporating realism-based and biophysical approaches. J. Ind. Microbiol. Biotechnol. 2022; 49(1):kuab074. DOI: https://doi.org/10.1093/jimb/kuab074
- Благонравова А.С., Ковалишена О.В., Саперкин Н.В. Маркетинговое исследование госпитального сегмента регионального рынка средств дезинфекции в медицинских учреждениях. Медицинский альманах. 2011;(4):143–5. Blagonravova A.S., Kovalishena O.V., Saperkin N.V. Marketing examination of hospital segment of regional market of disinfectants in medical establishments. Medical Almanac. 2011;(4):143–5. EDN: https://elibrary.ru/nujlqz
- Тарасова Е.Ю., Тремасова А.М., Хузин Д.А. и др. Анализ рынка дезинфицирующих средств, используемых в отдельных животноводческих хозяйствах Приволжского федерального округа. Ветеринарный врач. 2022;(3):58–66. Tarasova E.Yu., Tremasova A.M., Khuzin D.A., et al. Analysis of the disinfectants market used in some livestock farms of the Volga federal district. Veterinarian. 2022;(3):58–66. DOI: https://doi.org/10.33632/1998-698X.2021_58_66 EDN: https://elibrary.ru/vfvanq
- Boyce J.M. Quaternary ammonium disinfectants and antiseptics: tolerance, resistance and potential impact on antibiotic resistance. Antimicrob. Resist. Infect. Control. 2023;12(1):32. DOI: https://doi.org/10.1186/s13756-023-01241-z
- Hernando-Amado S., Blanco P., Alcalde-Rico M., et al. Multidrug efflux pumps as main players in intrinsic and acquired resistance to antimicrobials. Drug. Resist Updat. 2016;28:13–27. DOI: https://doi.org/10.1016/j.drup.2016.06.007
- Hassan K.A., Liu Q., Elbourne L.D.H., et al. Pacing across the membrane: the novel PACE family of efflux pumps is widespread in Gram-negative pathogens. Res. Microbiol. 2018;169(7-8): 450–4. https://doi.org/10.1016/j.resmic.2018.01.001
- LaBreck P.T., Bochi-Layec A.C., Stanbro J., et al. Systematic analysis of efflux pump-mediated antiseptic resistance in Staphylococcus aureus suggests a need for greater antiseptic stewardship. mSphere. 2020;5(1):e00959–19. DOI: https://doi.org/10.1128/msphere.00959-19
- Chieffi D., Fanelli F., Fusco V. Antimicrobial and biocide resistance in Staphylococcus aureus: genomic features, decontamination strategies, and the role of S. aureus complex-related species, with a focus on ready-to-eat food and food-contact surfaces. Front. Food. Sci. Technol. 2023;3:1165871. DOI: https://doi.org/10.3389/frfst.2023.1165871
- Ignak S., Nakipoglu Y., Gurler B. Frequency of antiseptic resistance genes in clinical staphycocci and enterococci isolates in Turkey. Antimicrob. Resist. Infect. Control. 2017;6:88. DOI: https://doi.org/10.1186/s13756-017-0244-6
- El Sayed Zaki M., Bastawy S., Montasser K. Molecular study of resistance of Staphylococcus aureus to antiseptic quaternary ammonium compounds. J. Glob. Antimicrob. Resist. 2019;17:94–7. DOI: https://doi.org/10.1016/j.jgar.2018.11.022
- Ghasemzadeh-Moghaddam H., Azimian A., Bayani G., et al. High prevalence and expression of antiseptic resistance genes among infectious t037/ST239 methicillin-resistant Staphylococcus aureus (MRSA) strains in North Khorasan Province, Iran. Iran J. Basic Med. Sci. 2022;25(6):775–80. DOI: https://doi.org/10.22038/ijbms.2022.63780.14055
- Sommer L.M., Krauss J.L., Hultén K.G., et al. The prevalence of antiseptic tolerance genes among staphylococci and enterococci in a pediatric population. Infect. Control. Hosp. Epidemiol. 2019;40(3):333–40. DOI: https://doi.org/10.1017/ice.2019.3
- Юшкевич Е.А. Поиск генов QacA и QacB, опосредующих устойчивость к хлоргексидину, у штаммов рода Staphylococcus. В кн.: Алешковский И.А., Андриянов А.В., Антипов Е.А., ред. Материалы Международного молодежного научного форума «ЛОМОНОСОВ-2020». М.;2020. Yushkevich E.A. Search for QacA and QacB genes describing chlorhexidine resistance in a strain of the genus Staphylococcus. In: Aleshkovsky I.A., Andriyanov A.V., Antipov E.A., eds. Materials of the International Youth Scientific Forum «Lomonosov-2020». Moscow;2020.
- Lin K.H., Lin C.Y., Huang C.C., et al. Differentiation of qacA and qacB using high-resolution melt curve analysis, and both qacA and qacB but not qacC or norA types increase chlorhexidine minimal inhibitory concentrations in Staphylococcus aureus isolates. J. Microbiol. Immunol. Infect. 2020;53(6):900–8. DOI: https://doi.org/10.1016/j.jmii.2020.09.006
- Noguchi N., Suwa J., Narui K., et al. Susceptibilities to antiseptic agents and distribution of antiseptic-resistance genes qacA/B and smr of methicillin-resistant Staphylococcus aureus isolated in Asia during 1998 and 1999. J. Med. Microbiol. 2005;54 (Pt. 6):557–65. DOI: https://doi.org/10.1099/jmm.0.45902-0
- Herlemann D.P., Labrenz M., Jürgens K., et al. Transitions in bacterial communities along the 2000 km salinity gradient of the Baltic Sea. ISME J. 2011;5(10):1571–9. DOI: https://doi.org/10.1038/ismej.2011.41
- Smith K., Gemmell C.G., Hunter I.S. The association between biocide tolerance and the presence or absence of qac genes among hospital-acquired and community-acquired MRSA isolates. J. Antimicrob. Chemother. 2008;61(1):78–84. DOI: https://doi.org/10.1093/jac/dkm395
- Bjorland J., Steinum T., Kvitle B., et al. Widespread distribution of disinfectant resistance genes among staphylococci of bovine and caprine origin in Norway. J. Clin. Microbiol. 2005;43(9):4363–8. DOI: https://doi.org/10.1128/jcm.43.9.4363-4368.2005
- Alam M.M., Kobayashi N., Uehara N., Watanabe N. Analysis on distribution and genomic diversity of high-level antiseptic resistance genes qacA and qacB in human clinical isolates of Staphylococcus aureus. Microb. Drug Resist. 2003;9(2):109–21. DOI: https://doi.org/10.1089/107662903765826697
- Chan M.K.L., Koo S.H., Quek Q., et al. Development of a real-time assay to determine the frequency of qac genes in methicillin resistant Staphylococcus aureus. J. Microbiol. Methods. 2018;153:133–8. DOI: https://doi.org/10.1016/j.mimet.2018.09.017
- Law J.W., Ab Mutalib N.S., Chan K.G., Lee L.H. Rapid methods for the detection of foodborne bacterial pathogens: principles, applications, advantages and limitations. Front. Microbiol. 2015;5:770. DOI: https://doi.org/10.3389/fmicb.2014.00770
Дополнительные файлы