Анализ циркуляции коксакивируса А6 в субъектах Дальневосточного федерального округа Российской Федерации в 2014–2019 годах
- Авторы: Бутакова Л.В.1, Сапега Е.Ю.1, Троценко О.Е.1
-
Учреждения:
- Хабаровский научно-исследовательский институт эпидемиологии и микробиологии
- Выпуск: Том 100, № 5 (2023)
- Страницы: 358-368
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/18483
- DOI: https://doi.org/10.36233/0372-9311-366
- EDN: https://elibrary.ru/svsjaw
- ID: 18483
Цитировать
Полный текст
Аннотация
Введение. Молекулярно-эпидемиологический мониторинг за энтеровирусной инфекцией (ЭВИ) в субъектах Российской Федерации показал, что коксакивирус А6 (КВ-А6) на протяжении последних лет был одним из доминирующих типов энтеровирусов (ЭВ), циркулировавших среди населения страны, и в отдельные годы явился причиной большинства вспышек ЭВИ.
Цель работы — провести анализ циркуляции КВ-А6 в субъектах Дальневосточного федерального округа (ДФО) в 2014–2019 гг. с использованием молекулярно-генетических методов.
Материалы и методы. Биологический материал, поступавший из 9 субъектов ДФО, исследовали методом полимеразной цепной реакции с обратной транскрипцией для обнаружения РНК ЭВ. Проводили амплификацию положительных образцов для получения нуклеотидных последовательностей фрагментов генов VP1 и VP2 для дальнейшего установления типа ЭВ. Для молекулярно-генетического анализа дальневосточных штаммов КВ-А6 получены нуклеотидные последовательности фрагментов генов VP1 и 3Dpol. Построение филогенетических деревьев осуществляли с использованием Байесовых филогенетических методов.
Результаты. C 2014 по 2019 г. получены 1773 нуклеотидные последовательности неполиомиелитных ЭВ 43 типов, циркулировавших в ДФО, при этом основная часть сиквенсов принадлежала КВ-А6 (524; 29,5%). В годы наибольшей идентификации КВ-А6 в субъектах ДФО наблюдались подъёмы заболеваемости ЭВИ с регистрацией вспышечных очагов. Из клинических проявлений ЭВИ, вызванных КВ-А6, в ДФО преобладали герпангина и экзантемные формы. Филогенетический анализ показал принадлежность дальневосточных штаммов КВ-А6 к превалирующему во всём мире субгенотипу D3, а также циркуляцию в анализируемый период времени нескольких рекомбинантных форм КВ-А6 (RF-A, -H, -L, -N, -R).
Заключение. Установленное генетическое разнообразие штаммов КВ-А6, циркулировавших в субъектах ДФО в 2014–2019 гг., требует дальнейшего изучения для получения новых знаний о молекулярной эпидемиологии КВ-А6 и совершенствования системы эпидемиологического надзора за ЭВИ.
Полный текст
Введение
Патогенными для человека являются более 100 типов энтеровирусов (ЭВ), относящихся к видам Enterovirus А–Enterovirus D рода Enterovirus, семейства Picornaviridae. Чаще всего энтеровирусная инфекция (ЭВИ) имеет бессимптомное или маловыраженное течение, в то же время известны более 20 клинических синдромов, вызываемых ЭВ: асептический менингит, герпангина, везикулярный стоматит с экзантемой, энцефалит, острые заболевания дыхательных путей, острый вялый паралич, миокардит, гастроэнтерит, конъюнктивит, увеит, плевродиния, неонатальный сепсис и др. [1]. К наиболее эпидемически значимым формам ЭВИ можно отнести энтеровирусный менингит и везикулярный стоматит с экзантемой (болезнь «кисть-стопа-рот», БКСР), т.к. с ними связано большинство регистрируемых в мире вспышек ЭВИ [2].
БКСР характеризуется поражением слизистой ротоглотки, экзантемой на коже верхних и нижних конечностей, а также общими симптомами в виде лихорадки, боли в горле, недомогания. Болеют преимущественно дети до 10 лет, хотя случаи заболевания регистрируются и у взрослых. При тяжёлом течении у ряда пациентов возможны развитие энцефалита, острого вялого паралича, нейрогенного отёка лёгких, миокардита, наступление смерти [3, 4].
Вызывать БКСР могут более 20 типов ЭВ, среди которых наиболее часто выявляют ЭВ А71, коксакивирусы (КВ) А16, А6, А10, А2, А4, А9, В2, В5 [5–7]. При этом ЭВ-А71 и КВ-А16 до недавнего времени были основными возбудителями крупных вспышек БКСР, особенно в странах Западно-Тихоокеанского региона [8, 9]. В частности, множество случаев данного заболевания с тяжёлыми осложнениями и высоким уровнем летальности зафиксировано на территории материкового Китая [10, 11]. Однако после вспышки БКСР в 2008 г. в Финляндии новый генетический вариант КВ-А6 распространился по всему миру, став одним из ведущих патогенов при последовавших вспышках БКСР в разных странах [12].
Характерная морфология и локализация сыпи при БКСР подробно описаны в 1958 г. [13]. В отличие от других ЭВ — возбудителей БКСР, КВ-А6 часто вызывает более тяжёлые и обширные, нередко болезненные поражения кожных покровов. Экзантема у пациентов может быть не только макулопапулёзной или везикулёзной, но также в виде булл, эрозий, язв, геморрагий, имитируя некоторые дерматологические заболевания (мультиформная эритема, синдром Стивенса–Джонсона, синдром Джанотти–Крости). У лиц, страдающих атопическим дерматитом, наблюдались герпесоподобные высыпания, получившие название eczema coxsackium [14–16]. Распространённым проявлением КВ-А6-инфекции является онихомадезис [17].
Постоянный молекулярно-эпидемиологический мониторинг за ЭВИ в субъектах Российской Федерации показал, что КВ-А6 в течение ряда лет был одним из доминирующих типов ЭВ, циркулировавших среди населения страны, и в отдельные годы детерминировал большинство вспышек ЭВИ. Так, в 2014 г. КВ-А6 идентифицирован более чем в 60% этиологически расшифрованных очагов групповой заболеваемости ЭВИ, а в 2017 г. — в 42 очагах [18, 19]. Кроме того, в 2017 г. среди российских граждан, находившихся на отдыхе в Турции, Вьетнаме, Тунисе, отмечены случаи экзантемных форм ЭВИ, в том числе БКСР, обусловленных КВ-А6 [20].
Цель настоящего исследования — провести анализ циркуляции КВ-А6 в субъектах Дальневосточного федерального округа (ДФО) с 2014 по 2019 г. с использованием молекулярно-генетических методов.
Материалы и методы
Для анализа заболеваемости ЭВИ в субъектах ДФО в 2014–2019 гг. использованы данные форм государственного статистического наблюдения № 1 и № 2 «Сведения об инфекционных и паразитарных заболеваниях», № 23–09 «Сведения о вспышках инфекционных заболеваний», материалы Управлений Роспотребнадзора и Центров гигиены и эпидемиологии субъектов ДФО.
Биологический материал (фекальные суспензии, носоглоточные мазки и смывы, ликвор, смывы из везикул от лиц с подозрением на ЭВИ; культуры клеток; образцы из объектов окружающей среды) для исследования молекулярно-генетическими методами с 2014 по 2019 г. поступал в лабораторию Дальневосточного регионального научно-методического центра по изучению ЭВИ Хабаровского НИИ эпидемиологии и микробиологии Роспотребнадзора из 9 субъектов ДФО: Хабаровский, Приморский, Камчатский края, Сахалинская, Амурская, Магаданская области, Еврейская автономная область (ЕАО), Республика Саха (Якутия), Чукотский автономный округ (ЧАО). Протокол исследования одобрен Этическим комитетом Хабаровского НИИ эпидемиологии и микробиологии (протокол № 2 от 14.01.2014).
РНК ЭВ в образцах определяли методом полимеразной цепной реакции (ПЦР) с обратной транскрипцией с помощью набора реагентов «АмплиСенс Enterovirus-FL» (ЦНИИ Эпидемиологии Роспотребнадзора). Для положительных образцов дополнительно проводили обратную транскрипцию с использованием набора реагентов «Реверта L» (ЦНИИ Эпидемиологии Роспотребнадзора) для получения из РНК кДНК, необходимой в качестве матрицы. Для амплификации используемых для типирования ЭВ коротких фрагментов генов VP1 и VP2 применяли ранее предложенные праймеры и программы [21, 22]. Получившиеся продукты ПЦР анализировали с помощью электрофореза в 2% агарозном геле, окрашенном бромидом этидия. Далее проводили очистку ПЦР-ампликонов реагентами «QIAGEN» и «Диа-М», мечение ампликонов с помощью набора «BigDye Terminator v. 3.1 Cycle Sequencing Kit» («Thermo Fisher Scientific»). Очищали продукты секвенирующей реакции и проводили секвенирование на автоматическом генетическом анализаторе «ABI 3500 Genetic Analyzer» («Applied Biosystems»). Тип ЭВ устанавливали, сравнивая полученные нуклеотидные последовательности с референсными, представленными в базе данных GenBank, при помощи поискового алгоритма BLAST.
Для молекулярно-генетического анализа дальневосточных КВ-А6 с помощью пар праймеров 2347s/3326a и 2407s/3296a получены 78 нуклеотидных последовательностей фрагмента капсидного белка VP1 длиной 774 нуклеотидных основания (н.о.) [23]. В результате анализа полногеномных последовательностей КВ-А6, представленных в GenBank, были подобраны праймеры для амплификации фрагмента гена полимеразы 3Dpol (753 н.о.).
Для формирования выборки референсных сиквенсов из GenBank были взяты доступные на момент проведения исследования (октябрь 2022 г.) 3142 нуклеотидные последовательности гена VP1 длиной 915 н.о. и 737 полногеномных последовательностей КВ-А6. После проведения выравнивания в «BioEdit v. 7.2.5» были исключены последовательности, имеющие ошибки секвенирования и отличающиеся друг от друга менее чем на 5%, как рекомендуется в публикации [24].
Построение филогенетических деревьев проводили с использованием Байесовых филогенетических методов, представленных в программном обеспечении «BEAST v. 1.8.4». Статистическая достоверность филогенетического дерева подтверждена в программе «Tracer v. 1.6» (эффективный размер выборки более 200). Суммирование выборки филогенетических деревьев, созданной «BEAST v. 1.8.4», в дерево с максимальным доверием к кладам осуществляли с помощью «TreeAnnotator v. 1.8.4» с дальнейшей визуализацией в программе «FigTree v. 1.4.4».
Результаты
Всего за 2014–2019 гг. исследованы 3737 образцов биологического материала из 9 субъектов ДФО. Методом секвенирования по Сэнгеру получены 1773 нуклеотидных последовательности неполиомиелитных ЭВ 43 типов, при этом бóльшая часть сиквенсов принадлежала КВ-А6 (524; 29,5 ± 1,1%). В основном КВ-А6 определяли из проб, собранных от людей. При исследовании положительных образцов (n = 85) из объектов окружающей среды КВ-А6 установлен только в 2 случаях. Вероятно, это было связано как с недостатками организации проведения мониторинга за циркуляцией полио- и неполиомиелитных ЭВ в объектах окружающей среды в некоторых субъектах округа, что привело в целом к низкому уровню выявления ЭВ в сточной воде с помощью ПЦР, так и со слабой идентификацией представителей КВ группы А при использовании классического вирусологического метода [25].
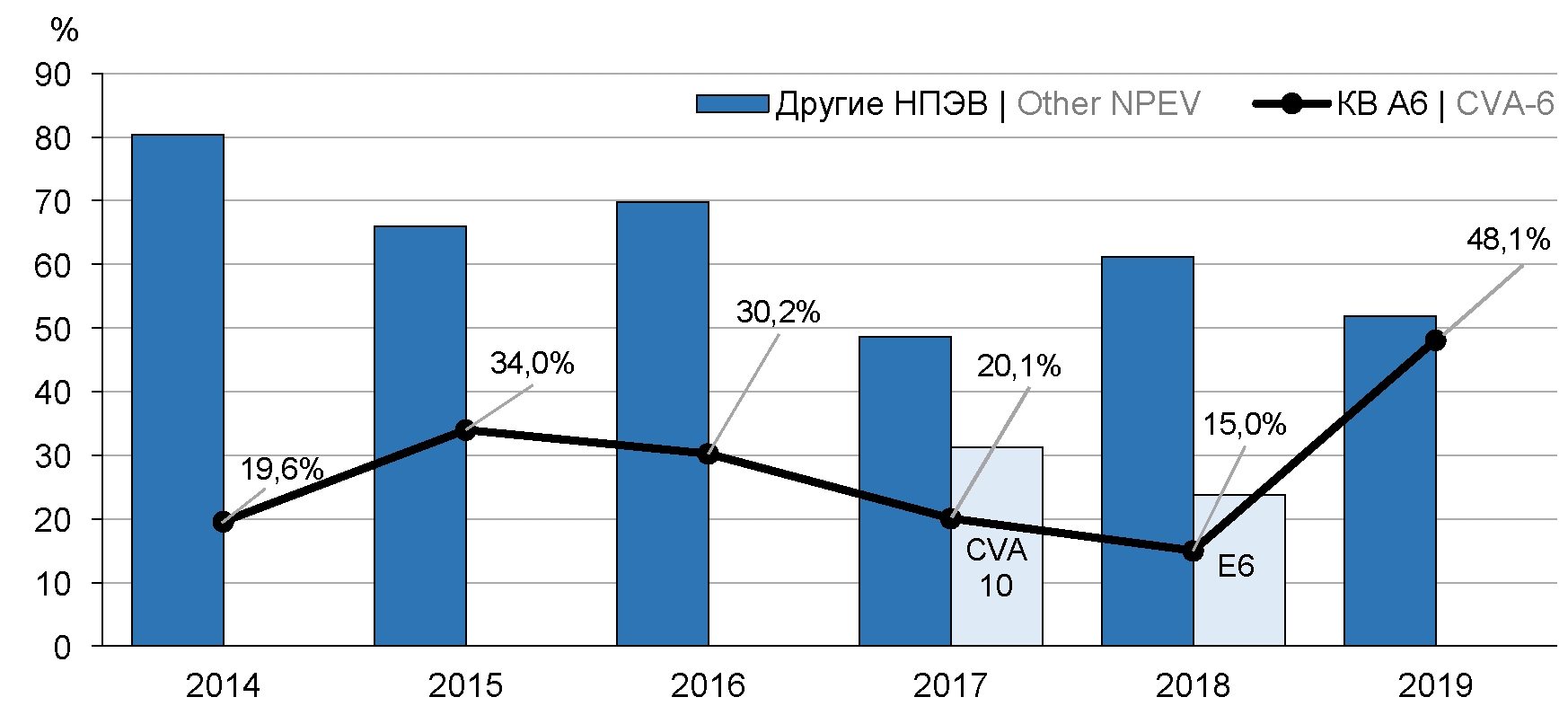
С 2014 по 2016 г. КВ-А6 выявляли чаще других ЭВ в целом по ДФО (от 19,6 до 34,0% от общего количества неполиомиелитных ЭВ), в 2017 г. лидером по числу обнаружений был КВ-А10 (31,3%), в 2018 г. — ECHO 6 (23,8%), в 2019 г. снова отмечено преобладание КВ-А6 (рис. 1).
Рис. 1. Динамика обнаружения КВ-А6 в ДФО в 2014–2019 гг.
На протяжении всего анализируемого периода КВ-А6 ежегодно идентифицировали в образцах, направляемых на исследование из Хабаровского края и ЕАО. В остальных субъектах ДФО наблюдалось неравномерное выделение данного ЭВ (табл. 1). При этом нельзя уверенно говорить о том, что КВ-А6 совсем не циркулировал среди населения этих территорий. К вероятным причинам таких «провалов» в обнаружении КВ-А6 можно отнести:
Таблица 1. Выявление КВ-А6 в субъектах ДФО в 2014–2019 гг.
Table 1. Detection of coxsackievirus A6 in the territories of the Far Eastern Federal District in 2014–2019
Субъекты ДФО Federal subjects of the FEFD | Количество выявленных КВ-А6 Number of detected CVA-6 | Всего КВ-А6/ неполиомиелитных ЭВ Total CVA-6/Total NPEV | |||||
2014 | 2015 | 2016 | 2017 | 2018 | 2019 | ||
Хабаровский край | Khabarovsk Krai | 26 | 57 | 11 | 10 | 12 | 73 | 189/652 |
ЕАО | Jewish Autonomous Region | 2 | 35 | 4 | 1 | 2 | 14 | 58/270 |
Амурская область | Amur Region | – | 6 | 7 | 6 | 13 | 7 | 39/93 |
Приморский край | Primorsky Krai | – | – | 7 | 2 | 2 | 82 | 93/257 |
Сахалинская область | Sakhalin Region | 2 | 6 | 28 | 9 | – | 8 | 53/174 |
Республика Саха (Якутия) The Republic of Sakha (Yakutia) | 23 | – | 1 | 9 | 5 | 5 | 43/198 |
Магаданская область | Magadan Region | – | – | 23 | – | – | – | 23/69 |
Камчатский край | Kamchatka Krai | 2 | – | – | 10 | – | 2 | 14/45 |
ЧАО | Chukotka Autonomous Okrug | – | – | – | 12 | – | – | 12/15 |
- малое количество проб биологического материала, поступавших в региональный центр для молекулярного типирования из субъектов в отдельные годы либо вовсе их отсутствие, что, в свою очередь, повлияло и на определение спектра других неполиомиелитных ЭВ;
- недостаточную диагностику и регистрацию случаев ЭВИ;
- циркуляцию вариантов вируса, к которым имеется коллективный иммунитет;
- географическое положение, климатические и экономические особенности некоторых субъектов ДФО, определяющие интенсивность внешних и внутренних миграционных потоков, способствующих распространению вирусов.
Тем не менее в годы наибольшего обнаружения КВ-А6 в субъектах ДФО также регистрировались подъёмы заболеваемости ЭВИ, что позволяет предположить появление новых вариантов КВ-А6 на территории округа с вовлечением в эпидемический процесс неиммунного населения. Так, в 2014 г. в Республике Саха (Якутия) наблюдался рост заболеваемости ЭВИ по сравнению с предыдущим годом в 2,4 раза (2013 г. — 8,7 на 100 тыс. населения; 2014 г. — 21,1 на 100 тыс. населения). В 2015 г. темпы прироста заболеваемости ЭВИ в Хабаровском крае (105,9 на 100 тыс. населения) и ЕАО (64,1 на 100 тыс. населения) относительно 2014 г. составили 35,4 и 67,8% соответственно. В Сахалинской области в 2016 г. заболеваемость ЭВИ выросла в 4,5 раза (в 2015 г. — 22,7 на 100 тыс. населения; в 2016 г. — 103,0 на 100 тыс. населения), а в Магаданской области — в 2,0 раза (в 2015 г. — 15,9 на 100 тыс. населения; в 2016 г. — 31,7 на 100 тыс. населения). В 2017 г. увеличение заболеваемости ЭВИ в 2,6 раза отмечено в Камчатском крае (в 2016 г. — 8,8 на 100 тыс. населения; в 2017 г. — 23,2 на 100 тыс. населения). Кроме того, интродукция в 2017 г. КВ-А6 на территорию ЧАО, где ранее не было регистрации случаев ЭВИ, привела к формированию очага вспышечной заболеваемости. В Амурской области в 2018 г. темп прироста ЭВИ составил 40,5% (в 2017 г. — 14,8 на 100 тыс. населения; в 2018 г. — 20,8 на 100 тыс. населения). В 2019 г. рост заболеваемости ЭВИ наблюдался в 3,8 раза в Приморском крае (в 2018 г. — 7,5 на 100 тыс. населения; в 2019 г. — 28,7 на 100 тыс. населения), а в ЕАО (в 2018 г. — 27,8 на 100 тыс. населения; в 2019 г. — 58,7 на 100 тыс. населения) и Хабаровском крае (в 2018 г. — 58,6 на 100 тыс. населения; в 2019 г. — 62,1 на 100 тыс. населения) — в 2,1 и 1,1 раза соответственно.
Кроме того, в 2014–2019 гг. в ДФО зарегистрированы 15 очагов групповой заболеваемости ЭВИ, вызванных КВ-А6, среди детей дошкольного возраста:
- в Хабаровском крае в 2015–2016, 2019 гг. — 7 очагов, 89 пострадавших с клиническими проявлениями в виде герпангины, экзантемы, БКСР;
- в Амурской области в 2016 и 2018 гг. — 3 очага, 25 пострадавших с клиническими проявлениями в виде БКСР, экзантемы, катаральной формы;
- в Сахалинской области в 2016–2017 гг. — 2 очага, 15 пострадавших 15 с клиническими проявлениями в виде БКСР, экзантемы, герпангины;
- в ЧАО в 2017 г. — 1 очаг, 18 пострадавших с клиникой БКСР;
- в Приморском крае в 2019 г. — 2 очага, 19 пострадавших, с клиническими проявлениями в виде экзантемы.
В 2 субъектах ДФО выявлены вероятные завозные случаи ЭВИ в форме БКСР, вызванные КВ-А6. В Хабаровском крае в 2015 г. инфекция обнаружена у 2 детей из одной семьи, прибывшей из Италии, в 2019 г. — у побывавшего в Таиланде взрослого. В Амурской области 3 года подряд (с 2016 по 2018 г.) регистрировались внутрисемейные случаи ЭВИ, развившейся в ближайшее время после возвращения из путешествия во Вьетнам.
В целом среди заболевших ЭВИ, у которых идентифицирован КВ-А6, преобладали лица мужского пола (62,4%), что сопоставимо с данными других исследователей [26]. В возрастной структуре пациентов с выявленным КВ-А6 дети до 3 лет составили 60,0%, 3–6 лет — 29,2%, 7–14 лет — 6,8%, лица старше 18 лет — 4,0%. Не всегда в направлениях на исследование были указаны формы ЭВИ, однако среди установленных клинических проявлений КВ-А6-инфекции (226 человек) у населения ДФО чаще встречались герпангина (41,2%) и экзантемные (включая БКСР) формы (36,3%). Также отмечены катаральная (15,5%) и кишечная формы (7,0%). Нетипичный характер экзантемы наблюдался у пострадавших из вспышечного очага ЭВИ в ЧАО в 2017 г., где, помимо макулопапулезных высыпаний, у детей выявлены геморрагические элементы.
Филогенетический анализ нуклеотидных последовательностей фрагмента гена VP1 (n = 78) показал, что все циркулировавшие в ДФО в 2014–2019 гг. КВ-А6 вошли в одну большую группу вместе со штаммами, выявленными в западных регионах России, странах Европы и Азиатско-Тихоокеанского региона (рис. 2).
Рис. 2. Филогенетическое дерево с максимальным доверием к кладам, построенное на основании анализа фрагментов капсидного белка VP1 КВ-А6 (774 н.о.). Чёрным цветом выделены дальневосточные штаммы КВ-А6: KHV — Хабаровский край; JAR — ЕАО; AMUR — Амурская область; PRIM — Приморский край; YKT — Республика Саха (Якутия); SAH — Сахалинская область; KAM — Камчатский край; MGD — Магаданская область; CHAO — ЧАО.
В настоящее время ряд зарубежных авторов придерживается разделения штаммов КВ-А6 на геногруппы А, В, С, D, Е на основе филогенетического анализа полных нуклеотидных последовательностей гена VP1 (915 н.о.). При этом различие между последовательностями, относящимися к разным геногруппам, составляет не менее 15%. В свою очередь среди геногруппы D выделяют субгенотипы D1–D3. К глобально доминирующему субгенотипу D3 принадлежат штаммы КВ-А6, ответственные за большинство вспышек БКСР во всём мире за последние 15 лет [27–29]. Основываясь на этой классификации, дальневосточные штаммы КВ-А6 2014–2019 гг. также относятся к субгенотипу D3.
Расчет p-дистанций для нуклеотидных последовательностей фрагмента гена VP1 (774 н.о.), полученных в ходе нашего исследования и взятых в качестве референсных из GenBank, показал, что среднее расстояние между геногруппами КВ-А6 составило 17,1%, между субгенотипами геногруппы D — 9,6%.
С помощью Байесовского коалесцентного анализа установлено, что самый недавний общий предок для КВ-А6 существовал 77 лет назад (95% ДИ 70–88 лет). Средняя скорость накопления замен составила 4,97 × 10–3 замен/сайт/год (95% ДИ 4,84–5,11 × 10–3 замен/сайт/год), что сходно с другими данными: 4,2 × 10–3 (95% ДИ 2,8–5,8) замен/сайт/год [23] и 4,71 × 10–3 (95% ДИ 4,04–5,46) замен/сайт/год [30].
Внутри географически разнообразного субгенотипа D3 дальневосточные КВ-А6 оказались разделены на три подгруппы, при этом отличие между штаммами разных подгрупп составило в среднем 7,2%.
Большинство полученных нами для филогенетического анализа КВ-А6 (n = 38; 48,7%) вошли в подгруппу D3.3 и в основном группировались со штаммами, выделенными в Китае, Вьетнаме, Японии. Скорее всего, штаммы D3.3 произошли от КВ-А6, циркулировавших на Тайване в 2009–2010 гг., и имели общего предка примерно в 2008 г. (95% ДИ 2008–2009). Дальневосточные штаммы D3.3 идентифицированы в 8 субъектах (исключение — ЧАО) и определены в течение всего анализируемого периода в целом по ДФО.
Во второй подгруппе, D3.2, дальневосточные КВ-А6 (n = 22; 28,2%) были наиболее тесно связаны с вирусами, обнаруженными во Франции, других регионах России, Китае, Японии. Вероятным временем существования последнего общего предка для второй подгруппы определён 2006 г. (95% ДИ 2005–2007). Вирусы субклады D3.2 выявлены в разных субъектах ДФО с 2015 по 2019 г.
КВ-А6 подгруппы D3.1 (n = 18; 23,1%) достоверно кластеризовались с французскими и российскими штаммами и были идентифицированы в 5 субъектах (Хабаровский и Приморский края, ЕАО, Сахалинская и Амурская области) с 2014 по 2017 г. и в 2019 г. Общий предок для D3.1 циркулировал приблизительно в 2005 г. (95% ДИ 2005–2007).
Секвенированный нами архивный изолят КВ-А6 (7224_KHV_2010) из Хабаровского края 2010 г. на филограмме сгруппировался со штаммами, входящими в субгенотип D2 (на рис. 2 не показан). Учитывая, что с 2014 г. КВ-А6 субгенотипа D2 на Дальнем Востоке не выявлены, можно допустить, что смена субгенотипов произошла между 2010 и 2014 гг.
Предложенная для ЭВ дифференциация рекомбинантных форм ECHO30 (Е30) используется и для КВ-А6 [31]. На основании филогенетического анализа нуклеотидных последовательностей фрагмента гена 3Dpol (753 н.о.) КВ-А6, циркулировавшие в 2014–2019 гг. в субъектах ДФО, отнесены к 5 рекомбинантным формам: A, H, L, N и R (рис. 3). Большинство полученных нами сиквенсов составило рекомбинантную форму А (67,9%), второй по частоте оказалась рекомбинантная форма H (23,1%). Штамм 7224_KHV_2010 кластеризовался со штаммами рекомбинантной формы Е.
Рис. 3. Филогенетическое дерево, построенное на основании анализа фрагментов гена полимеразы 3Dpol КВ-А6 (753 н.о.). Цветом выделены дальневосточные штаммы КВ-А6. Прописными латинскими буквами справа указаны рекомбинантные формы КВ-А6 (от RF-A до RF-P).
Среди дальневосточных штаммов КВ-А6, относящихся к преобладающей подгруппе D3.3, идентифицированы 3 рекомбинантные формы. КВ-А6 варианта D3.3/A (n = 34) сохранили ту же кластеризацию, что и на филогенетическом дереве VP1, выявлены в 2014 г. в Хабаровском крае, Сахалинской области и Республике Саха (Якутия); в 2015 г. — в Сахалинской области; в 2016 г. — в Хабаровском и Приморском краях, Амурской и Сахалинской областях; в 2017 г. — в Хабаровском крае, Приморье и ЕАО; в 2018 г. — в Хабаровском и Приморском краях, Амурской области и Республике Саха (Якутия); в 2019 г. — в Хабаровском, Приморском, Камчатском краях, ЕАО, Амурской и Сахалинской областях, Республике Саха (Якутия). КВ-А6 варианта D3.3/L обнаружены в 2016 г. в Приморском крае и Магаданской области, D3.3/R — в 2017 г. в Камчатском крае.
Топология КВ-А6 из подгруппы D3.2 на дереве для последовательностей 3Dpol отличается от таковой в VP1, две рекомбинантные формы D3.2 (H и N) сформировали обособленные кластеры. КВ-А6 варианта D3.2/H (n = 18) в 2014–2015 гг. не выявлены; в 2016 г. циркулировали в Приморском крае, Магаданской области и Республике Саха (Якутия); в 2017 г. — в Амурской области, Республике Саха (Якутия), Камчатском крае и ЧАО; в 2018 г. — в Хабаровском, Приморском краях и ЕАО; в 2019 г. — в Республике Саха (Якутия), ЕАО и Амурской областях. Вариант D3.2/N обнаружен в Приморском крае в 2017 г. и Сахалинской области в 2019 г. Два штамма D3.2/A из Хабаровского края (2015 г.) и Амурской области (2018 г.) сгруппировались на дереве 3Dpol вместе со штаммами D3.3/A.
Несоответствие филогенетической группировки последовательностей 3Dpol КВ-А6 вариантов D3.2/A, D3.2/H, D3.3/L, D3.2/N и D3.3/L по сравнению с VP1 указывает на то, что эти штаммы возникли вследствие событий рекомбинации [32, 33].
Все 18 штаммов КВ-А6 подгруппы D3.1 относятся к рекомбинантной форме А и также заняли то же положение, что и на дереве VP1. Вариант КВ-А6 D3.1/A не идентифицирован в Республике Саха (Якутия), Магаданской области, Камчатском крае и ЧАО.
Следует отметить, что в одном и том же субъекте могли одновременно циркулировать разные варианты КВ-А6. Наибольшее разнообразие выявлено в Приморском крае в 2016 г. (4 варианта: D3.3/A, D3.3/L, D3.2/H и D3.1/A) и в ЕАО в 2019 г. (3 варианта: D3.3/A, D3.2/H и D3.1/A).
Обсуждение
Проведённое нами исследование показало генетическое разнообразие популяции КВ-А6 в субъектах ДФО в 2014–2019 гг. После 2008 г. КВ-А6 постепенно вытеснил ЭВ А71 и КВ-А16 с позиции доминирующих возбудителей при вспышках БКСР. Произошедшая с конца 2012 г. смена доминирующего субгенотипа КВ-6, отмеченная в нескольких регионах материкового Китая, привела к значительному росту заболеваемости и увеличению числа вспышек БКСР [34, 35]. Наличие протяжённой сухопутной (проходит через 4 субъекта ДФО: Хабаровский край, ЕАО и Амурская области, Приморский край) и водной российско-китайской границы, обеспечивающей тесные социально-экономические и туристические связи между государствами, несомненно способствовало распространению субгенотипа D3 КВ-А6 на Дальнем Востоке. Однако на филогенетическом дереве VP1 дальневосточные штаммы КВ-А6, выделенные в один год из одного субъекта, группировались не только с вирусами, выделенными в Китае и прочих странах Азии, но и из европейских стран и других регионов России, что свидетельствует о независимых путях заноса разных вариантов КВ-А6 в ДФО, которым содействует внешняя и внутренняя миграция населения.
Нам не удалось выявить быстрой смены и преобладания тех или иных вариантов КВ-А6 на территории ДФО в анализируемый период времени, что, например, характерно для эпидемиологического профиля ЭВ ECHO30 [36, 37]. Возможно, это связано как с ограничениями, наложенными небольшим количеством изученных последовательностей из каждого субъекта или их отсутствием за определённый год, так и с тем, что оборот линий КВ-А6 может происходить не так часто, как у E30, для которого установлены 3–5-летние циклы циркуляции отдельных вариантов [31].
В большинстве тех субъектов ДФО, где впервые были идентифицированы потенциальные рекомбинанты D3.2/A, D3.2/H, D3.3/L и D3.3/R, в тот же год наблюдался рост ЭВИ, что подтверждает то, что появление нового вируса в неиммунной популяции вызывает ухудшение эпидемической ситуации. Коциркуляция нескольких вариантов КВ-А6 на одной территории тоже определённо внесла вклад в уровень заболеваемости ЭВИ, однако не стоит исключать и влияния других типов ЭВ. Так, несмотря на то что в Приморском крае в 2016 г. одновременно циркулировали 4 варианта КВ-А6, с чем можно было бы связать рост регистрации случаев ЭВИ в 5,8 раза по сравнению с предыдущим годом, среди всех идентифицированных в этот период неполиомиелитных ЭВ преобладал Е30. В то же время в ЕАО в 2019 г. отмечены и подъём заболеваемости ЭВИ с доминированием КВ-А6 в спектре обнаруженных неполиомиелитных ЭВ, и совместная циркуляция 3 различных линий вируса. В связи с этим представляется обоснованным, что на проявления эпидемического процесса КВ-А6-инфекции в субъектах ДФО в 2014–2019 гг. всё же в достаточной мере воздействовал уровень популяционного иммунитета, на что указывает и тот факт, что основными выделителями КВ-А6 являлись дети до 3 лет.
Популяции ЭВ имеют квазивидовую структуру за счёт высокой скорости мутаций и рекомбинаций, которые являются следствием молекулярных механизмов, свойственных процессу репликации вирусной РНК [38]. Фрагменты генома ЭВ эволюционируют независимо друг от друга даже на уровне микроэволюции, обеспечивая ЭВ существование в виде всемирного высокодинамичного пула генетической информации [39, 40]. Частая и свободная перетасовка фрагментов между вирусными геномами может потенциально привести к появлению новых геновариантов ЭВ с изменёнными патогенетическими свойствами, повышенной вирулентностью и трансмиссивностью [40–42].
До 2008 г. в Европе и 2009 г. в США случаи ЭВИ, связанные с КВ-А6, регистрировались достаточно редко, а в Азии до 2009 г. КВ-А6 вызывал вспышки герпангины [12, 43, 44]. Однако события рекомбинации в геноме КВ-А6, предшествовавшие серии крупных вспышек с 2008 г., не только привели к смене основной клинической формы КВ-А6-инфекции на БКСР и появлению атипичной симптоматики этого заболевания, но и, возможно, к большей трансмиссивности вируса. Учитывая, что уже около 10 лет в мире выявляют только субгенотип D3 КВ-А6 [28, 29], эволюция и появление множественных линий вируса прежде всего связаны с рекомбинацией неструктурных областей генома.
Исследование S.H. Wang и соавт. показало, что белок 2С может играть важную роль в патогенности штаммов КВ-А6, а генетические изменения, затрагивающие эту область, могут оказывать влияние на исходы КА6-инфекции [45]. Сопоставление клинических данных и молекулярного анализа КВ-А6, обнаруженных во время вспышки 2012–2013 гг. в Шанхае (КНР), выявило, что штаммы, относящиеся к рекомбинантной форме J, вызывали более обширное поражение кожных покровов, чем штаммы линии А [35].
В работе E. Gaunt и соавт. показана связь между возникновением атипичной экзантемы в виде обширных герпесоподобных высыпаний, диагностированных во время вспышки БКСР в Эдинбурге (Великобритания) в 2014 г., и установленной у заболевших рекомбинантной формы H КВ-А6 [23]. Следует отметить, что штаммы КВ-А6, идентифицированные нами при расследовании вспышки БКСР в ЧАО в 2017 г. и вызвавшие появление геморрагической сыпи у детей, также принадлежали линии H. Эти наблюдения показывают, что события рекомбинации в неструктурных областях генома КВ-А6 могут способствовать появлению вариантов вируса, вызывающих различные клинические фенотипы и более тяжёлое течение КВ-А6-инфекции. В связи с этим выявление взаимосвязей между рекомбинацией и изменениями патогенетических свойств вируса имеет важное значение и подчёркивает необходимость дальнейшего изучения.
Учитывая генетическую изменчивость ЭВ и риск возникновения вариантов с высоким патогенным и эпидемическим потенциалом, постоянный надзор за ЭВ является обязательным инструментом, направленным на своевременное выявление и отслеживание таких штаммов, быструю оценку эпидемической ситуации и принятие адекватных управленческих решений [24]. Важность непрерывного надзора также определяется тем, что интродукция и циркуляция «нового» вируса в популяции может быть незаметной довольно длительное время, пока не появятся критические изменения в геноме, способные привести к вспышке. Так, расследование вспышки менингоэнцефалита, произошедшей среди детей в Ростове-на-Дону в 2013 г. и вызванной ЭВ А71, показало опасность скрытой циркуляции на территории России занесённого, предположительно, из Китая вируса в течение нескольких лет до её возникновения [46].
Рекомбинация играет ключевую роль в эволюции ЭВ и других представителей семейства Picornaviridae, обеспечивая не только их разнообразие, но и стабильное существование в качестве глобального генофонда [47]. Появление множественных геновариантов КВ-А6 свидетельствует о том, что рекомбинация является одним из важнейших механизмов адаптации вируса и сохранения его патогенности в человеческой популяции.
Планируемое продолжение исследования может помочь внести ясность в особенности молекулярной эпидемиологии КВ-А6 в отдельных субъектах ДФО. В свою очередь, полученные знания будут способствовать совершенствованию эпидемиологического надзора за ЭВИ.
Об авторах
Людмила Васильевна Бутакова
Хабаровский научно-исследовательский институт эпидемиологии и микробиологии
Автор, ответственный за переписку.
Email: evi.khv@mail.ru
ORCID iD: 0000-0002-7238-3691
н.с. Дальневосточного регионального научно-методического центра по изучению энтеровирусных инфекций Хабаровского НИИ эпидемиологии и микробиологии, Хабаровск
Россия, ХабаровскЕлена Юрьевна Сапега
Хабаровский научно-исследовательский институт эпидемиологии и микробиологии
Email: evi.khv@mail.ru
ORCID iD: 0000-0003-4438-6913
к.м.н., в.н.с., рук. Дальневосточного регионального научно-методического центра по изучению энтеровирусных инфекций Хабаровского НИИ эпидемиологии и микробиологии
Россия, ХабаровскОльга Евгеньевна Троценко
Хабаровский научно-исследовательский институт эпидемиологии и микробиологии
Email: evi.khv@mail.ru
ORCID iD: 0000-0003-3050-4472
д.м.н., директор Хабаровского НИИ эпидемиологии и микробиологии
Россия, ХабаровскСписок литературы
- Tapparel C., Siegrist F., Petty T.J., Kaiser L. Picornavirus and enterovirus diversity with associated human diseases. Infect. Genet. Evol. 2013;14:282–93. DOI: https://doi.org/10.1016/j.meegid.2012.10.016
- Gonzalez G., Carr M.J., Kobayashi M., et al. Enterovirus-associated hand-foot and mouth disease and neurological complications in Japan and the rest of the world. Int. J. Mol. Sci. 2019;20(20):5201. DOI: https://doi.org/10.3390/ijms20205201
- Repass G.L., Palmer W.C., Stancampiano F.F. Hand, foot, and mouth disease: identifying and managing an acute viral syndrome. Cleve Clin. J. Med. 2014;81(9):537–43. DOI: https://doi.org/10.3949/ccjm.81a.13132
- Zhao T.S., Du J., Sun D.P., et al. A review and meta-analysis of the epidemiology and clinical presentation of coxsackievirus A6 causing hand-foot-mouth disease in China and global implications. Rev. Med. Virol. 2020;30(2):e2087. DOI: https://doi.org/10.1002/rmv.2087
- Yao X., Bian L.L., Lu W.W., et al. Enterovirus spectrum from the active surveillance of hand foot and mouth disease patients under the clinical trial of inactivated Enterovirus A71 vaccine in Jiangsu, China, 2012-2013. J. Med. Virol. 2015;87(12):2009–17. DOI: https://doi.org/10.1002/jmv.24275
- Guo W.P., Lin X.D., Chen Y.P., et al. Fourteen types of co-circulating recombinant enterovirus were associated with hand, foot, and mouth disease in children from Wenzhou, China. J. Clin. Virol. 2015;70:29–38. DOI: https://doi.org/10.1016/j.jcv.2015.06.093
- Pons-Salort M., Parker E.P.K., Grassly N.C. The epidemiology of non-polio enteroviruses: recent advances and outstanding questions. Curr. Opin. Infect. Dis. 2015;28(5):479–87. DOI: https://doi.org/10.1097/QCO.0000000000000187
- Solomon T., Lewthwaite P., Perera D., et al. Virology, epidemiology, pathogenesis, and control of enterovirus 71. Lancet Infect. Dis. 2010;10(11):778–90. DOI: https://doi.org/10.1016/S1473-3099(10)70194-8
- Mao Q., Wang Y., Yao X., et al. Coxsackievirus A16: epidemiology, diagnosis, and vaccine. Hum. Vaccin. Immunother. 2014;10(2):360–7. DOI: https://doi.org/10.4161/hv.27087
- Xing W., Liao Q., Viboud C., et al. Epidemiological characteristics of hand-foot-and-mouth disease in China, 2008–2012. Lancet Infect. Dis. 2014;14(4):308–18. DOI: https://doi.org/10.1016/S1473-3099(13)70342-6
- Yang Z.Y., Chen X.Q., Sun D., Wei D. Mortality in children with severe hand, foot and mouth disease in Guangxi, China. Indian Pediatr. 2018; 55(2):137–9.
- Bian L., Wang Y., Yao X., et al. Coxsackievirus A6: a new emerging pathogen causing hand, foot and mouth disease outbreaks worldwide. Expert Rev. Anti Infect. Ther. 2015;13(9):1061–71. DOI: https://doi.org/10.1586/14787210.2015.1058156
- Robinson C.R., Doane F.W., Rhodes A.J. Report of an outbreak of febrile illness with pharyngeal lesions and exanthem: Toronto, summer 1957 – isolation of group A Coxsackie virus. Can. Med. Assoc. J. 1958;79(8):615–21.
- Nassef C., Ziemer C., Morrell D.S. Hand-foot-and-mouth disease: a new look at a classic viral rash. Curr. Opin. Pediatr. 2015;27(4):486–91. DOI: https://doi.org/10.1097/MOP.0000000000000246
- Kimmis B.D., Downing C., Tyring S. Hand-foot-and-mouth disease caused by coxsackievirus A6 on the rise. Cutis. 2018;102(5):353–6.
- No T.H., Jo K.M., Jung S.Y., et al. Coxsackievirus A6-induced hand-foot-and-mouth disease mimicking Stevens–Johnson syndrome in an immunocompetent adult. Infect. Chemother. 2020;52(4):634–40. DOI: https://doi.org/10.3947/ic.2020.52.4.634
- Cheng F.F., Zhang B.B., Cao M.L., et al. Clinical characteristics of 68 children with atypical hand, foot, and mouth disease caused by coxsackievirus A6: a single-center retrospective analysis. Transl. Pediatr. 2022;11(9):1502–9. DOI: https://doi.org/10.21037/tp-22-352
- Новикова Н.А., Голицына Л.Н., Зверев В.В. Энтеровирусы в Российской Федерации в 2014 году. В кн.: Информационный бюллетень «Заболеваемость, этиологическая структура и вопросы профилактики энтеровирусной (неполио) инфекции». Том 2. Нижний Новгород;2015:7–10. Novikova N.A., Golitsyna L.N., Zverev V.V. Enteroviruses in the Russian Federation in 2014. In: Newsletter «Morbidity, Etiological Structure and Issues of Prevention of Enterovirus (Non-Polio) Infection». Volume 2. Nizhny Novgorod;2015:7–10. EDN: https://elibrary.ru/xwaren
- Голицына Л.Н., Зверев В.В., Епифанова Н.В. и др. Неполиомиелитные энтеровирусы в Российской Федерации в 2017 году. В кн.: Информационный бюллетень «Заболеваемость, этиологическая структура и вопросы профилактики энтеровирусной (неполио) инфекции». Нижний Новгород; 2018;5:5–12. Golitsyna L.N., Zverev V.V., Epifanova N.V., et al. Non-polio enteroviruses in the Russian Federation in 2017. In: Newsletter «Morbidity, Etiological Structure and Issues of Prevention of Enterovirus (Non-Polio) Infection». Volume 5. Nizhny Novgorod; 2018;5:5–12. EDN: https://elibrary.ru/osqidp
- Сапега Е.Ю., Бутакова Л.В., Троценко О.Е. и др. Современные молекулярно-генетические технологии в изучении интенсивности эпидемического процесса энтеровирусной инфекции в ряде субъектов Сибирского федерального округа Российской Федерации. Здоровье населения и среда обитания – ЗНиСО. 2019;(12):42–50. Sapega E.Yu., Butakova L.V., Trotsenko O.E., et al. State-of-the-art molecular genetic techniques in surveillance over intensity of enterovirus infection epidemic process in some constituent entities of the Siberian Federal district of the Russian Federation. Public Health and Life Environment – PH&LE. 2019;(12):42–50. DOI: https://doi.org/10.35627/2219-5238/2019-321-12-42-50 EDN: https://elibrary.ru/roivaw
- Nix W.A., Oberste M.S., Pallansch M.A. Sensitive, seminested PCR amplification of VP1 sequences for direct identification of all enterovirus serotypes from original clinical specimens. J. Clin. Microbiol. 2006;44(8):2698–704. DOI: https://doi.org/10.1128/JCM.00542-06
- Nasri D., Bouslama L., Omar S., et al. Typing of human enterovirus by partial sequencing of VP2. J. Clin. Microbiol. 2007;45(8):2370–9. DOI: https://doi.org/10.1128/JCM.00093-07
- Gaunt E., Harvala H., Österback R., et al. Genetic characterization of human coxsackievirus A6 variants associated with atypical hand, foot and mouth disease: a potential role of recombination in emergence and pathogenicity. J. Gen. Virol. 2015;96(Pt. 5):1067–79. DOI: https://doi.org/10.1099/vir.0.000062
- Лукашев А.Н., Голицына Л.Н., Вакуленко Ю.А. и др. Современные возможности и направления развития молекулярно-эпидемиологического мониторинга в надзоре за энтеровирусными инфекциями. Опыт Российской Федерации. Инфекция и иммунитет. 2018;8(4):452–64. Lukashev A.N., Golitsina L.N., Vakulenko Y.A., et al. Current possibilities and potential development of molecular enterovirus surveillance. Experience of Russian Federation. Infection and Immunity. 2018;8(4):452–64. DOI: https://doi.org/10.15789/2220-7619-2018-4-452-464 EDN: https://elibrary.ru/ywwjhn
- Muir P., Kämmerer U., Korn K., et al. Molecular typing of enteroviruses: current status and future requirements. The European Union Concerted Action on Virus Meningitis and Encephalitis. Clin. Microbiol. Rev. 1998;11(1):202–27. DOI: https://doi.org/10.1128/CMR.11.1.202
- Anh N.T., Nhu L.N.T., Van H.M.T., et al. Emerging coxsackievirus A6 causing hand, foot and mouth disease, Vietnam. Emerg. Infect. Dis. 2018;24(4):654–62. DOI: https://doi.org/10.3201/eid2404.171298
- He Y.Q., Chen L., Xu W.B., et al. Emergence, circulation, and spatiotemporal phylogenetic analysis of сoxsackievirus A6- and Coxsackievirus A10-associated hand, foot, and mouth disease infections from 2008 to 2012 in Shenzhen, China. J. Clin. Microbiol. 2013;51(11):3560–6. DOI: https://doi.org/10.1128/JCM.01231-13
- Song Y., Zhang Y., Ji T., et al. Persistent circulation of Coxsackievirus A6 of genotype D3 in mainland of China between 2008 and 2015. Sci. Rep. 2017;7(1):5491. DOI: https://doi.org/10.1038/s41598-017-05618-0
- Ngangas S.T., Bisseux M., Jugie G., et al. Coxsackievirus A6 recombinant subclades D3/A and D3/H were predominant in hand-foot-and-mouth disease outbreaks in the paediatric population, France, 2010–2018. Viruses. 2022;14(5):1078. DOI: https://doi.org/10.3390/v14051078
- Puenpa J., Saengdao N., Khanarat N., et al. Evolutionary and genetic recombination analyses of Coxsackievirus A6 variants associated with hand, foot, and mouth disease outbreaks in Thailand between 2019 and 2022. Viruses. 2023;15(1):73. DOI: https://doi.org/10.3390/v15010073
- McWilliam Leitch E.C., Bendig J., Cabrerizo M., et al. Transmission networks and population turnover of echovirus 30. J. Virol. 2009;83(5):2109–18. DOI: https://doi.org/10.1128/JVI.02109-08
- Lukashev A.N., Lashkevich V.A., Ivanova O.E., et al. Recombination in circulating enteroviruses. J. Virol. 2003;77:10423–31. DOI: https://doi.org/10.1128/jvi.77.19.10423-10431.2003
- Lindberg M.A., Andersson P., Savolainen C., et al. Evolution of the genome of Human enterovirus B: incongruence between phylogenies of the VP1 and 3CD regions indicates frequent recombination within the species. J. Gen. Virol. 2003;84:1223–35. DOI: https://doi.org/10.1099/vir.0.18971-0
- Lu J., Zeng H., Zheng H., et al. Hand, foot and mouth disease in Guangdong, China, in 2013: new trends in the continuing epidemic. Clin. Microbiol. Infect. 2014;20(7):O442–5. DOI: https://doi.org/10.1111/1469-0691.12468
- Feng X., Guan W., Guo Y., et al. A novel recombinant lineage's contribution to the outbreak of coxsackievirus A6-associated hand, foot and mouth disease in Shanghai, China, 2012–2013. Sci. Rep. 2015;5:11700. DOI: https://doi.org/10.1038/srep11700
- Lukashev A.N., Ivanova O.E., Eremeeva T.P., Gmyl L.V. Analysis of echovirus 30 isolates from Russia and new independent states revealing frequent recombination and reemergence of ancient lineages. J. Clin. Microbiol. 2008;46(2):665–70. DOI: https://doi.org/10.1128/JCM.02386-06
- Benschop K.S.M., Broberg E.K., Hodcroft E., et al. Molecular epidemiology and evolutionary trajectory of emerging echovirus 30, Europe. Emerg. Infect. Dis. 2021;27(6):1616–26. DOI: https://doi.org/10.3201/eid2706.203096
- Domingo E., Martin V., Perales C., Escarmis C. Coxsackieviruses and quasispecies theory: evolution of enteroviruses. Curr. Top. Microbiol. Immunol. 2008;323:3–32. DOI: https://doi.org/10.1007/978-3-540-75546-3_1
- Lukashev A.N., Lashkevich V.A., Ivanova O.E., et al. Recombination in circulating Human enterovirus B: independent evolution of structural and non-structural genome regions. J. Gen. Virol. 2005;86(Pt. 12):3281–90. DOI: https://doi.org/10.1099/vir.0.81264-0
- Лукашев А.Н. Роль рекомбинации в эволюции энтеровирусов. Вопросы вирусологии. 2005;50(3):46–52. Lukashev A.N. Role of recombination in evolution of enteroviruses. Problems of Virology. 2005;15(3):157–67. DOI: https://doi.org/10.1002/rmv.457 EDN: https://elibrary.ru/hsghuv
- Combelas N., Holmblat B., Joffret M.L., et al. Recombination between poliovirus and coxsackie A viruses of species C: a model of viral genetic plasticity and emergence. Viruses. 2011;3(8):1460–84. DOI: https://doi.org/10.3390/v3081460
- Ngangas S.T., Lukashev A., Jugie G., et al. Multirecombinant enterovirus A71 subgenogroup C1 isolates associated with neurologic disease, France, 2016-2017. Emerg. Infect. Dis. 2019;25(6):1204–8. DOI: https://doi.org/10.3201/eid2506.181460
- Khetsuriani N., LaMonte-Fowlkes A., Oberste M.S., Pallansch M.A. Enterovirus surveillance-United States, 1970–2005. MMWR Surveill. Summ. 2006;55(8):1–20.
- Abedi G.R., Watson J.T., Pham H., et al. Enterovirus and human parechovirus surveillance – United States, 2009–2013. MMWR Morb. Mortal Wkly Rep. 2015;64(34):940–3. DOI: https://doi.org/10.15585/mmwr.mm6434a3
- Wang S.H., Wang A., Liu P.P., et al. Divergent pathogenic properties of circulating Coxsackievirus A6 associated with emerging hand, foot, and mouth disease. J. Virol. 2018;92(11):e00303-18. DOI: https://doi.org/10.1128/JVI.00303-18
- Akhmadishina L.V., Govorukhina M.V., Kovalev E.V., et al. Enterovirus A71 meningoencephalitis outbreak, Rostov‐on‐Don, Russia, 2013. Emerg. Infect. Dis. 2015;21(8):1440–3. DOI: https://doi.org/10.3201/eid2108.141084
- Lukashev A.N. Recombination among picornaviruses. Rev. Med. Virol. 2010;20(5):327–37. DOI: https://doi.org/10.1002/rmv.660
Дополнительные файлы