Изучение генетического разнообразия вируса ветряной оспы в отдельных регионах Российской Федерации при помощи высокопроизводительного секвенирования
- Авторы: Надтока М.И.1, Лысенков В.Г.1, Аглетдинов М.Р.1, Мишкин А.А.1, Афонина Н.М.1, Плоскирева А.А.1, Михеева И.В.1, Хафизов К.Ф.1, Акимкин В.Г.1
-
Учреждения:
- Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
- Выпуск: Том 100, № 5 (2023)
- Страницы: 267-275
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/18473
- DOI: https://doi.org/10.36233/0372-9311-423
- EDN: https://elibrary.ru/tkklhp
- ID: 18473
Цитировать
Аннотация
Введение. Вирус ветряной оспы (VZV) — возбудитель одноимённого заболевания и опоясывающего лишая, филогенетически подразделяется на 8 клад, для распространения которых характерна географическая привязка к тем или иным регионам мира. Для большинства стран установлены циркулирующие на их территориях клады VZV, однако для России аналогичная информация практически отсутствует.
Цель исследования — разработка эффективной методики типирования VZV с использованием технологий высокопроизводительного секвенирования для выявления распространённости различных клад VZV в Москве, Московской области и Ставропольском крае.
Материалы и методы. Для генотипирования VZV достаточно задействовать 7 нуклеотидных позиций, по уникальным сочетаниям которых возможно отнести вирус к одной из клад. Короткие участки нуклеотидных последовательностей открытых рамок считывания получали при помощи разработанного набора праймеров.
Результаты. Разработана и оптимизирована методика генотипирования VZV. При помощи данной методики получены первичные данные о распределении клад VZV в исследуемых регионах. Таким образом, было установлено, что в Москве и ряде других регионов распространены преимущественно 1, 3 и 5-я клады VZV.
Заключение. Разработанная методика, включающая праймерную панель и алгоритм генотипирования, позволяет произвести типирование VZV в короткие сроки при снижении затрат на пробоподготовку и одновременном увеличении количества образцов в одном цикле секвенирования. Результаты, полученные с использованием данного протокола, позволяют сделать предположение о том, что в Москве, Московской области и Ставропольском крае наибольшую представленность имеют клады 1, 3 и 5 VZV. Для подтверждения данной гипотезы требуется включить в последующие исследования большее количество клинических образцов, в том числе из других регионов страны.
Полный текст
Введение
Вирус ветряной оспы (Varicella-Zoster virus, VZV) относится к семейству Herpesviridae, роду Varicellovirus, подсемейству Alphaherpesvirinae. Характерными чертами представителей данного подсемейства являются короткий репродуктивный цикл, быстрое распространение и эффективное разрушение инфицированных клеток. VZV способен реплицироваться в ограниченном спектре организмов-хозяев, который включает исключительно клетки человеческого и обезьяньего происхождения. Ежегодная заболеваемость ветряной оспой (ВО) колеблется от 13 до 16 случаев на 1000 человек с существенными вариациями по годам. В странах с умеренным климатом заболеваемость ВО, в зависимости от возраста, наиболее высока среди детей дошкольного (1–4 года) или младшего школьного возраста (5–9 лет) и составляет более 100 случаев на 1000 детей в год. Таким образом, более 90% людей заражаются до подросткового возраста, и лишь небольшая часть взрослых (< 5–10%) остаётся восприимчивой к заболеванию. В тропическом климате заражение происходит в более высоком общем среднем возрасте, при этом доля случаев заболевания среди взрослых выше [1, 2].
Инфицирование VZV имеет ярко выраженный сезонный характер, пик заболеваемости приходится на зимний и весенний периоды, при этом с периодичностью 2–3 года происходят вспышки заболеваемости. В развитых странах 5 из 1000 больных ВО госпитализируются, а 2–3 случая заболевания на 100 тыс. инфицированных заканчиваются летально [2]. В довакцинальную эпоху ВО являлась повсеместно встречающейся детской инфекцией в странах с умеренным климатом. К примеру, в США порядка 98% населения к возрасту 20 лет были сероположительны к VZV.
Первая живая аттенуированная вакцина (vОка) против данного возбудителя была получена в 1974 г. путём серии пассажей клинического изолята VZV (Ока) дикого типа. Позднее данная вакцина была усовершенствована, в результате был выпущен препарат «Varivax» («Merck Sharp & Dohme Corp.») [3]. Живая аттенуированная вакцина состоит из смеси различных геновариантов VZV, содержащих 42 однонуклеотидных полиморфизма, которые отличают vОка от родительского штамма Ока дикого типа [2]. Благодаря разработанной вакцине и введению в разных странах обязательных программ вакцинации против VZV, госпитализация и смертность от ВО среди детей снизилась более чем на 90% [2, 3].
Геном вируса ветряной оспы
Как и все герпесвирусы, VZV является двуцепочечным ДНК-вирусом и имеет геном размером 125 000 п.н., включающий 72 открытые рамки считывания (ORF), образующие 71 ген. Поскольку 3 гена представляют из себя копии других генов, то геном содержит 68 уникальных генов. Геном вируса состоит из двух основных кодирующих областей: уникальной длинной (UL) и уникальной короткой (US). Регион UL фланкирован короткими (около 88 п.н.) инвертированными повторами TRL и IRL, тогда как область US окружена длинными (7319 п.н.) повторами TRS и IRS. Пять участков генома содержат тандемные прямые повторы (R1, R2, R3, R4 и R5), которые представляют собой короткие последовательности с высоким содержанием G + C [4, 5]. Три из этих повторяющихся последовательностей (R1, R2 и R3) расположены в кодирующей области генов ORF11, ORF14 и ORF22 соответственно и, таким образом, могут оказывать влияние на функции белков. Две копии R4 находятся внутри повторов IR и TR, соседних с точкой начала репликации (OriS), а R5 расположен между ORF60 и ORF61 [5].
Генотипирование VZV
До 2008 г. не существовало единой номенклатуры для типирования VZV. Ранняя классификация VZV была основана на результатах анализа полиморфизма длин рестрикционных фрагментов (ПДРФ). Маркеры ПДРФ, задействованные в данных исследованиях, включали полиморфизм ORF38 (PstI), ORF54 (BglI) и ORF62 (SmaI) [6, 7]. Таким образом, большинство штаммов дикого типа из США и Европы были охарактеризованы как PstI+BglI–; штаммы, распространённые на территории Азии и Африки, — как BglI+; Ока-подобные штаммы дикого типа из Японии — как PstI+/PstI–BglI+SmaI–; вакцинные штаммы Ока — как PstI–BglI+SmaI+ [7, 8].
Предпринимались попытки идентификации вариантов VZV при помощи полногеномного скрининга на наличие однонуклеотидных замен посредством гетеродуплексной мобильности. Данный подход использовался для оценки замен в ORF1, ORF21, ORF50 и ORF54, что позволило выделить 4 основные клады (геноварианта), названные A, B, C и J. В кладе A кластеризовались штаммы из Африки и Азии, тогда как клады B и C преимущественно состояли из европейских штаммов. Впоследствии к этой схеме генотипирования была добавлена клада J для учёта японских штаммов [9, 10].
Другой подход к генотипированию основывается на анализе однонуклеотидных полиморфизмов в последовательностях генов 5 гликопротеинов (gH, gI, gL, gB, gE), а также основного гена трансактиватора IE62. Благодаря этому штаммы VZV были классифицированы в 4 клады: A, B, C и D. Клады A и D были представлены изолятами, отобранными на территории Северной Америки и Европы, клады B и C состояли из представителей VZV, распространённых на территории Сингапура и Японии соответственно [11, 12]. Необходимо отметить, что хотя номенклатура клад из данного исследования во многом схожа с номенклатурой, использованной в работах W. Barrett‐Muir и соавт. [8, 9], она не имеет корреляции между рассматриваемыми методами типирования.
Более поздние филогенетические анализы, включающие как полные геномы VZV, так и их фрагменты, позволили соотнести геномные вариации со специфичными геновариантами и предположительно рекомбинантными вирусами. Посредством мультилокусного анализа полиморфизмов в геномных последовательностях VZV были выделены 3 основных геноварианта (клады): E (Европейский), J (Японский) и M (мозаичный) [13]. Впоследствии геновариант M был разделён на 4 отдельных варианта: M1, M2, M3, M4 [13, 14]. Принадлежащие к геновариантам М1 и М2 штаммы были наиболее распространены в тропических регионах, геновариант Е — в умеренных широтах, геновариант J — в Японии. В отдельных исследованиях штамм с геновариантом М3 был обнаружен в США, а штаммы с геновариантом М4 — в Испании и Франции [14, 15].
Поскольку номенклатура клад/геновариантов VZV была основана на различных методах молекулярного типирования, в 2008 г. была введена новая универсальная номенклатура, разделяющая геноварианты на 5 основных клад (1–5) с 2 условными (кандидатными) кладами (VI и VII) [16]. Также теперь для выделения VZV в отдельную кладу необходимо, чтобы к ней относились как минимум 2 представителя, для которых получены последовательности полного генома. В ином случае такая клада считается кандидатной и обозначается римскими цифрами.
Благодаря секвенированию полных геномов из клинических образцов VZV, взятых на территории Германии, были обнаружены как штаммы, принадлежащие к кладам 1, 3 и 5, так и штаммы, которые не относились ни к одной из известных клад. Поэтому данные представители (1483/2005 и 457/2008) были выделены в отдельные кандидатные клады — VIII и IX соответственно [17]. Позднее, в ходе работы по исследованию событий рекомбинации между представителями различных клад VZV, был обнаружен штамм, который сгруппировался с единственным представителем клады VI на филогенетическом древе. Таким образом, в 2015 г. кандидатная клада VI была преобразована в основную кладу 6 [18]. В 2017 г. ранее использовавшаяся схема генотипирования VZV была усовершенствована, за счёт чего удалось выявить новых представителей кандидатной клады IX (одного полного и одного частичного генома). При этом ранее был идентифицирован и описан только один изолят, принадлежащий к данной кладе (JN704710). Наличие полной последовательности генома изолята KY037798, идентифицированного в данном исследовании, соответствовало требованию к преобразованию кандидатной клады IX в основную кладу 9 [19].
На сегодняшний день разнообразие VZV представлено 8 кладами: 7 основных (1–6 и 9) и 1 кандидатная клада VIII; клада VII стала неактуальной ввиду отсутствия новых изолятов. Основные клады VZV, как правило, имеют географическую привязку к определённым регионам. В частности, клады 1, 3 и 6 в основном распространены в Европе, Северной Америке и Австралии, клада 2 — преимущественно в Азии. Клады 4 и 5, как правило, имеют распространение во множестве регионов по всему миру; клада 5 является единственной, о циркуляции которой сообщается в Африке [20]. Несмотря на обширное количество информации о циркулирующих по всему миру кладах VZV, распределение клад VZV в России не освещено в достаточной степени. В научной литературе практически отсутствует актуальная информация по данной теме. Сообщается, что в северном регионе Китая Синьцзян, который граничит с Россией, преобладают клады 1 и 3 [21].
В качестве первого шага к получению достоверных данных о циркулирующих на территории России представителях различных клад VZV нами была разработана специфичная праймерная панель, позволяющая типировать VZV в соответствии со схемой, предложенной N.J. Jensen и соавт. [19]. Панель включает 6 пар праймеров, охватывающих короткие (порядка 200 п.н.) области открытых рамок считывания: ORF21, ORF22, ORF 29, ORF38, ORF55, ORF67, которые содержат 7 однонуклеотидных замен, по уникальным композициям которых возможно определить принадлежность вируса к одной из известных клад. Благодаря модификации праймеров путём объединения с адаптерными последовательностями Nextera в совокупности с возможностью секвенирования получаемых ДНК-библиотек на платформе «Illumina» с наборами реагентов для коротких прочтений, удаётся получить результаты типирования в течение 48 ч. Таким образом, разработанная праймерная панель позволяет сократить финансовые и временны́е затраты при единовременном увеличении количества образцов в одном запуске прибора для высокопроизводительного секвенирования.
Разработанное нами решение, предназначенное для быстрого скрининга большого числа клинических образцов, было протестировано на образцах из Москвы, Московской области и Ставропольского края, что позволило рассмотреть представленность VZV в отдельных регионах РФ в первом приближении.
Материалы и методы
Образцы VZV
В исследовании использованы 75 образцов VZV, собранных в период с июня 2022 г. по июль 2023 г. Клинические образцы вирусов были получены от пациентов преимущественно из Москвы (61 образец), а также из Московской области (9 образцов) и Ставропольского края (5 образцов). Забор биологического материала производили после получения добровольного информированного согласия пациентов или их представителей. Проводимое исследование одобрено на заседании Локального этического комитета ЦНИИ Эпидемиологии Роспотребнадзора от 24.05.2022 (протокол № 124).
Выделение ДНК и получение фрагментов геномов VZV
Вирусную ДНК выделяли из клинических образцов с использованием набора реагентов «РИБО-преп» («АмплиСенс»). Полученную ДНК затем использовали в качестве матрицы для проведения ПЦР с целью амплификации фрагментов генома VZV. Области открытых рамок считывания (ORF21, ORF22, ORF 29, ORF38, ORF55, ORF67), содержащие дискриминирующие мутации, в соответствии с которыми возможно произвести типирование VZV, получали при помощи разработанного набора праймеров (табл. 1), смешанных в пул (табл. 2).
Таблица 1. Список праймеров, используемых для амплификации фрагментов генома VZV
Table 1. List of primers used to amplify VZV genome fragments
Праймер Primer | Последовательность праймера с адаптерной последовательностью Nextera (5’→3’) Primer sequence with Nextera adapter sequence (5'→3') |
VZV_1_fwd | TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGgcggttttaacttcacaatgtaat |
VZV_1_rev | GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGctcatctggacgaagcca |
VZV_2.2_fwd | TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGttacccacaagcacgtcag |
VZV_2.2_rev | GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGtcatcaaaccatgttaaccctc |
VZV_3_fwd | TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGaatatgttacggggacctttga |
VZV_3_rev | GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGcaaacaccccaataggttga |
VZV_4_fwd | TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGgccatataccgcaacaactg |
VZV_4_rev | GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGcctcgccataaagccactac |
VZV_5_fwd | TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGccaccacggtggactatg |
VZV_5_rev | GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGgaggagaccgtacgcga |
VZV_6_fwd | TCGTCGGCAGCGTCAGATGTGTATAAGAGACAGctttgatcttcaagggcgac |
VZV_6_rev | GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGcgctatagtttgtcccggt |
Примечание. Прописными буквами обозначены адаптерные последовательности Nextera, «сшитые» с праймерами (обозначены строчными буквами).
Note. Capital letters indicate Nextera adapter sequences fused to primers (indicated in lower case letters).
Таблица 2. Состав смеси праймеров для типирования VZV
Table 2. Composition of the mixture of primers for VZV typing
Праймер Primer | Концентрация праймеров в стоке, пмоль/мкл Primer concentration in mix, pmol/μl | Концентрация после разведения в 2 раза, пмоль/мкл Concentration after 2-fold dilution, pmol/μL |
VZV_1_fwd | 10,7 | 5,35 |
VZV_1_rev | 10,7 | 5,35 |
VZV_2.2_fwd | 7,1 | 3,55 |
VZV_2.2_rev | 7,1 | 3,55 |
VZV_3_fwd | 10,7 | 5,35 |
VZV_3_rev | 10,7 | 5,35 |
VZV_4_fwd | 9,5 | 4,75 |
VZV_4_rev | 9,5 | 4,75 |
VZV_5_fwd | 7,1 | 3,55 |
VZV_5_rev | 7,1 | 3,55 |
VZV_6_fwd | 4,7 | 2,35 |
VZV_6_rev | 4,7 | 2,35 |
Амплификацию проводили в 25 мкл реакционной смеси со следующим составом: 10 мкл ПЦР-смесь-2 blue («АмплиСенс»), 1,8 мкл 4,4 мМ дезоксинуклеозидтрифосфата («АмплиСенс»), 1 мкл смеси праймеров, 5 мкл ДНК-матрицы; до требуемого объёма реакционную смесь доводили H2O mQ. Для амплификации использовали следующий температурно-временной протокол: 95ºC — 2 мин; затем 40 циклов: 95ºC — 15 с, 60°C — 30 с, 72ºC — 1 мин; финальная элонгация: 72ºC — 3 мин. Целевые фрагменты геномов VZV идентифицировали по свечению бромистого этидия в 1,7% агарозном геле. Визуализацию результатов электрофоретического анализа проводили при помощи «GelDoc EZ» («Bio-Rad»).
Подготовка библиотек для секвенирования
Очистку ПЦР-продукта от реакционной смеси осуществляли с использованием «AMPureXP beads» («Beckman Coulter») в соотношении 1 : 0,8 (образец : частицы). Праймеры, используемые для амплификации сегментов генома VZV, модифицированы путём совмещения с адаптерами «Nextera» («Illumina»), в результате чего получаемые ампликоны оказываются фланкированы специфичными последовательностями, облегчающими процесс подготовки библиотек. Баркодирование проводили с использованием 10 мкл ПЦР-смеси-2 blue («АмплиСенс»), 1 мкл EvaGreen («Biotium») в качестве флуоресцентного интеркалирующего красителя, 2 мкл каждого из баркодов (5 пмоль) и 8 мкл очищенного ПЦР-продукта. Для баркодирования использовали следующий температурно-временной протокол: 98ºC — 30 с; 12 циклов: 98ºC — 10 с, 65ºC — 1 мин 15 с. Концентрацию ДНК после баркодирования измеряли на флуориметре «Qubit 4.0» с использованием набора «Qubit dsDNA HS Assay Kit» («Thermo Fisher Scientific»). Готовые библиотеки были объединены и очищены при помощи магнитных частиц «AMPureXP beads» («Beckman Coulter»). Длины готовых библиотек идентифицировали с использованием системы автоматизированного электрофореза «2100 Bioanalyzer» («Agilent Technologies»).
Секвенирование библиотек VZV
Секвенирование осуществляли на платформе «Illumina MiSeq» с использованием набора реагентов «MiSeq Reagent Kit v2» (300 циклов) и «MiSeq Reagent Kit v3» (600 циклов). При этом на один образец отводилось менее 0,5% запуска «MiSeq».
Определение последовательностей ДНК и геновариантов образцов
После получения «сырых» данных с прибора прочтения ДНК были триммированы от вышеописанных праймерных последовательностей с помощью программного инструмента fastp 0.23.0 [22]. Далее с помощью bwa 0.7.17 [23] прочтения были выровнены («картированы») на референсную последовательность вируса NC_001348.1, отсортированы и индексированы с помощью samtools 1.15.1 [24]. Получившиеся выравнивания были представлены в виде черновых сборок генома с помощью samtools 1.15.1 и собственного скрипта, после чего перекартированы с помощью MAFFT v7.490 [25] для получения финальных сборок (консенсусных последовательностей).
Согласно вышеупомянутой номенклатуре вирусных клад [19], для успешного генотипирования достаточно задействовать всего 7 позиций, сочетание вариантов (однонуклеотидных замен) в которых указывает на ту или иную кладу (табл. 3). Сконструированные нами праймеры (табл. 1) составлены таким образом, чтобы включать данные варианты.
Таблица 3. Схема генотипирования, предложенная N.J. Jensen и соавт. [19] (адаптировано)
Table 3. The genotyping scheme proposed by N.J. Jensen et al. [19] (adapted)
Примечание. Чёрным выделены наименее распространённые «дискриминирующие» варианты.
Note. The least common “discriminatory” variants are highlighted in black.
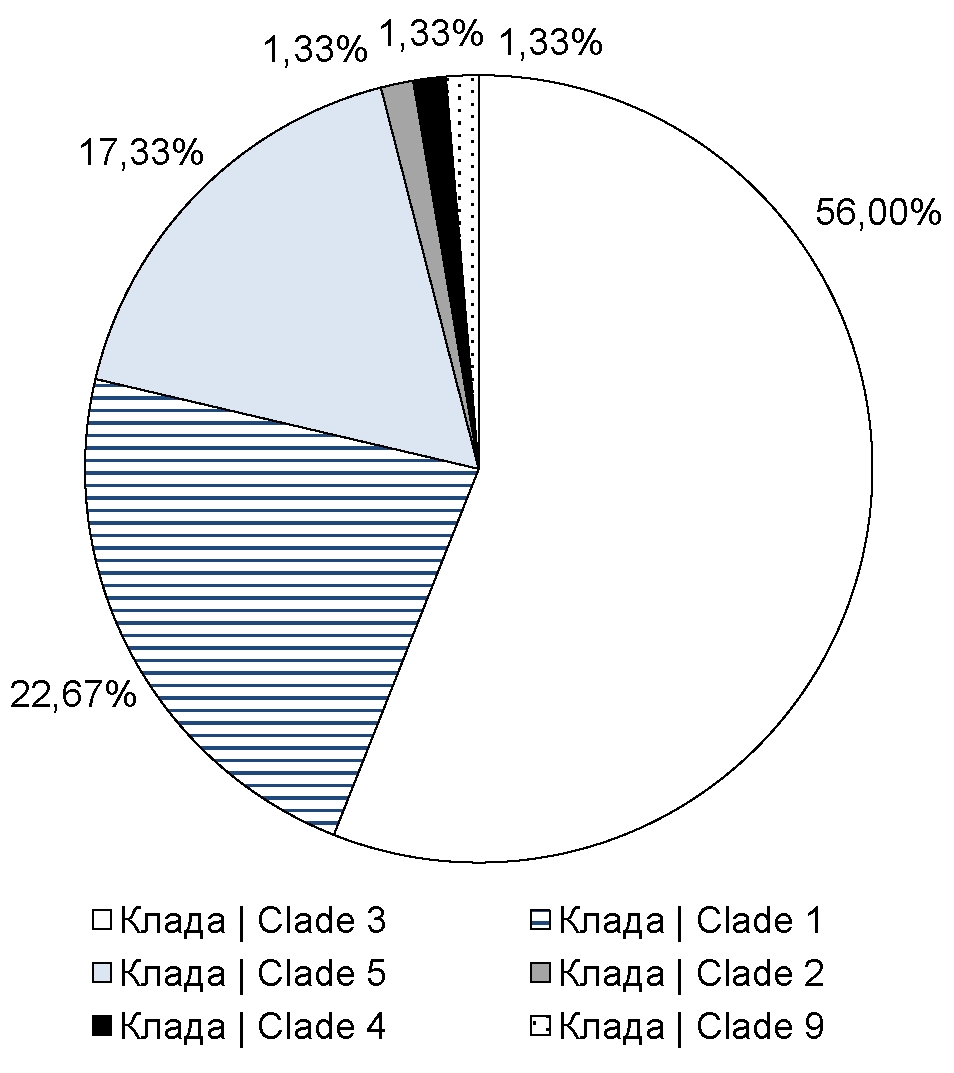
Представленность различных клад VZV в отдельных регионах РФ.
Representation of various clades of VZV in the Russian Federation
Результаты
Разработанная нами праймерная панель, описанная в предыдущем разделе, охватывает короткие сегменты 6 открытых рамок считывания: ORF21, ORF22, ORF29, ORF38, ORF55 и ORF67, которые содержат однонуклеотидные замены, являющиеся целевыми для осуществления типирования VZV. С использованием данной панели нами было произведено секвенирование клинических образцов VZV, полученных от пациентов из Москвы, Московской области и Ставропольского края. Полученные последовательности сегментов открытых рамок считывания были отсеквенированы на приборе «Illumina MiSeq», при этом среднее покрытие составило приблизительно 104. Минимальное покрытие для каждого сегмента было около 300 прочтений. Консенсусные последовательности получали путём обработки «сырых» данных секвенирования с использованием ранее описанных общедоступных биоинформатических инструментов и собственного скрипта. Собранные консенсусные последовательности использовали в качестве входных данных для разработанного алгоритма, осуществляющего генотипирование VZV на основе дискриминирующих мутаций. Применяя данный алгоритм генотипирования, мы установили, что 17 образцов (22,67% от общего числа анализируемых последовательностей) приходились на кладу 1, 42 образца (56%) — на кладу 3 (рисунок), 13 (17,33%) образцов принадлежали к кладе 5. Помимо этого, среди всех секвенированных образцов нами выявлены единичные случаи инфекций, вызванных вирусами клад 2 (1,4%), 4 (1,4%) и 9 (1,4%) VZV (рисунок).
Представленность различных клад VZV в отдельных регионах РФ.
Обсуждение
В ходе данного исследования нами была разработана и оптимизирована методика быстрого типирования большого количества образцов VZV. Более того, используя описанный протокол генотипирования, мы получили первичные данные по кладам VZV, которые получили распространение на территории Москвы, Московской области и Ставропольского края. С использованием технологий секвенирования следующего поколения мы установили, что в рассматриваемых регионах получили распространение преимущественно вирусы, принадлежащие к кладам 1, 3 и 5. Также нами были обнаружены единичные образцы, относящиеся к кладе 2, кладе 4 и кладе 9, которые на фоне остальных клад являются минорной частью циркулирующих в этих областях РФ VZV. Стоит уточнить, что полученная картина распределения клад является наиболее правдоподобной для Москвы, так как наибольшее количество клинических образцов было получено именно из этого региона РФ. В дальнейшем, при наличии соответствующей возможности, планируется как увеличить общее количество образцов из регионов, рассматриваемых в данной работе, так и включить в исследование другие регионы России.
На текущий момент данные, полученные в результате нашего исследования, лишь позволяют предположить о во многом схожей картине распределения представителей VZV в исследуемых регионах РФ и в странах Европы [20], а также соответствуют ранее приведенным данным о циркулирующих кладах VZV в регионе Китая, граничащем с Россией [21]. Однако в исследуемых регионах, по-видимому, значительно бо́льшую долю занимают вирусы, принадлежащие к кладе 3, кроме этого, процент представленности вирусов клады 5 в данных областях РФ также более высокий, нежели в европейских странах.
Заключение
VZV, этиологический агент ВО и опоясывающего герпеса, подразделяется на несколько ветвей, характеризующихся различными генетическими свойствами. Мы успешно использовали метод секвенирования следующего поколения для генотипирования VZV, что включает специальную панель праймеров и алгоритм генотипирования — подход, позволяющий ускорить процесс сбора данных и снизить затраты на подготовку образцов, особенно при увеличении количества образцов в одном цикле секвенирования. Используя эту методологию, мы установили, что в Москве, Московской области и Ставропольском крае преобладают клады 1, 3 и 5 VZV. Идентификация этих клад VZV предоставляет ценные эпидемиологические данные, полезные для формирования стратегий общественного здравоохранения, и приоткрывает завесу над распространённостью клад VZV в некоторых регионах РФ.
Отметим, что настоящее исследование является «пилотным» и по большей части носит методологический характер. Стоит также упомянуть, что в данной работе нами продемонстрирована распространённость клад VZV только для 3 из множества регионов РФ, таким образом, вопрос о глобальной представленности различных клад вируса в нашей стране остаётся открытым и поэтому следует продолжить, а также расширить будущие исследования в целях формирования достоверной картины распределения клад VZV на территории России.
Об авторах
Максим Игоревич Надтока
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Автор, ответственный за переписку.
Email: maximnadtoka@gmail.com
ORCID iD: 0009-0002-3217-0963
м.н.с. лаб. геномных исследований ЦНИИ Эпидемиологии Роспотребнадзора
Россия, МоскваВладислав Геннадиевич Лысенков
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: maximnadtoka@gmail.com
ORCID iD: 0000-0002-1468-1631
биоинформатик лаб. геномных исследований ЦНИИ Эпидемиологии Роспотребнадзора
Россия, МоскваМатвей Рашидович Аглетдинов
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: maximnadtoka@gmail.com
ORCID iD: 0000-0003-2249-7196
биоинформатик лаб. геномных исследований ЦНИИ Эпидемиологии Роспотребнадзора
Россия, МоскваАндрей Альбертович Мишкин
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: maximnadtoka@gmail.com
ORCID iD: 0000-0001-6911-6296
м.н.с. ЦНИИ Эпидемиологии Роспотребнадзора
Россия, МоскваНаталия Михайловна Афонина
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: maximnadtoka@gmail.com
ORCID iD: 0000-0002-3205-4025
к.м.н., н.с. лаб. иммунопрофилактики ЦНИИ Эпидемиологии Роспотребнадзора
Россия, МоскваАнтонина Александровна Плоскирева
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: maximnadtoka@gmail.com
ORCID iD: 0000-0002-3612-1889
д.м.н., зам. директора ЦНИИ Эпидемиологии Роспотребнадзора по клинической работе
Россия, МоскваИрина Викторовна Михеева
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: maximnadtoka@gmail.com
ORCID iD: 0000-0001-8736-4007
д.м.н., профессор, зав. лаб. иммунопрофилактики ЦНИИ Эпидемиологии Роспотребнадзора
Россия, МоскваКамиль Фаридович Хафизов
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: maximnadtoka@gmail.com
ORCID iD: 0000-0001-5524-0296
к.б.н., зав. лаб. геномных исследований ЦНИИ Эпидемиологии Роспотребнадзора
Россия, МоскваВасилий Геннадьевич Акимкин
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: maximnadtoka@gmail.com
ORCID iD: 0000-0003-4228-9044
д.м.н., профессор, академик РАН, директор ЦНИИ Эпидемиологии Роспотребнадзора
Россия, МоскваСписок литературы
- Laing K.J., Ouwendijk W.J.D., Koelle D.M., Verjans G.M.G.M. Immunobiology of varicella-zoster virus infection. J. Infect. Dis. 2018;218(Suppl. 2):S68–74. DOI: https://doi.org/10.1093/infdis/jiy403
- Gershon A.A., Breuer J., Cohen J.I., et al. Varicella zoster virus infection. Nat. Rev. Dis. Primers. 2015;1:15016. DOI: https://doi.org/10.1038/nrdp.2015.16
- Schmid D.S., Jumaan A.O. Impact of varicella vaccine on varicella-zoster virus dynamics. Clin. Microbiol. Rev. 2010; 23(1):202–17. DOI: https://doi.org/10.1128/cmr.00031-09
- Schmidt-Chanasit J., Sauerbrei A. Evolution and world-wide distribution of varicella-zoster virus clades. Infect. Genet. Evol. 2011;11(1):1–10. DOI: https://doi.org/10.1016/j.meegid.2010.08.014
- Depledge D.P., Sadaoka T., Ouwendijk W.J.D. Molecular aspects of varicella-zoster virus latency. Viruses. 2018;10(7):349. DOI: https://doi.org/10.3390/v10070349
- LaRussa P., Lungu O., Hardy I., et al. Restriction fragment length polymorphism of polymerase chain reaction products from vaccine and wild-type varicella-zoster virus isolates. J. Virol. 1992;66(2):1016–20. DOI: https://doi.org/10.1128/jvi.66.2.1016-1020.1992
- Loparev V.N., Argaw T., Krause P.R., et al. Improved identification and differentiation of varicella-zoster virus (VZV) wild-type strains and an attenuated varicella vaccine strain using a VZV open reading frame 62-based PCR. J. Clin. Microbiol. 2000;38(9):3156–60. DOI: https://doi.org/10.1128/jcm.38.9.3156-3160.2000
- Quinlivan M., Hawrami K., Barrett-Muir W., et al. The molecular epidemiology of varicella-zoster virus: evidence for geographic segregation. J. Infect. Dis. 2002;186(7):888–94. DOI: https://doi.org/10.1086/344228
- Barrett-Muir W., Scott F.T., Aaby P., et al. Genetic variation of varicella-zoster virus: evidence for geographical separation of strains. J. Med. Virol. 2003;70(Suppl. 1):S42–7. DOI: https://doi.org/10.1002/jmv.10319
- Muir W.B., Nichols R., Breuer J. Phylogenetic analysis of varicella-zoster virus: evidence of intercontinental spread of genotypes and recombination. J. Virol. 2002;76(4):1971–9. DOI: https://doi.org/10.1128/jvi.76.4.1971-1979.2002
- Faga B., Maury W., Bruckner D.A., Grose C. Identification and mapping of single nucleotide polymorphisms in the varicella-zoster virus genome. Virology. 2001;280(1):1–6. DOI: https://doi.org/10.1006/viro.2000.0775
- Wagenaar T.R., Chow V.T., Buranathai C., et al. The out of Africa model of varicella-zoster virus evolution: single nucleotide polymorphisms and private alleles distinguish Asian clades from European/North American clades. Vaccine. 2003;21(11-12):1072–81. DOI: https://doi.org/10.1016/s0264-410x(02)00559-5
- Loparev V.N., Gonzalez A., Deleon-Carnes M., et al. Global identification of three major genotypes of varicella-zoster virus: longitudinal clustering and strategies for genotyping. J. Virol. 2004;78(15):8349–58. DOI: https://doi.org/10.1128/jvi.78.15.8349-8358.2004
- Loparev V., Martro E., Rubtcova E., et al. Toward universal varicella-zoster virus (VZV) genotyping: diversity of VZV strains from France and Spain. J. Clin. Microbiol. 2007;45(2):559–63. DOI: https://doi.org/10.1128/jcm.01738-06
- Sergeev N., Rubtcova E., Chizikov V., et al. New mosaic subgenotype of varicella-zoster virus in the USA: VZV detection and genotyping by oligonucleotide-microarray. J. Virol. Methods. 2006;136(1-2):8–16. DOI: https://doi.org/10.1016/j.jviromet.2006.03.021
- Breuer J., Grose C., Norberg P., et al. A proposal for a common nomenclature for viral clades that form the species varicella-zoster virus: summary of VZV Nomenclature Meeting 2008, Barts and the London School of Medicine and Dentistry, 24-25 July 2008. J. Gen. Virol. 2010;91(Pt. 4):821–8. DOI: https://doi.org/10.1099/vir.0.017814-0
- Zell R., Taudien S., Pfaff F., et al. Sequencing of 21 varicella-zoster virus genomes reveals two novel genotypes and evidence of recombination. J. Virol. 2012;86(3):1608–22. DOI: https://doi.org/10.1128/jvi.06233-11
- Norberg P., Depledge D.P., Kundu S., et al. Recombination of globally circulating varicella-zoster virus. J. Virol. 2015; 89(14):7133–46. DOI: https://doi.org/10.1128/jvi.00437-15
- Jensen N.J., Rivailler P., Tseng H.F., et al. Revisiting the genotyping scheme for varicella-zoster viruses based on whole-genome comparisons. J. Gen. Virol. 2017;98(6):1434–8. DOI: https://doi.org/10.1099/jgv.0.000772
- Pontremoli C., Forni D., Clerici M., et al. Possible European origin of circulating varicella zoster virus strains. J. Infect. Dis. 2020;221(8):1286–94. DOI: https://doi.org/10.1093/infdis/jiz227
- Xu S., Chen M., Zheng H., et al. Nationwide distribution of varicella-zoster virus clades in China. BMC Infect. Dis. 2016; 16(1):542. DOI: https://doi.org/10.1186/s12879-016-1863-x
- Chen S., Zhou Y., Chen Y., Gu J. fastp: an ultra-fast all-in-one FASTQ preprocessor. Bioinformatics. 2018;34(17):i884–90. DOI: https://doi.org/10.1093/bioinformatics/bty560
- Li H., Durbin R. Fast and accurate short read alignment with Burrows–Wheeler transform. Bioinformatics. 2009;25(14): 1754–60. DOI: https://doi.org/10.1093/bioinformatics/btp324
- Li H., Handsaker B., Wysoker A., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 2009;25(16): 2078–9. DOI: https://doi.org/10.1093/bioinformatics/btp352
- Katoh K., Misawa K., Kuma K., Miyata T. MAFFT: a novel method for rapid multiple sequence alignment based on fast Fourier transform. Nucleic. Acids Res. 2002;30(14):3059–66. DOI: https://doi.org/10.1093/nar/gkf436