Бактерии рода Filifactor у больных пародонтитом и сахарным диабетом по данным метагеномного анализа микробиома пародонта
- Авторы: Царева Т.В.1, Янушевич О.О.1, Царев В.Н.1, Балмасова И.П.1
-
Учреждения:
- Московский государственный медико-стоматологический университет имени А.И. Евдокимова
- Выпуск: Том 100, № 6 (2023)
- Страницы: 485-494
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/16885
- DOI: https://doi.org/10.36233/0372-9311-428
- EDN: https://elibrary.ru/yqoxxk
- ID: 16885
Цитировать
Аннотация
Введение. Заболевания пародонта — распространённая патология, наиболее тяжёлой формой которой является хронический пародонтит. Проблема этого полимикробного заболевания в последние годы приобрела особое значение в связи с возможностью развития ассоциированных с ним системных эффектов. Довольно часто хронический пародонтит сочетается с сахарным диабетом 2-го типа (СД2). Основную роль в возникновении и развитии патологии пародонта играют бактерии, наименее изученным среди которых является относительно недавно открытый пародонтопатоген — Filifactor alocis.
Целью исследования являлось выявление бактерий рода Filifactor в составе микробиома пародонта при ассоциации хронического пародонтита и СД2 и уточнение механизмов их возможного влияния на ассоциированные метаболические процессы на основании сравнительного метагеномного анализа.
Материалы и методы. Проведено метагеномное исследование образцов микробиома пародонтальных карманов 28 пациентов с ассоциацией хронического пародонтита и СД2 и 22 пациентов с хроническим пародонтитом, а также микробиома зубодесневой борозды у 19 клинически здоровых людей. Для определения таксономического состава микробиома использовалось 16S-cеквенирование гена рибосомальной РНК, проводилось прогнозирование метаболических путей с участием микробиома методом дробовика.
Результаты. Полученные результаты позволили установить, что только при ассоциации хронического пародонтита и СД2 одними из наиболее часто встречающихся микроорганизмов были бактерии рода Filifactor, процент регистрации которых коррелировал с низкими показателями метагеномного прогнозирования биосинтеза жирных кислот и метаболизма пиримидина в очагах поражения.
Вывод. Частота встречаемости бактерий рода Filifactor у пациентов с ассоциацией хронического пародонтита и СД2 отрицательно коррелирует с отдельными особенностями предполагаемых метаболических путей микробиома, в число которых входят биосинтез жирных кислот и метаболизм пиримидина.
Полный текст
Введение
Заболевания пародонта широко распространены среди взрослого населения нашей планеты. Так, около 740 млн человек в мире страдают тяжёлой формой пародонтита [1]. Актуальность этой проблемы ещё более возрастает в связи с тем, что между заболеваниями полости рта и соматическими заболеваниями установлена в последние годы не только патогенетическая, но и причинно-следственная связь [2]. В литературе широко обсуждается вопрос о связи между пародонтитом и такими системными патологическими состояниями, как сахарный диабет 2-го типа (СД2), ревматоидный артрит, атеросклероз, неблагоприятные исходы беременности и др. [3–5]. Особое значение придаётся устойчивой ассоциации СД2 и хронического пародонтита (ХП) как двух заболеваний, имеющих наиболее широкую распространённость во всём мире [6, 7].
По прогнозам Международной федерации диабета, частота встречаемости сахарного диабета долго будет неуклонно возрастать и число заболевших к 2030 г. превысит 500 млн человек [8, 9]. Среди больных сахарным диабетом около 90% составляют пациенты с СД2 — неинсулинозависимым диабетом, обусловленным сочетанием падения секреции инсулина β-клетками поджелудочной железы и ростом устойчивости клеток к инсулину [10]. Многие исследователи показывают, что изменения метаболизма, ассоциированные с сахарным диабетом, проявляются на системном уровне и способствуют поражению многих органов и тканей, в том числе развитию ХП [11, 12], хотя также не исключается взаимное влияние этих патологических процессов [3, 5].
ХП имеет полимикробную природу, вызывается совокупностью микроорганизмов со свойствами пародонтопатогенов, характеризуется необратимостью течения, сопровождается хроническим воспалением пародонтальных тканей, их деструкцией с нарушением костной структуры и зубоальвеолярного связочного аппарата, что в конечном итоге приводит к потере зубов. Все основные возбудители этого заболевания (Porphyromonas gingivalis, Tannerella forsythia, Aggregatibacter actinomycetemcomitans, Filifactor alocis, Porphyromonas endodontalis, Treponema denticola, Prevotella intermedia, Fusobacterium nucleatum и др.), так называемые пародонтопатогены I и II порядка или, как их обозначали первоначально в зарубежной литературе, бактерии «красного» и «оранжевого» комплексов, находятся в сложнейших взаимодействиях между собой и с иммунной системой организма-хозяина, что провоцирует как развитие локальных изменений со стороны тканей пародонта, так и последствия этих воспалительно-дегенеративных изменений на системном уровне [3, 13, 14].
Благодаря развитию биотехнологий накопилось достаточно доказательств того, что именно микробиом ротовой полости при ХП служит основным фактором развития сахарного диабета [15], хотя в исследовании роли отдельных пародонтопатогенов в этом вопросе ещё существуют «белые пятна».
Большинство исследований взаимосвязи болезней пародонта c заболеваниями системного характера в первую очередь посвящены роли P. gingivalis, которая рассматривается как ключевой пародонтоген [3, 14]. В последние годы делается акцент на изучении других пародонтопатогенов I порядка («красного комплекса»). В дополнение к бактериям «красного комплекса» уделяется внимание значению других культивируемых бактерий (P. intermedia, F. nucleatum, Eykinella corrodens, Eubacterium nodatum, Wolinella recta), связанных с пародонтитом, а также некультивируемых (в условиях обычной клинической лаборатории) бактерий — Selenomonas, Sinergistes, Desulfobulbus, TM7, F. alocis, идентифицированных в качестве новых потенциальных патогенов или пародонтопатогенов II порядка [3, 13, 16, 17].
Один из представителей этих видов, F. alocis, является грамположительной споронеобразующей палочкой c облигатно-анаэробным типом дыхания, формирующей нитевидные скопления в биоплёнке десны, которая чрезвычайно медленно растёт на питательных средах и обладает низкой способностью проявлять биохимическую активность, что затрудняет её идентификацию [18, 19]. F. alocis — асахаролитическая бактерия, использующая для своего роста определённые аминокислоты, включая аргинин [20].
Основным местом обитания F. alocis в организме человека являются десневая борозда и, вероятно, кишечник, при этом микробиом здорового человека ротовой полости, как правило, не содержит данный микроорганизм (за исключением курящих людей), в отличие от людей, страдающих заболеваниями пародонта [20]. Бактерии F. alocis обладают уникальными свойствами, такими как устойчивость к окислительному стрессу, способность к индукции системной потери костной массы, наличие генов, кодирующих хорошо развитый путь метаболизма аминокислот, что позволяет им колонизировать ткани пародонта, вызывать, подобно другим традиционным пародонтопатогенам, явления пародонтита в стрессовой среде формирующегося пародонтального кармана [21–23]. Эти особенности F. alocis в дополнение к способности взаимодействовать с другими видами микробов, формируя полимикробные синергетические отношения, могут усиливать инвазивные качества данных бактерий [24] и вызывать хроническое воспаление [25]. F. alocis может проникать внутрь эпителиальных клеток десны и индуцировать секрецию ими провоспалительных цитокинов, что способствует проявлениям патогенности этого микроба [26].
В совокупности все эти наблюдения указывают на специфическую роль F. alocis в полости рта, которая может иметь значение в патологическом процессе [27, 28]. Из-за отсутствия генетических инструментов для изучения этого микроорганизма долгое время было мало что известно о его способности влиять на микробный метаболизм в биоплёнке, о механизмах вирулентности и, особенно, роли в индукции системных эффектов [21].
В соответствии с этим целью настоящего исследования являлось выявление бактерий рода Filifactor в составе микробиома пародонта при ассоциации ХП и СД2 и уточнение механизмов их возможного влияния на ассоциированные метаболические процессы на основании метагеномного анализа.
Mатериалы и методы
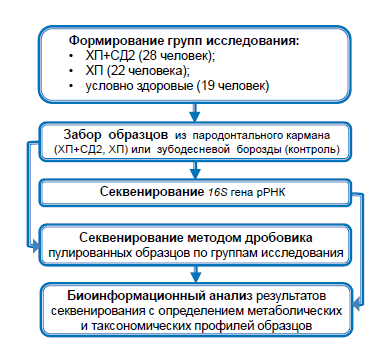
Под наблюдением находились 69 человек в возрасте 40–65 лет. Основная группа включала 28 пациентов с ассоциацией ХП и СД2; группа сравнения состояла из 22 пациентов с ХП без сопутствующей соматической патологии; в контрольную группу были включены 19 условно здоровых лиц с интактным пародонтом, без клинически выраженных проявлений соматической патологии, с нормальным уровнем сахара и гликированного гемоглобина в крови (рис. 1). Число женщин и мужчин в группах сравнения было сопоставимым: 13 (46,5%) и 15 (53,5%) — в группе ХП+СД2, 10 (45,4%) и 12 (54,6%) — в группе ХП, 9 (47,4%) и 10 (2,6%) — в контрольной группе.
Рис. 1. Дизайн исследования.
Fig. 1. Study design.
В связи с необходимостью использования для метагеномных исследований пулированных (объединённых) образцов биологического материала требовалась максимальная однородность групп исследования, поэтому из исследования были исключены больные с лёгким и тяжёлым течением патологических процессов, т.е. все больные имели среднетяжёлое течение изучаемых заболеваний. Пациенты не получали лечения по поводу ХП в течение последних 6 мес. Среди больных СД2 11 человек получали инсулинотерапию, 13 человек — пероральные сахароснижающие препараты, 4 человека — комбинированную терапию, что учитывалось при распределении образцов для последующего пулирования.
ХП диагностировали на основе клинико-рентгенологических данных в соответствии с классификацией болезней пародонта 2018 г. [29]. Критерии включения этих пациентов в исследование: пародонтит средней степени тяжести с генерализованным поражением, значением потери прикрепления десны CAL 3–4 мм, глубиной карманов зондирования 4–6 мм, потерей костной ткани вокруг зубов не более 1/3 длины корня, отсутствием потери зубов, связанных с заболеванием. Для подтверждения диагноза проводили анализ данных анамнеза, определение РHP (Patient Hygiene Performance), OHI-S (упрощённый индекс гигиены полости рта), CAL (клинический уровень прикрепления), MMI (индекс мобильности Миллера), PBI (индекс папиллярного кровотечения), TL (потеря зубов), данных ортопантомографии.
Диагноз СД2 устанавливался эндокринологом в соответствии с диагностическими критериями Всемирной организации здравоохранения 1999/2006/2011 [30] с учётом клинических, анамнестических и лабораторных данных. Критерии включения в исследование: длительность заболевания 3–7 лет; течение заболевания умеренно компенсированное; стадия ремиссии; уровень глюкозы крови менее 7,8 ммоль/л, уровень гликированного гемоглобина менее 8%.
Условно здоровые люди группы контроля включались в исследование только после консультации со стоматологом и эндокринологом.
Материалом для исследования служило содержимое пародонтальных карманов у больных с ХП и зубодесневой борозды у здоровых субъектов. Материал забирали из 4 участков в области зубодесневой борозды с помощью стерильных бумажных эндодонтических штифтов (№ 30), которые помещали в пробирку с 0,2 мл стерильного физиологического раствора и встряхивали. Образцы хранились при температуре –20ºC. Исследование проводилось при добровольном информированном согласии пациентов. Протокол исследования одобрен Межвузовским комитетом по этике г. Москвы (протокол № 06-22 от 16.06.2022).
Тотальную ДНК экстрагировали из образцов с использованием набора «QIAamp DNA Investigator Kit» («Qiagen»), содержание геномной ДНК определяли на флуорометре «Qubit 2.0» («Invitrogen») в соответствии с инструкциями производителя. Обогащённую микробную ДНК (50–100 нг) фрагментировали с помощью системы «Covaris S220» («Covaris»). Конечный размер фрагмента определяли с использованием биоанализатора «Agilent 2100» («Agilent Technologies») в соответствии с инструкциями производителя. Выделенную ДНК амплифицировали с применением стандартных праймеров для гена 16S рибосомальной РНК (рРНК), комплементарных области V3–V4 и содержащих адаптерные последовательности 5'-illumina.
Секвенирование библиотек и анализ полученных данных осуществляли с использованием генетического анализатора «MiSeq» («Illumina») и «MiSeq Reagent Kit v2» («Фарма»).
Для проведения таксономического анализа результатов секвенирования вариабельных участков гена 16S рРНК были применены биоинформационная платформа микробиома QIIME2 (Quantitative Insights into Microbial Ecology) [31] и база данных SILVA [32]. Выявление различий на уровне таксономических композиций проводилось путём дисперсионного анализа данных на основе теста PERMANOVA для микробных сообществ из функционала QIIME2 [33]. Для прогнозирования метаболического профиля по данным сиквенса и профилирования гена 16S рРНК был использован метод PICRUST2 (Phylogenetic Investigation of Communities by Reconstruction of Unobserved States) [34], а для выявления статистических различий между группами на уровне предсказанных метаболических путей применяли статистический анализ метаболических профилей STAMP [35]. Данные метагеномного анализа визуализировали с помощью программного обеспечения GNU/R и пакета Vegan [36].
Результаты
На первом этапе проводили секвенирование гена 16S малой субъединицы рРНК, которое позволило определить представительство различных таксономических категорий бактерий в составе образцов из содержимого зубодесневой борозды пациентов.
Проведённый биоинформационный анализ выявил довольно значимые и не отмеченные другими исследователями особенности микробного состава содержимого зубодесневой борозды при ХП, ассоциированном с СД2: значительное преобладание в составе микробиоты пациентов 5 таксономических групп бактерий — представителей родов Filifactor и Mycoplasma, семейств Flavobacteriaceae и Porphyromonadaceae, порядка Bacillales — и более низкой встречаемости 3 групп: TM7 как ассоциантов актиномицетов, родов Atopobium и Fusobacterium (рис. 2).
Рис. 2. Таксономический профиль микробиома пародонтальных карманов при сравнении групп исследования.
Fig. 2. Taxonomic profile of periodontal pocket microbiome in comparison of study groups.
Сравнительный анализ таксономического профиля микробиома пациентов с ХП, ассоциированным и не ассоциированным с СД2, показал более частую встречаемость в первой группе бактерий родов Filifactor и Treponema и более низкую частоту регистрации присутствия в микробиоме 5 таксономических групп: родов Bordetella и Atopobium, семейств Fusobacteriaceae и Veillonellaceae, ТМ7.
У пациентов с ХП без соматической патологии по сравнению со здоровыми людьми более часто встречались бактерии 5 таксономических групп: родов Anaerosinus, Bulleidia, а также порядков Bacillales, Bacteroidales, Macellibacteroides.
В целом основной особенностью таксономического профиля микробиома зубодесневой борозды при ассоциации ХП+СД2 является преобладание в составе биологического материала бактерий с пародонтопатогенными свойствами, принадлежащих к родам Filifactor и Treponema. Представляет определённый интерес и требует дополнительного анализа более частая встречаемость по сравнению со здоровыми людьми прокариот рода Mycoplasma, семейства Flavobacteriaceae и порядка Bacillales.
Особого внимания заслуживает тот факт, что наиболее значимой особенностью группы ХП+СД2 является преобладающее представительство в составе микробиома бактерий рода Filifactor. По этому признаку микробиом указанной группы отличается и от группы здоровых людей, и от группы пациентов с ХП среднетяжёлого течения, в которых бактерии рода Filifactor встречаются в единичных случаях. Возникает только вопрос, насколько эта особенность связана с развитием сахарного диабета и теми метаболическими сдвигами, которые наблюдаются при СД2 и ассоциированы преимущественно с действием микробиома.
На следующем этапе выполнялось секвенирование пулированных образцов с определением количественного преобладания отдельных таксономических категорий бактерий в составе микробиома по отдельным пулам биологического материала с функциональными признаками, позволяющими предсказать основные метаболические пути анализируемых пулированных образцов микробиома, принадлежащих разным клиническим группам (по 15 пулированных образцов в каждой группе).
Различия по таксономическому богатству метагеномов в полной мере отразились и на уровне различий в предсказанном функциональном потенциале микробных сообществ по группам исследования (рис. 3).
Рис. 3. Прогнозируемый профиль метаболических путей микробиома пародонтальных карманов при сравнении групп исследования.
Fig. 3. Predicted profile of periodontal pocket microbiome metabolic pathways in the comparison of the clinical study groups.
В группе ХП+СД2 выявлено снижение по сравнению с контролем относительной представленности 8 метаболических путей: биосинтеза жирных кислот, метаболизма пиримидина, метаболизма метана, метаболизма глицеролипидов, метаболизма сфинголипидов, метаболизма тирозина, метаболизма аскорбата и альдарата, биосинтеза убихинона и других терпеноид-хинонов.
При сравнении групп ХП, ассоциированного и не ассоциированного с СД2, выявлены различия в представленности 5 метаболических путей: в группе ХП+СД2 отмечено повышение метаболизма цистеина и метионина и снижение метаболизма пиримидина, биосинтеза жирных кислот, метаболизма метана, метаболизма сфинголипидов.
Для группы ХП в сравнении с контролем установлено снижение относительной представленности 4 метаболических путей: цистеина и метионина, серы, гистидина, глицеролипидов.
В конечном итоге можно выделить 4 метаболических пути, низкий уровень которых отличает группу ХП+СД2, от контроля и группы ХП: метаболизм метана, метаболизм сфинголипидов, биосинтез жирных кислот, метаболизм пиримидина. Для ХП, независимо от ассоциации с СД2, было характерно снижение метаболизма глицеролипидов.
Функциональный анализ пулированных образцов с использованием критерия Bray–Curtis подтвердил, что в целом группа больных ХП по характеристике метаболических путей была значительно ближе к контролю, чем группа ХП+СД2.
Для определения взаимосвязи выявленных особенностей метаболических профилей по группам исследования и наличия в составе микробиома пародонтальных карманов представителей рода Filifactor был проведён корреляционный анализ с использованием коэффициента корреляции Спирмена (r). Частота встречаемости этих бактерий при ХП+СД2 в наших исследованиях показывала достоверный уровень отрицательной корреляции (r > 0,723) с прогнозированием двух метаболических путей — биосинтеза жирных кислот и метаболизма пиримидина, притом что все эти признаки у пациентов данной группы регистрировались в 3–5 раз реже, чем в остальных случаях.
Обсуждение
Установленная нами наиболее высокая встречаемость бактерий рода Filifactor, относительно недавно причисленных к группе пародонтопатогенов I порядка [19], при ассоциации ХП+СД2 отмечена и другими авторами в числе преобладающих представителей микробиома пародонтальных карманов [37–40], хотя и без детального сравнения встречаемости этого микроорганизма в других группах.
Полученные нами данные об особенностях метаболизма микробиома пародонтальных карманов у больных с ХП+СД2 свидетельствуют о довольно значительной роли нарушений обмена жирных кислот, пиримидина, метана, глицеролипидов, сфинголипидов. Два из этих признаков (снижение биосинтеза жирных кислот и метаболизма пиримидина) оказались корреляционно связаны с наличием в составе микробиома бактерий рода Filifactor. Факт наличия такой корреляции отмечен нами впервые, в то же время значение указанных метаболических путей микробиома при ассоциации ХП+СД2 обсуждается в современной научной литературе.
Так, при оценке метаболизма F. alocis выделяется значение биосинтеза этими бактериями насыщенной жирной кислоты — масляной кислоты, участвующей в патогенезе пародонтита [41]. Более того, предлагается даже использовать микробную масляную кислоту в качестве маркера пародонтита, при этом подчёркивается, что это соединение может влиять на чувствительность организма к инсулину [42]. Существует точка зрения, что среда полости рта при ХП+СД2 может характеризоваться уменьшением количества бактерий, продуцирующих масляную кислоту [43]. Это входит в некоторое противоречие с данными нашего исследования, по крайней мере, по значительному представительству рода Filifactor в микробиоме при рассматриваемой патологии. В то же время установленный нами дефицит жирных кислот при ассоциации ХП+СД2 может иметь и другое объяснение. Например, если имеет место присутствие в микробиоме субгингивальной биоплёнки герпесвирусов (особенно вируса Эпштейна–Барр), то они обладают способностью активно потреблять ту же масляную кислоту для своей реактивации [44]. Bcё это показывает возможность неоднозначного трактования полученных результатов и необходимость дальнейших исследований в данном направлении.
Установленное нами снижение метаболизма пиримидина, коррелирующее с встречаемостью F. alocis, тоже отмечено в научной литературе как признак ХП. Например, имеются сведения о том, что гены, участвующие в синтезе пиримидина, имели значительно более низкую относительную численность у пациентов с пародонтитом по сравнению со здоровыми [45]. Пиримидин является агонистом рецептора GR119, связанного с гипогликемическим воздействием и протективным эффектом в отношении β-клеток поджелудочной железы, что позволяет даже рекомендовать производные пиримидина в качестве лечебных средств при СД2 [46]. C этой точки зрения состояние метаболизма пиримидина у микробиома тканей пародонта с участием F. alocis в перспективе может оказаться своеобразным маркером ассоциации пародонтита с сахарным диабетом, а возможно, и одним из многочисленных элементов патогенеза СД2.
Таким образом, дальнейшее исследование этих феноменов перспективно не только с точки зрения расшифровки этиопатогенетической роли бактерий рода Filifactor, но и с позиций совершенствования способов диагностики и лечения обозначенной сочетанной патологии.
Выводы
- Микробиом тканей пародонта при ассоциации ХП+СД2 обладает рядом особенностей, касающихся как его функциональных признаков, так и таксономического состава. В последнем случае особое внимание исследователей привлекают недавно установленные и пока мало изученные бактериальные представители микробиома, плохо культивируемые на питательных средах и обладающие выраженными пародонтопатогенными свойствами. К таким микроорганизмам относятся, в частности, бактерии вида F. alocis.
- При метагеномном анализе микробиома пародонтальных карманов пациентов установлено, что бактерии рода Filifactor встречаются в составе микробиома пациентов с ХП+СД2 значительно чаще, чем при наличии только пародонтита или у здоровых людей. Частота встречаемости бактерий рода Filifactor у пациентов с ассоциацией ХП+СД2 отрицательно коррелировала с отдельными особенностями предполагаемых метаболических путей микробиома, в число которых входили биосинтез жирных кислот и метаболизм пиримидина.
- Отмеченные метаболические признаки, в частности снижение метаболизма пиримидина, коррелирующее с встречаемостью F. alocis, по данным литературы, с определённой долей вероятности могут считаться патогенетическим фактором развития пародонтита и/или СД2 и рассматриваться как диагностический маркер.
Об авторах
Татьяна Викторовна Царева
Московский государственный медико-стоматологический университет имени А.И. Евдокимова
Email: tancha-leo84@mail.ru
ORCID iD: 0000-0001-9571-0520
SPIN-код: 2028-8404
Scopus Author ID: 39762981000
https://www.msmsu.ru/obrazovanie/teaching-staff/2952/
к.м.н., доцент кафедры микробиологии, вирусологии, иммунологии
Россия, МоскваОлег Олегович Янушевич
Московский государственный медико-стоматологический университет имени А.И. Евдокимова
Email: msmsu@msmsu.ru
ORCID iD: 0000-0003-0059-4980
SPIN-код: 1452-1387
Scopus Author ID: 57371027600
https://www.msmsu.ru/university/rektorat/12/
д.м.н., профессор, академик РАН, зав. каф. пародонтологии, ректор
Россия, МоскваВиктор Николаевич Царев
Московский государственный медико-стоматологический университет имени А.И. Евдокимова
Email: nikola777@rambler.ru
ORCID iD: 0000-0002-3311-0367
SPIN-код: 8180-4941
Scopus Author ID: 7005569282
https://www.msmsu.ru/obrazovanie/teaching-staff/1303/
д.м.н., профессор, зав. каф. микробиологии, вирусологии, иммунологии
Россия, МоскваИрина Петровна Балмасова
Московский государственный медико-стоматологический университет имени А.И. Евдокимова
Автор, ответственный за переписку.
Email: iri.balm@mail.ru
ORCID iD: 0000-0001-8194-2419
SPIN-код: 8025-8611
https://www.msmsu.ru/obrazovanie/teaching-staff/6807/
д.м.н., профессор, зав. лаб. патогенеза и методов лечения инфекционных заболеваний
Россия, МоскваСписок литературы
- Eke P.I., Wei L., Borgnakke W.S., et al. Periodontitis prevalence in adults ≥65years of age, in the USA. Periodontol. 2000. 2016;72(1):76–95. DOI: https://doi.org/10.1111/prd.12145
- Мамедов М.Н., Куценко В.А., Керимов Э.Ф. и др. Связь состояния соматического здоровья с заболеваниями тканей пародонта и зубов в реальной клинической практике. Профилактическая медицина. 2022;25(2):66–73. Mamedov M.N., Kutsenko V.A., Kerimov E.F., et al. Relationship between the state of somatic health and diseases of periodontal tissues and teeth in real clinical practice. The Russian Journal of Preventive Medicine. 2022;25(2):66–73. DOI: https://doi.org/10.17116/profmed20222502166 EDN: https://elibrary.ru/xckwlj
- Балмасова И.П., Царев В.Н., Янушевич О.О. и др. Микроэкология пародонта. Взаимосвязь локальных и системных эффектов. М.; 2021:175–225. Balmasova I.P., Tsarev V.N., Yanushevich O.O., et al. Microecology of Periodontium. The Relationship Between Local and Systemic Effects. Moscow; 2021:175–225. EDN: https://elibrary.ru/myzmbu
- Hajishengallis G., Chavakis T. Local and systemic mechanisms linking periodontal disease and inflammatory comorbidities. Nat. Rev. Immunol. 2021;21(7):426–40. DOI: https://doi.org/10.1038/s41577-020-00488-6
- Nikolaeva E.N., Tsarev V.N., Tsareva T.V., et al. Interrelation of cardiovascular diseases with anaerobic bacteria of subgingival biofilm. Contemp. Clin. Dent. 2019;10(4):637–42. DOI: https://doi.org/10.4103/ccd.ccd_84_19
- Bascones-Martínez A., Muñoz-Corcuera M., Bascones-Ilundain J. Diabetes and periodontitis: A bidirectional relationship. Med. Clin. (Barc.). 2015;145(1):31–5. DOI: https://doi.org/10.1016/j.medcli.2014.07.019 (in Spanish)
- Portes J., Bullón B., Quiles J.L., et al. Diabetes mellitus and periodontitis share intracellular disorders as the main meeting point. Cells. 2021;10(9):2411. DOI: https://doi.org/10.3390/cells10092411
- Alam U., Asghar O., Azmi S., Malik R.A. General aspects of diabetes mellitus. Handb. Clin. Neurol. 2014;126:211–22. DOI: https://doi.org/10.1016/b978-0-444-53480-4.00015-1
- Ida S., Kaneko R., Murata K. Utility of real-time and retrospective continuous glucose monitoring in patients with type 2 diabetes mellitus: A meta-analysis of randomized controlled trials. J. Diabetes Res. 2019;2019:4684815. DOI: https://doi.org/10.1155/2019/4684815
- American Diabetes Association. Classification and diagnosis of diabetes: standards of medical care in diabetes – 2021. Diabetes Care. 2021;44(Suppl. 1):15–33. DOI: https://doi.org/10.2337/dc21-s002
- Patil V.S., Patil V.P., Gokhale N., et al. Chronic periodontitis in type 2 diabetes mellitus: oxidative stress as a common factor in periodontal tissue injury. J. Clin. Diagn. Res. 2016;10(4):BC12–6. DOI: https://doi.org/10.7860/jcdr/2016/17350.7542
- Унаньян К.Г., Балмасова И.П., Царев В.Н. и др. Церамиды как биомаркеры хронического пародонтита, ассоциированного с сахарным диабетом второго типа. Вестник Российского университета дружбы народов. Серия: Медицина. 2020;24(4):325–37. Unanyan K.G., Balmasova I.P., Tsarev V.N., et al. Ceramides as biomarkers of chronic periodontitis associated with type II diabetes. RUDN Journal of Medicine. Series: Medicine. 2020;24(4):325–37. DOI: https://doi.org/10.22363/2313-0245-2020-24-4-325-337 EDN: https://elibrary.ru/kykdxt
- Царев В.Н., Николаева Е.Н., Ипполитов Е.В. Пародонтопатогенные бактерии как основные факторы возникновения и развития пародонтита. Журнал микробиологии, эпидемиологии, иммунобиологии. 2017;94(5):101–12. Tsarev V.N., Nikolaeva E.N., Ippolitov E.V. Periodontal pathogenic bacteria as the main factors in the occurrence and development of periodontitis. Journal of Microbiology, Epidemiology and Immunobiology. 2017;94(5):101–12. DOI: https://doi.org/10.36233/0372-9311-2017-5-101-112 EDN: https://elibrary.ru/ctbcar
- Howard K.C., Gonzalez O.A., Garneau-Tsodikova S. Porphyromonas gingivalis: where do we stand in our battle against this oral pathogen? RSC Med. Chem. 2021;12(5):666–704. DOI: https://doi.org/10.1039/d0md00424c
- Zhou M., Rong R., Munro D., et al. Investigation of the effect of type 2 diabetes mellitus on subgingival plaque microbiota by high-throughput 16S rDNA pyrosequencing. PLoS One. 2013;8(4):e61516. DOI: https://doi.org/10.1371/journal.pone.0061516
- Dewhirst F.E., Chen T., Izard J., et al. The human oral microbiome. J. Bacteriol. 2010;192(19):5002–17. DOI: https://doi.org/10.1128/jb.00542-10
- Griffen A.L., Beall C.J., Campbell J.H., et al. Distinct and complex bacterial profiles in human periodontitis and health revealed by 16S pyrosequencing. ISME J. 2012;6(6):1176–85. DOI: https://doi.org/10.1038/ismej.2011.191
- Jalava J., Eerola E. Phylogenetic analysis of Fusobacterium alocis and Fusobacterium sulci based on 16S rRNA gene sequences: proposal of Filifactor alocis (Cato, Moore and Moore) comb. nov. and Eubacterium sulci (Cato, Moore and Moore) comb. nov. Int. J. Syst. Bacteriol. 1999;49(Pt. 4):1375–9. DOI: https://doi.org/10.1099/00207713-49-4-1375
- Янушевич О.О., Царев В.Н., Николаева Е.Н. и др. Первый отечественный опыт выявления ассоциации анаэробных бактерий Filifactor alocis и Porphyromonas gingivalis молекулярно-биологическими методами при заболеваниях пародонта и коморбидной патологии (сравнительное исследование). Вестник Российской академии медицинских наук. 2022;77(6):437–46. Yanushevich O.O., Tsarev V.N., Nikolaeva E.N., et al. The first domestic experience of detecting the association of anaerobic bacteria Filifactor alocis and Porphyromonas gingivalis by molecular biological methods in periodontal diseases and comorbid pathology (comparative research). Annals of the Russian academy of medical sciences. 2022;77(6):437–46. DOI: https://doi.org/10.15690/vramn2262 EDN: https://elibrary.ru/pzajok
- Uematsu H., Sato N., Hossain M.Z., et al. Degradation of arginine and other amino acids by butyrate-producing asaccharolytic anaerobic G am-positive rods in periodontal pockets. Arch. Oral Biol. 2003;48(6):423–9. DOI: https://doi.org/10.1016/s0003-9969(03)00031-1
- Aruni A.W., Roy F., Fletcher H.M. Filifactor alocis has virulence attributes that can enhance its persistence under oxidative stress conditions and mediate invasion of epithelial cells by Porphyromonas gingivalis. Infect. Immun. 2011;79(10):3872–86. DOI: https://doi.org/10.1128/iai.05631-11
- Moffatt C.E., Whitmore S.E., Griffen A.L., et al. Filifactor alocis interactions with gingival epithelial cells. Mol. Oral Microbiol. 2011;26(6):365–73. DOI: https://doi.org/10.1111/j.2041-1014.2011.00624.x
- Kim H.Y., Song M.K., Gho Y.S., et al. Extracellular vesicles derived from the periodontal pathogen Filifactor alocis induce systemic bone loss through Toll-like receptor 2. J. Extracell. Vesicles. 2021;10(12):e12157. DOI: https://doi.org/10.1002/jev2.12157
- Aruni A.W., Zhang K., Dou Y., Fletcher H. Proteome analysis of coinfection of epithelial cells with Filifactor alocis and Porphyromonas gingivalis shows modulation of pathogen and host regulatory pathways. Infect. Immun. 2014;82(8):3261–74. DOI: https://doi.org/10.1128/iai.01727-14
- Fine D.H., Markowitz K., Fairlie K., et al. A consortium of Aggregatibacter actinomycetemcomitans, Streptococcus parasanguinis and Filifactor alocis are present in sites prior to bone loss in a longitudinal study of localized aggressive periodontitis. J. Clin. Microbiol. 2013;51(9):2850–61. DOI: https://doi.org/10.1128/jcm.00729-13
- Aja E., Mishra A., Dou Y., Fletcher H.M. Role of the Filifactor alocis hypothetical protein FA519 in oxidative stress resistance. Microbiol. Spectr. 2021;9(3):e0121221. DOI: https://doi.org/10.1128/spectrum.01212-21
- Armstrong C.L., Miralda I., Neff A.C., et al. Filifactor alocis promotes neutrophil degranulation and chemotactic activity. Infect. Immun. 2016;84(12):3423–33. DOI: https://doi.org/10.1128/iai.00496-16
- Aruni A.W., Mishra A., Dou Y., et al. Filifactor alocis a new emerging periodontal pathogen. Microbes Infect. 2015;17(7):517–30. DOI: https://doi.org/10.1016/j.micinf.2015.03.011
- Graetz C., Mann L., Krois J., et al. Comparison of periodontitis patients’ classification in the 2018 versus 1999 classification. J. Clin. Periodontol. 2019;46(9):908–17. DOI: https://doi.org/10.1111/jcpe.13157
- American Diabetes Association. Diagnosis and classification of diabetes mellitus. Diabetes Care. 2012;35(Suppl. 1):S64–71. DOI: https://doi.org/10.2337/dc12-s064
- Callahan B.J., McMurdie P.J., Rosen M.J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nat. Methods. 2016;13(7):581–3. DOI: https://doi.org/10.1038/nmeth.3869
- Quast C., Pruesse E., Yilmaz P., et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools. Nucleic Acids Res. 2013;41(Database):D590–6. DOI: https://doi.org/10.1093/nar/gks1219
- Tang Z.Z., Chen G., Alekseyenko A.V. PERMANOVA-S: association test for microbial community composition that accommodates confounders and multiple distances. Bioinformatics. 2016;32(17):2618–25. DOI: https://doi.org/10.1093/bioinformatics/btw311
- Langille M.G., Zaneveld J., Caporaso J.G., et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nat. Biotechnol. 2013;31(9):814–21. DOI: https://doi.org/10.1038/nbt.2676
- Parks D.H., Tyson G.W., Hugtnholtz P., Beiko R.G. STAMP: statistical analysis of taxonomic and functional profiles. Bioinformatics. 2014;30(21):3123–4. DOI: https://doi.org/10.1093/bioinformatics/btu494
- Saeb A.T.M., Al-Rubeaan K.A., Aldosary K., et al. Relative reduction of biological and phylogenetic diversity of the oral microbiota of diabetes and pre-diabetes patients. Microb. Pathog. 2019;128:215–29. DOI: https://doi.org/10.1016/j.micpath.2019.01.009
- Casarin R.C., Barbagallo A., Meulman T., et al. Subgingival biodiversity in subjects with uncontrolled type-2 diabetes and chronic periodontitis. J. Periodontal Res. 2013;48(1):30–6. DOI: https://doi.org/10.1111/j.1600-0765.2012.01498.x
- Tam J., Hoffmann T., Fischer S., et al. Obesity alters composition and diversity of the oral microbiota in patients with type 2 diabetes mellitus independently of glycemic control. PLoS One. 2018;13(10):e0204724. DOI: https://doi.org/10.1371/journal.pone.0204724
- Tang B., Yan C., Shen X., Li Y. The bidirectional biological interplay between microbiome and viruses in periodontitis and type 2 diabetes mellitus. Front. Immunol. 2022;13:885029. DOI: https://doi.org/10.3389/fimmu.2022.885029
- Ganesan S.M., Joshi V., Fellows M., et al. A tale of two risks: smoking, diabetes and the subgingival microbiome. ISME J. 2017;11(9):2075–89. DOI: https://doi.org/10.1038/ismej.2017.73
- Uematsu H., Sato N., Hossain M.Z., et al. Degradation of arginine and other amino acids by butyrate-producing asaccharolytic anaerobic Gram-positive rods in periodontal pockets. Arch. Oral Biol. 2003;48(6):423–9. DOI: https://doi.org/10.1016/s0003-9969(03)00031-1
- Cueno M.E., Ochiai K. Gingival periodontal disease (PD) level-butyric acid affects the systemic blood and brain organ: Insights into the systemic inflammation of periodontal disease. Front. Immunol. 2018;9:1158. DOI: https://doi.org/10.3389/fimmu.2018.01158
- Hajishengallis G. Immunomicrobial pathogenesis of periodontitis: keystones, pathobionts, and host response. Trends Immunol. 2014;35(1):3–11. DOI: https://doi.org/10.1016/j.it.2013.09.001
- Imai K., Ogata Y. How does Epstein-Barr virus contribute to chronic periodontitis? Int. J. Mol. Sci. 2020;21(6):1940. DOI: https://doi.org/10.3390/ijms21061940
- Li Y., He J., He Z., et al. Phylogenetic and functional gene structure shifts of the oral microbiomes in periodontitis patients. ISME J. 2014;8(9):1879–91. DOI: https://doi.org/10.1038/ismej.2014.28
- Fang Y., Xu J., Li Z., et al. Design and synthesis of novel pyrimido[5,4-d]pyrimidine derivatives as GPR119 agonist for treatment of type 2 diabetes. Bioorg. Med. Chem. 2018;26(14):4080–7. DOI: https://doi.org/10.1016/j.bmc.2018.06.035
Дополнительные файлы