Геномные особенности резистентных изолятов Klebsiella pneumoniae, выделенных из кровяного русла и ликвора пациентов детского стационара
- Авторы: Воронина О.Л.1, Кунда М.С.1, Рыжова Н.Н.1, Аксенова Е.И.1, Садеева З.З.2, Новикова И.Е.2, Лазарева А.В.2, Карасева О.В.2,3, Фисенко А.П.2, Гинцбург А.Л.1
-
Учреждения:
- Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
- Национальный медицинский исследовательский центр здоровья детей
- Научно-исследовательский институт неотложной детской хирургии и травматологии
- Выпуск: Том 100, № 6 (2023)
- Страницы: 399-409
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/16594
- DOI: https://doi.org/10.36233/0372-9311-430
- EDN: https://elibrary.ru/ylxbdz
- ID: 16594
Цитировать
Аннотация
Введение. Klebsiella pneumoniae, продуцирующие карбапенемазы и относящиеся к международным клонам особого риска, адаптируясь к условиям стационара, эволюционируют в конвергентные патотипы. Такие варианты сочетают признаки двух генетических линий: множественно резистентной (MDR) и гипервирулентной. Патотипы, наряду с MDR K. pneumoniae, при системной инфекции представляют особую угрозу для пациентов раннего возраста.
Цель исследования — подробный молекулярно-генетический анализ MDR-изолятов K. pneumoniae, выявленных при мониторинге резистентных грамотрицательных бактерий в НМИЦ здоровья детей в 2014–2021 гг.
Материалы и методы. Проведено полногеномное секвенирование с последующим биоинформационным анализом 8 MDR-изолятов из крови и ликвора.
Результаты. MDR-изоляты относились к 4 сублиниям (SL): SL307, SL395, SL29 и SL1198. В геномах 6 пан-резистентных (PDR) изолятов выявили гены устойчивости ко всем категориям антибиотиков, рекомендованных для терапии Enterobacteriaceae. Плазмиды, присутствовавшие во всех геномах, помимо генов антибиотикорезистентности, в 6 изолятах содержали опероны устойчивости к ионам тяжёлых металлов. В переносе генов резистентности участвовали также профаги в составе плазмид. Изолят ST395 из ликвора по показателям резистентности и вирулентности относился к конвергентному патотипу. Сопоставление геномов внутри SLs выявило рекомбинационные события в областях K- и O-локусов и оперона иерсиниябактина.
Заключение. В выборке резистентных изолятов K. pneumoniae, выделенных из кровотока и ликвора, обнаружили 6 изолятов PDR, один из которых относится к конвергентному патотипу ST395.
Полный текст
Введение
Среди энтеробактерий, ставших причиной внутрибольничных инфекций, Klebsiella pneumoniae занимает лидирующее положение в России [1]. Вызывает обеспокоенность рост доли устойчивых к карбапенемам K. pneumoniae и выявление изолятов, в геномах которых одновременно присутствуют гены карбапенемаз разных классов (A, B, D) [1] (carbapenemase-producing, CP-Kp). По данным за 2020 г., CP-Kp в России принадлежали клональным группам (CG, clonal group) CG395, CG11, CG147 и CG307 [1], относящимся к международным клонам особого риска. CP-Kp ST395 превалируют при инфекциях кровотока у пациентов с гематологическими злокачественными новообразованиями (37,7%) [2], пациентов нейрохирургии и отделений реанимации и интенсивной терапии (27,1%) [3], вызывают тяжёлые системные инфекции [4]. Национальная система мониторинга антибиотикорезистентности AMRmap1 позволила выявить с 2013 по 2018 г. 108 изолятов CP-Kp ST395, вошедших в международное геномное исследование, которое показало появление в CG395 конвергентного патотипа, сочетающего маркеры резистентности и гипервирулентности [5]. Впервые о конвергенции признаков двух генетических линий K. pneumoniae: множественно резистентной (Multy Drug Resistance, MDR) и гипервирулентной (hvKp) сообщили D. Gu и соавт. в отношении изолята ST11 [6]. Р. Starkova и соавт. исследовали конвергентные (CP-hvKp) изоляты ST15, ST147, ST395, ST874, выделенные в клиниках Санкт-Петербурга, и показали наличие в них гибридных плазмид, несущих гены вирулентности и NDM β-лактамазу [7]. Наряду с плазмидами, другие мобильные генетические элементы: фаги, интегроны, транспозоны, способствуют эволюции геномов бактерий [8]. Конъюгативным транспозонам (интегративным конъюгативным элементам, ICE) отводится особая роль в горизонтальном транспорте генов K. pneumoniae [9].
Высокая скорость адаптации и эволюции K. pneumoniae в условиях стационаров представляет особую угрозу для пациентов раннего возраста, нуждающихся в оперативном вмешательстве. Инфекции кровотока у таких пациентов приводят к неблагоприятному клиническому прогнозу [10]. В задачу нашего исследования входил анализ MDR-изолятов K. pneumoniae, выделенных из крови и ликвора пациентов НМИЦ здоровья детей в 2014–2021 гг.
Материалы и методы
Материалы
Изучали 8 культур K. pneumoniae (табл. 1), выделенных из кровотока и ликвора в НМИЦ здоровья детей и НИИ неотложной детской хирургии и травматологии в ходе мониторинга лекарственной устойчивости внутрибольничных изолятов K. pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa (KAP) в 2014–2021 гг. Исследование проводили при добровольном информированном согласии пациентов и их законных представителей, протокол исследования одобрен Локальным этическим комитетом НМИЦ здоровья детей (протокол № 5a от 02.06.2022).
Таблица 1. Характеристики антибиотикорезистентных изолятов K. pneumoniae
Table 1. Characteristics of antibiotic-resistant K. pneumoniae isolates
Пациент Patient | Изолят Isolate | Дата выделения Data of isolation | Номер в GenBank Assembly Accession/Accession number | Генотип Genotype | Сублиния Sublineage | Клональная группа (CG) Clonal group (CG) | wzi аллель wzi allele | Соответствующий KL-тип Associated KL type | O-локус O locus | O-тип O type | Показатель вирулентности Virulence score | Показатель резистентности Resistance scores |
P9 | SCCH70:Kpn76815 | 19.06.2017 | JAUPHS000000000 | ST307 | SL307 | CG307 | – | KL102* | O1/O2v2 | O2afg | 1 | 3 |
P11 | SCCH72:Kpn881723 | 20.09.2018 | JAUPHU000000000 | ST2975 | SL307 | CG307 | 173 | KL102 | OL102 | OL102 | 1 | 3 |
P8 | SCCH69:Kpn69138 | 13.04.2016 | JAUOCC000000000 | ST395 | SL395 | CG395 | 2 | KL2 | O1/O2v2 | O2afg | 4 | 3 |
P18 | SCCH87:Kpn2182174 | 05.11.2021 | CP122350-CP122355 | ST395 | SL395 | CG395 | 2 | KL2 | O1/O2v1 | O1 | 1 | 3 |
P17 | SCCH84:Kpn2082401 | 12.09.2020 | JAUTIO000000000 | ST866 | SL1198 | CG1198 | 478 | KL46 | O3b | O3b | 1 | 3 |
P17 | SCCH86:Kpn207262 | 02.10.2020 | JAUTIQ000000000 | ST866 | SL1198 | CG1198 | 478 | KL46 | O3b | O3b | 1 | 3 |
P10 | SCCH71:Kpn863165 | 17.12.2018 | JAUPHT000000000 | ST985 | SL29 | CG985 | 39 | KL39 | O1/O2v2 | O1 | 1 | 3 |
P12 | SCCH73:Kpn96857 | 11.03.2019 | JAUPHV000000000 | ST29 | SL29 | CG29 | 19 | KL19 | O1/O2v2 | O1 | 1 | 3 |
Культуры характеризовались фенотипической MDR, широкой (Extremely Drug Resistance, XDR) или пан-резистентностью (Pan Drug Resistance, PDR) в отношении категорий антибиотиков, рекомендованных EUCAST для Enterobacteriaceae [11, 12], и в соответствии с критериями EUCAST [11].
Методы
Культивирование K. pneumoniae выполняли на кровяном и Uri-select агаре («BioRad») при 37ºС в течение 24–48 ч. Для идентификации использовали масс-спектрометр «MALDI-TOF» («Bruker Daltonics»).
Минимальные ингибирующие концентрации (minimum inhibitory concentration, MIC) определяли методом микроразведений в отношении антимикробных препаратов следующих категорий:
- аминогликозиды: гентамицин, тобрамицин, амикацин, нетилмицин;
- карбапенемы: имипенем, меропенем;
- цефалоспорины расширенного спектра (3 и 4 поколений): цефалоспорин, цефепим;
- фторхинолоны: ципрофлоксацин, левофлоксацин;
- ингибиторы метаболизма фолатов: триметоприм/сульфаметоксазол;
- монобактамы: азтреонам;
- фосфоновые кислоты: фосфомицин;
- полимиксины: колистин (полимиксин Е), полимиксин В;
- пенициллины с ингибиторами β-лактамаз: тикарциллин/клавуланат, пиперациллин/тазобактам.
ДНК из изолятов выделяли по методике [13], дополненной очисткой от полисахаридов с помощью CTAB (cetyltrimethylammonium bromide).
Библиотеки ДНК готовили по протоколу «Nextera DNA Flex Library Prep» («Illumina») и секвенировали на приборе «NextSeq 500/550» («Illumina»), используя картридж «Mid Output 300 cycles».
Для сборки геномов применяли «CLC Genomic Workbench v.21.0.1» («Qiagen») и «SPAdes v. 3.13.0»2. Для визуализации результатов сборки репликонов и сравнения геномов использовали «CGView Server»3 [14]. Аннотацию выполняли с помощью Rapid Annotations Subsystems Technology [15] и NCBI Prokaryotic Genome Annotation Pipeline [16]. Для поиска областей профагов использовали «PHASTER»4 [17]. Результаты полногеномного секвенирования депонировали в GenBank в биопроект PRJNA561493 под номерами, представленными в табл. 1.
Анализ геномных данных проводили с помощью ресурсов платформы «Pathogenwatch v. 21.0.0»5, позволяющей взаимодействовать с «Kleborate v. 2.2.0»6 [18] — ресурсом, специально разработанным для исследования K. pneumoniae complex. С помощью «Kleborate v. 2.2.0» определяли генотип в контексте генов MLST (MultiLocus Sequence Typing), факторы вирулентности: иерсиниябактин (ybt), аэробактин (iuc), колибактин (clb), сальмохелин (iro), гипермукоидность (rmpA, rmpA2), детерминанты антимикробной резистентности. С обращением к сайту Kaptive7 [19] определяли аллели гена wzi, типировали K-локусы (капсула) и O-локусы (липополисахарид).
Критерии, разработанные «Kleborate v. 2.2.0», использовали для оценки степени вирулентности и резистентности (virulence score, antimicrobial resistance score).
Для вирулентности:
- степень 5 — ybt, iuc, clb;
- степень 4 — ybt, iuc;
- степень 3 — iuc;
- степень 2 — ybt, clb (или только clb);
- степень 1 — ybt;
- степень 0 — все отсутствуют.
Для резистентности:
- степень 3 — карбапенемазы и устойчивость к колистину;
- степень 2 — карбапенемазы;
- степень 1 — β-лактамазы расширенного спектра без карбапенемаз;
- степень 0 — отсутствие β-лактамаз расширенного спектра [18].
Для анализа расширенного спектра факторов вирулентности использовали базу данных VFDB8 [20]. К веб-платформе BIGSdb-Pasteur9 обращались для уточнения генотипов для ybt, iuc, clb, а также для выявления локусов резистентности к ионам тяжёлых металлов.
Более полный спектр генов резистентности определяли с помощью ресурса CARD10 [21], а также BV-BRC11, образованного на основе PATRIC [22].
PlasmidFinder 2.112 [23] использовали для выявления групп несовместимости (incompatibility, Inc) репликонов плазмид.
Результаты
В ходе мониторинга лекарственной устойчивости внутрибольничных изолятов K. pneumoniae, A. baumannii, P. aeruginosa (KAP) в двух многопрофильных детских стационарах: НМИЦ здоровья детей и НИИ неотложной детской хирургии и травматологии, в 2014–2021 гг. выделили 685 культур из кровотока и ликвора. K. pneumoniae составила 9,2% (63 изолята). Восемь изолятов, представленных в табл. 1, отличались устойчивостью ко множественным категориям антибиотиков. Изоляты пациентов P8–P12, P18 были PDR, по данным MIC, для всех 9 категорий антибиотиков. От пациента P17 исследовали 2 изолята, выделенных с разницей в 3 нед. Оба изолята (табл. 2) были чувствительны к фторхинолонам (к левофлоксацину при увеличенной экспозиции). Первый по времени выделения был, кроме того, чувствителен к ингибиторам метаболизма фолатов, а также показывал MIC в отношении полимиксина B, соответствующую пограничному значению резистентности. Таким образом, этот изолят можно признать MDR, а второй — XDR. Из 8 изолятов 7 выделили из кровотока, а один (P8) — из спинномозговой жидкости. Отметим, что пациенты, за исключением P12 (8 лет), были в возрасте до 1 года, преимущественно имели генетически обусловленные заболевания, приводящие к порокам развития, требующим хирургического вмешательства. P8 имел тяжёлую сочетанную травму, P12 — системный васкулит, сопровождавшийся гемолитико-уремическим синдромом. В 2 случаях (P10 и P12) зафиксирован летальный исход. Для выявления механизмов резистентности и анализа факторов вирулентности 8 изолятов исследовали с помощью полногеномного секвенирования.
Таблица 2. Сравнение антибиотикограмм изолятов K. pneumoniae ST866 от пациента Р17 по трем категориям антибиотиков
Table 2. Comparison of antibiograms of K. pneumoniae ST866 isolates from patient P17 in three categories of antibiotics
Изолят Isolate | МИК, мг/л | MIC, mg/L | |||
фторхинолоны fluoroquinolones | ингибиторы метаболизма фолатов folate pathway inhibitors | полимиксины polymyxins | ||
ципрофлоксацин ciprofloxacin | левофлоксацин levofloxacin | триметоприм/сульфаметоксазол trimethoprim/sulfamethoxazole | полимиксин В Polymixin B | |
SCCH84:Kpn2082401 | ≤ 0,25 | 1* | 2 | 2 |
SCCH86:Kpn207262 | 0,5* | 1* | 16/304 | 16 |
Референсные значения МИК, мг/л Resistance — MIC breakpoints, mg/L | 0,5 | 1 | 4 | 2 |
Примечание. *Чувствительность при увеличенной экспозиции; серым цветом выделены ячейки, соответствующие чувствительности изолята к антимикробному препарату.
Note. *The sensitivity at increased exposure; the cells corresponding to the sensitivity of the isolate to the antimicrobial drug are highlighted in grеy.
Основные характеристики резистентных изолятов. Все 8 изолятов относились к филогруппе Kp1 и подразделялись на 4 сублинии (SL) (табл. 1), из которых 2 (307 и 395) относятся к наиболее представленным у нозокомиальных K. pneumoniae в России [1]. Три сублинии включали по одной CG. SL29 была представлена двумя CG. Несмотря на то что ST985 отличается по одному локусу (gapA) аллельного профиля от ST29 (single locus variant, SLV), он относится к другой клональной группе — CG985. Отметим, что в CG307 ST2975 также является SLV ST307 по локусу rpoB.
Изоляты SL29 имели одинаковый вариант О-антигена — O1/O2v2. Такой же вариант характерен для изолятов ST307 и ST395 P8. Другой изолят ST395 (P18) имел О-антиген O1/O2v1. Разные варианты О-антигена определяются мономером, составляющим полисахарид. В варианте 1 — d-galactan I, в варианте 2 — d-galactan III [24]. Таким образом, даже геномы изолятов одного генотипа могут различаться структурой оперона, отвечающего за О-антиген. У изолятов P17 свой вариант — O3b, но так же, как и варианты О1/О2, превалирующий у изолятов, вызывающих заболевания человека [24]. У изолята ST2975 О-антиген новый, потому назван OL102 по номеру капсульного антигена.
Капсульные полисахариды, напротив, различают изоляты SL29, но одинаковы у изолятов ST395. У изолятов SL307 новый KL-тип — KL102. Кроме того, если у изолята ST2975 капсульный оперон представлен полностью, то в геноме изолята ST307 из генов 5’-конца оперона (galF, cpsACP, wzi, wza, wzb, wzc) есть только полноценный cpsACP. Ген galF является псевдогеном, а ещё 4 гена, а также следующий за ними wbaP, отсутствуют. Ген wbaP кодирует гликозилтрансферазу, которая инициирует синтез капсульного полисахарида, перенося галактозу-1-фосфат на акцептор удепренил-фосфат. В отсутствие этого фермента синтез капсулы невозможен [25]. Гены wzi, wza отвечают за белки-каналы наружной мембраны, а wzc, wzb — за регуляторы полимеризации и транспорта полисахаридов капсулы на поверхность бактериальной клетки.
Опероны фимбрий fimA–fimK (1-го типа) и mrkA–mrkJ (3-го типа), а также ген pilW пилей IV типа присутствовали в геномах всех изолятов.
Определяя резистентность и вирулентность выбранных изолятов на основе геномных данных, мы воспользовались количественными оценками, которые предлагает «Kleborate v. 2.2.0» [19] (табл. 1). Геномы всех изолятов содержали оперон ybt, который находится в ICEKp: ICEKp4 (ybt10) у изолятов CG307, ICEKp5 (ybt14) у изолятов ST395 P8 и ST29, ICEKp12 (ybt16) у изолятов ST395 P18 и ST985, ICEKp15 (ybt18) у изолятов ST866.
Большинство изолятов имело показатель вирулентности 1. У изолята ST395 P8 показатель 4 — самый высокий, поскольку помимо ybt, геном изолята кодирует аэробактин (AbST95). Из дополнительных генов вирулентности peg-344 [26], отвечающий за пермеазу транспортера лекарств и метаболитов, присутствует только в геноме ST395 P8. Гены гипермукоидности rmpA, rmpA2 в этой группе изолятов не выявлены.
Оценка резистентности у всех изолятов самая высокая (3), поскольку в геномах присутствуют как гены карбапенемаз, так и мутации в генах (PhoP_26Q), обеспечивающие устойчивость к колистину (полимиксину Е) [27].
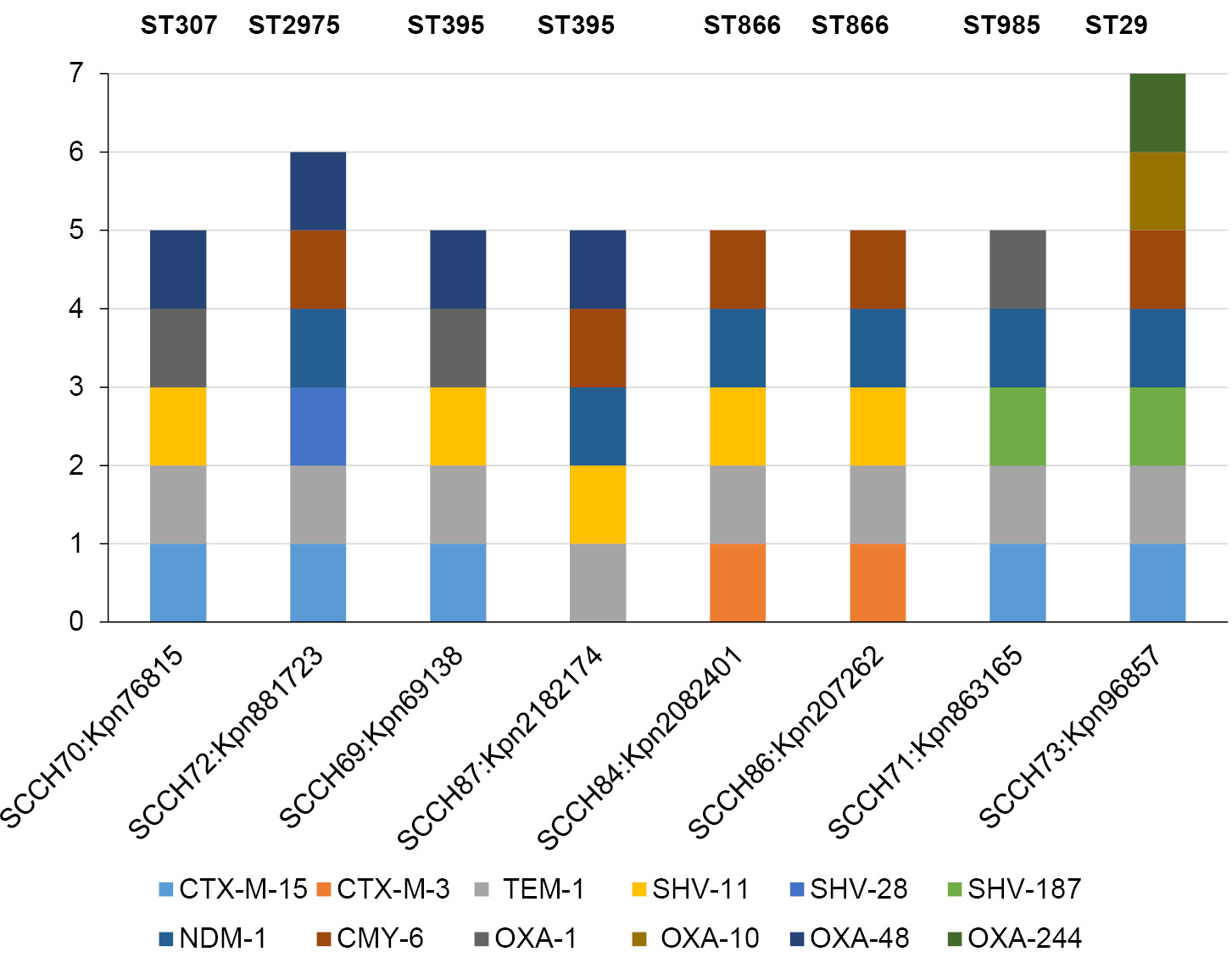
Спектр генов β-лактамаз в геномах изолятов обширен (рис. 1). Во всех геномах присутствует хотя бы одна карбапенемаза: OXA-48 (класс D) или NDM-1 (класс B, металло-β-лактамаза), а в геномах ST2975 и ST395 P18 есть обе β-лактамазы. NDM-1 также относят к β-лактамазам расширенного спектра [28], к которым принадлежат и CTX-M-15 и CTX-M-3 (класс A). Только у изолята ST395 P18 не было CTX-M-лактамазы. Из других β-лактамаз класса A лактамазы TEM и SHV есть во всех геномах. Цефалоспориназа CMY-6 (класс C) присутствует в большинстве геномов, её нет только в геномах изолятов ST307 и ST395 P8. Однако в этих геномах есть дополнительная β-лактамаза класса D — OXA-1, которая присутствует также в геноме ST985. Геном изолята ST29 отличает наличие двух дополнительных β-лактамаз класса D — OXA-10 и OXA-244. Таким образом, в геномах изолятов есть, по крайней мере, 5 генов β-лактамаз разных классов, а в геноме ST29 их 7.
Рис. 1. Разнообразие генов β-лактамаз в геномах исследованных изолятов.
Fig. 1. Diversity of beta-lactamase genes in the genomes of the studied isolates.
Гены резистентности иных категорий антибиотиков представлены на рис. 2. У изолятов ST866 выявили минимальное количество дополнительных генов резистентности — 6. Среди них отсутствуют гены устойчивости к фторхинолонам и триметоприму, что подтверждает фенотипические данные. Максимальное количество дополнительных генов у изолята ST395 P8 — 16. У него и у изолята ST985 (14 генов) для каждой категории антибиотиков есть хотя бы один ген, определяющий устойчивость. Отметим, что только у изолятов-лидеров есть гены устойчивости к макролидам.
Рис. 2. Разнообразие генов резистентности к иным категориям антибиотиков в геномах исследованных изолятов.
Fig. 2. Diversity of resistance genes to other categories of antibiotics in the genomes of the studied isolates.
Гены устойчивости к ионам тяжёлых металлов обнаружены в 6 из 8 геномов (рис. 3). Это опероны устойчивости к мышьяку (arsABCDR), меди (pcoABCDERS), серебру (silABCEFGPRS) и теллуру (terABCDEWXYZ). Все 4 оперона присутствуют в геноме ST307, по 3 — в геномах ST2975 и ST985, по 2 — в ST866, а в геноме ST395 P8 — только оперон устойчивости к теллуру.
Рис. 3. Гены устойчивости к ионам металлов в геномах исследованных изолятов. Красной точкой отмечен неполный оперон arsBCRD.
Fig. 3. Genes of resistance to heavy metal ions in the genomes of the studied isolates. The red dot marks the incomplete arsBCRD operon.
Плазмиды резистентных изолятов. Как правило, опероны тяжёлых металлов, многие гены резистентности присутствуют в плазмидах [29]. Мы оценили количество групп Inc-плазмид для геномных данных каждого изолята (рис. 4). Максимальное количество Inc обнаружили у изолята ST307 (7), минимальное — у изолятов ST985 (1) и ST866 (3). Данные подтверждают наличие плазмид в геномах всех изолятов.
Рис. 4. Разнообразие ориджинов репликации (Inc) плазмид в исследованных изолятах.
Fig. 4. Diversity of origins of replication (Inc) plasmids in the studied isolates.
Обращает на себя внимание различный спектр Inc у изолятов одного генотипа ST395. Сборка плазмидных репликонов изолятов ST395 P8 и ST395 P18 показала, что в геноме ST395 P18 есть 5 плазмид, 3 из которых способны к конъюгативному переносу, а в геноме ST395 P8 присутствуют 4 плазмиды, одна из которых криптическая (к конъюгативному переносу способны 2). Плазмидомы изолятов различались по количеству и набору генов резистентности и вирулентности (рис. 5). Плазмидом ST395 P8 отличало наличие генов β-лактамаз расширенного спектра, аэробактина, peg-344, оперона устойчивости к теллуру и большего количества генов β-лактамаз. В плазмидоме ST395 P18 было больше генов карбапенемаз, генов устойчивости к сульфонамидам и аминогликозидам. Таким образом, плазмидом изолятов ST395 претерпел существенные изменения с 2016 по 2021 г.
Рис. 5. Гены резистентности и вирулентности в плазмидах K. pneumoniae ST395. Обведены факторы, присутствующие только в плазмидах изолята ST395 P8.
Fig. 5. Resistance and virulence genes in K. pneumoniae ST395 plasmids. Circled are factors that are present only in the plasmids of the ST395 P8 isolate.
Сравнение профагов изолятов ST395 показало различия и по этим мобильным генетическим элементам. В геноме ST395 P8 идентифицировали 8 профагов, в геноме ST395 P18 — 7; 5 профагов были общими для геномов изолятов ST395. Однако плазмиды ST395 P8 содержали в 2 раза больше профагов (10), чем плазмиды ST395 P18 (5). Все профаги имели гомологию с 17 различными бактериальными фагами класса Caudoviricetes. Профаги хромосом содержали по 3 гена холинов, формирующих поры в клеточной мембране [30], и гены лизинов, повреждающих пептидогликан: 4 гена лизоцима — ST395 P8 и 5 генов лизоцима и ген мембранно-связанной литической трансгликозилазы D — ST395 P18. При этом CRISPR-Cas (Clustered Regularly Interspaced Short Palindromic Repeats) системы в обоих геномах отсутствовали. В то же время анализ профагов плазмид изолятов ST395 подтвердил наблюдение, что фаги могут быть переносчиками генов антибиотикорезистентности [31]. В профагах плазмид изолята ST395 P8 находились гены β-лактамаз (5), хлорамфеникол ацетилтрансфераз (3), аминогликозид ацетилтрансфераз (2), дигидрофолат редуктазы, стрептомицин аденилтрансферазы и оперон устойчивости к теллуру. Не уступали им и профаги плазмид изолята ST395 P18, которые содержали гены аминогликозид ацетилтрансфераз (3), дигидроптероат синтаз (2), β-лактамаз TEM и OXA-48, хлорамфеникол ацетилтрансферазы и 3 эффлюксных транспортеров SMR (Small multidrug resistance)
Обсуждение
Проведённое исследование выявило большое разнообразие генотипов MDR K. pneumoniae, выделенных из ликвора и крови. Из них, по данным Института Пастера13, лидирующим в мире является ST307 (1786 изолятов, 21 в России), за ним следуют ST395 (380, 188 в России) и ST29 (255, 2 в России), ST985 (48), ST2975 (29, 12 в России), ST866 (7). Инфицирование PDR K. pneumoniae ST29 и ST985 привело к смертельным исходам. Инфекция, являясь системной, помимо кровотока, была выявлена и в других локусах как пациентов P10, P12, так и пациентов P9, P11, P17, P18. У пациента P8 инфекция обнаружена только в ликворе и была вызвана наиболее вирулентным изолятом ST395 (степень 4). По показателям резистентности (PDR) и вирулентности изолят ST395 P8 соответствует характеристикам конвергентного патотипа ST395 [5]. Исследование плазмид изолята подтверждает перенос факторов резистентности (в том числе к ионам тяжёлых металлов) и вирулентности именно этими мобильными генетическими элементами и профагами в составе плазмид. Сопоставление геномов изолятов ST395 P8 и ST395 P18 выявляет свидетельства рекомбинаций в «горячих точках», охарактеризованных E.R. Shaidullina и соавт. [5]. У изолятов различаются области О-антигена, ICEKp и профагов. Изолят ST395 P18 имеет ICEKp12 (ybt16), превалирующий в CG305 и в субкладе B1 (KL2KL30) [5], при этом характеристика K-локуса изолята соответствует характеристике субклады. У изолята ST395 P8 ICEKp5 (ybt14) — редкий [5] и относящийся к субкладе A1 (KL108). Однако у изолята ST395 P8 K-локус — KL2, что может быть ещё одним свидетельством рекомбинационных событий.
У изолятов CG307 отличия в наборе генов резистентности связаны с разным плазмидным профилем. В хромосоме изоляты имеют одинаковые области ICEKp4 (ybt10), но существенные отличия в главной «горячей точке» рекомбинации — K- и O-локусах.
В SL29 изоляты сходны только O-локусом, имеют разные K-локусы, ICEKp, плазмидный профиль и спектр генов резистентности. Отметим, что единственная плазмида изолята ST985 (IncFIB(K)) содержит 3 оперона устойчивости к ионам тяжёлых металлов и большой перечень генов, определяющих вместе с генами хромосомы PDR изолята.
Изоляты ST866 — наименее резистентные, тем не менее являются MDR и XDR, несут плазмиды, содержащие в том числе 2 оперона устойчивости к ионам тяжёлых металлов.
Возвращаясь к актуальной проблеме резистентности K. pneumoniae к карбапенемам, необходимо отметить наличие в выборке 2 изолятов, содержащих по 2 гена карбапенемаз: OXA-48 и NDM-1. Изоляты относятся к международным клонам особого риска CG395 и CG307.
Таким образом, в выборке резистентных изолятов K. pneumoniae, выделенных из кровотока и ликвора, обнаружили 6 изолятов PDR, один из которых относится к конвергентному патотипу ST395.
1 База данных AMRmap. URL: https://AMRmap.ru
2 St. Petersburg genome assembler, Russia.
URL: http://cab.spbu.ru/software/spades
3 CGView Server.
URL: http://stothard.afns.ualberta.ca/cgview_server
4 PHAge Search Tool Enhanced Release. URL: https://phaster.ca
5 Pathogenwatch v. 21.0.0. URL: https://pathogen.watch
6 Kleborate v. 2.2.0.
URL: https://github.com/klebgenomics/Kleborate/wiki
7 Kaptive. URL: https://kaptive-web.erc.monash.edu/
8 Virulence factor database. URL: http://www.mgc.ac.cn/VFs
9 Institut Pasteur Klebsiella pneumoniae species complex.
URL: https://bigsdb.pasteur.fr/klebsiella
10 Comprehensive Antibiotic Resistance Database.
11 Bacterial and Viral Bioinformatics Resource Center.
12 PlasmidFinder 2.1.
URL: https://cge.food.dtu.dk/services/PlasmidFinder
13 Institut Pasteur. URL: bigsdb.pasteur.fr/klebsiella
Об авторах
Ольга Львовна Воронина
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: olv550@gmail.com
ORCID iD: 0000-0001-7206-3594
к.б.н., доцент, зав. лаб. анализа геномов
Россия, МоскваМарина Сергеевна Кунда
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Автор, ответственный за переписку.
Email: markunda99@gmail.com
ORCID iD: 0000-0003-1945-0397
к.б.н., с.н.с. лаб. анализа геномов
Россия, МоскваНаталья Николаевна Рыжова
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: rynatalia@yandex.ru
ORCID iD: 0000-0001-5361-870X
к.б.н., с.н.с. лаб. анализа геномов
Россия, МоскваЕкатерина Ивановна Аксенова
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: aksenova16@yandex.ru
ORCID iD: 0000-0003-2704-6730
к.б.н., с.н.с. лаб. анализа геномов НИЦЭМ им. Н.Ф. Гамалеи
Россия, МоскваЗульфиря Закиевна Садеева
Национальный медицинский исследовательский центр здоровья детей
Email: zulfiryasadeeva@yandex.ru
ORCID iD: 0000-0002-4587-0902
м.н.с. лаб. молекулярной микробиологии
Россия, МоскваИрина Евгеньевна Новикова
Национальный медицинский исследовательский центр здоровья детей
Email: novikovayudina@outlook.com
ORCID iD: 0000-0003-4234-0209
м.н.с. лаб. молекулярной микробиологии
МоскваАнна Валерьевна Лазарева
Национальный медицинский исследовательский центр здоровья детей
Email: lasarevaav@nczd.ru
ORCID iD: 0000-0003-3896-2590
д.м.н., г.н.с. лаб. молекулярной микробиологии, зав. лаб. микробиологии
Россия, МоскваОльга Витальена Карасева
Национальный медицинский исследовательский центр здоровья детей; Научно-исследовательский институт неотложной детской хирургии и травматологии
Email: karaseva.o@list.ru
ORCID iD: 0000-0001-9418-4418
д.м.н., руководитель отделения неотложной хирургии и травм детского возраста НМИЦ здоровья детей, Москва, Россия; зам. директора по научной работе, руководитель отдела сочетанной травмы, анестезиологии-реанимации НИИ неотложной детской хирургии и травматологии
Россия, Москва; МоскваАндрей Петрович Фисенко
Национальный медицинский исследовательский центр здоровья детей
Email: olv550@gmail.com
ORCID iD: 0000-0001-8586-7946
д.м.н., профессор, директор
Россия, МоскваАлександр Леонидович Гинцбург
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: gintsburg@gamaleya.org
ORCID iD: 0000-0003-1769-5059
д.б.н., профессор, академик РАН, директор
Россия, МоскваСписок литературы
- Белобородов В.Б., Голощапов О.В., Гусаров В.Г. и др. Методические рекомендации Российской некоммерческой общественной организации «Ассоциация анестезиологов-реаниматологов», Межрегиональной общественной организации «Альянс клинических химиотерапевтов и микробиологов», Межрегиональной ассоциации по клинической микробиологии и антимикробной химиотерапии (МАКМАХ), общественной организации «Российский Сепсис Форум» «Диагностика и антимикробная терапия инфекций, вызванных полирезистентными штаммами микроорганизмов» (обновление 2022 г.). Вестник анестезиологии и реаниматологии. 2022;19(2):84–114. Beloborodov V.B., Goloschapov O.V., Gusarov V.G., et al. Guidelines of the Association of anesthesiologists-intensivists, the Interregional non-governmental organization Alliance of clinical chemotherapists and microbiologists, the Interregional Association for Clinical Microbiology and Antimicrobial Chemotherapy (IACMAC), and NGO Russian sepsis forum diagnostics and antimicrobial therapy of the infections caused by multiresistant microorganisms (update 2022). Messenger of Anesthesiology and Resuscitation. 2022;19(2):84–114. DOI: https://doi.org/10.21292/2078-5658-2022-19-2-84-114 EDN: https://elibrary.ru/vjuogq
- Khrulnova S., Fedorova A., Frolova I., et al. Distribution of virulence genes and capsule types in Klebsiella pneumoniae among bloodstream isolates from patients with hematological malignancies. Diagn. Microbiol. Infect. Dis. 2022;104(1):115744. DOI: https://doi.org/10.1016/j.diagmicrobio.2022.115744
- Fursova A.D., Fursov M.V., Astashkin E.I., et al. Early response of antimicrobial resistance and virulence genes expression in classical, hypervirulent, and hybrid hvKp-MDR Klebsiella pneumoniae on antimicrobial stress. Antibiotics (Basel). 2021;11(1):7. DOI: https://doi.org/10.3390/antibiotics11010007
- Fursova N.K., Astashkin E.I., Ershova O.N., et al. Multidrug-resistant Klebsiella pneumoniae causing severe infections in the Neuro-ICU. Antibiotics (Basel). 2021;10(8):979. DOI: https://doi.org/10.3390/antibiotics10080979
- Shaidullina E.R., Schwabe M., Rohde T., et al. Genomic analysis of the international high-risk clonal lineage Klebsiella pneumoniae sequence type 395. Genome Med. 2023;15(1):9. DOI: https://doi.org/10.1186/s13073-023-01159-6
- Gu D., Dong N., Zheng Z., et al. A fatal outbreak of ST11 carbapenem-resistant hypervirulent Klebsiella pneumoniae in a Chinese hospital: a molecular epidemiological study. Lancet Infect. Dis. 2018;18(1):37–46. DOI: https://doi.org/10.1016/S1473-3099(17)30489-9
- Starkova P., Lazareva I., Avdeeva A., et al. Emergence of hybrid resistance and virulence plasmids harboring New Delhi metallo-β-lactamase in Klebsiella pneumoniae in Russia. Antibiotics (Basel). 2021;10(6):691. DOI: https://doi.org/10.3390/antibiotics10060691
- Khedkar S., Smyshlyaev G., Letunic I., et al. Landscape of mobile genetic elements and their antibiotic resistance cargo in prokaryotic genomes. Nucleic Acids Res. 2022;50(6):3155–68. DOI: https://doi.org/10.1093/nar/gkac163
- Johnson C.M., Grossman A.D. Integrative and Conjugative Elements (ICEs): What they do and how they work. Annu. Rev. Genet. 2015;49:577–601. DOI: https://doi.org/10.1146/annurev-genet-112414-055018
- Shapaka J.T., Muloiwa R., Buys H. Association of full blood count findings with risk of mortality in children with Klebsiella pneumoniae bloodstream infection at a South African children's hospital. BMC Pediatr. 2023;23(1):302. DOI: https://doi.org/10.1186/s12887-023-04104-z
- The European Committee on Antimicrobial Susceptibility Testing. Breakpoint tables for interpretation of MICs and zone diameters. Version 13.1; 2023. URL: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_13.1_Breakpoint_Tables.pdf
- Magiorakos A.P., Srinivasan A., Carey R.B., et al. Multidrug-resistant, extensively drug-resistant and pandrug-resistant bacteria: an international expert proposal for interim standard definitions for acquired resistance. Clin. Microbiol. Infect. 2012;18(3):268–81. DOI: https://doi.org/10.1111/j.1469-0691.2011.03570.x
- Wilson K. Preparation of genomic DNA from bacteria. Curr. Protoc. Mol. Biol. 2001;Chapter 2:Unit2.4. DOI: https://doi.org/10.1002/0471142727.mb0204s56
- Grant J.R., Stothard P. The CGView Server: A comparative genomics tool for circular genomes. Nucleic Acids Res. 2008;36(Web Server issue):W181–4. DOI: https://doi.org/10.1093/nar/gkn179
- Brettin T., Davis J.J., Disz T., et al. RASTtk: a modular and extensible implementation of the RAST algorithm for building custom annotation pipelines and annotating batches of genomes. Sci. Rep. 2015;5:8365. DOI: https://doi.org/10.1038/srep08365
- Li W., O'Neill K.R., Haft D.H., et al. RefSeq: expanding the Prokaryotic Genome Annotation Pipeline reach with protein family model curation. Nucleic Acids Res. 2021;49(D1):D1020–8. DOI: https://doi.org/10.1093/nar/gkaa1105
- Arndt D., Grant J.R., Marcu A., et al. PHASTER: а better, faster version of the PHAST phage search tool. Nucleic Acids Res. 2016;44(W1):W16–21. DOI: https://doi.org/10.1093/nar/gkw387
- Lam M.M.C., Wick R.R., Watts S.C., et al. A genomic surveillance framework and genotyping tool for Klebsiella pneumoniae and its related species complex. Nat. Commun. 2021;12(1):4188. DOI: https://doi.org/10.1038/s41467-021-24448-3
- Lam M.M.C., Wick R.R., Judd L.M., et al. Kaptive 2.0: updated capsule and lipopolysaccharide locus typing for the Klebsiella pneumoniae species complex. Microb. Genom. 2022;8(3):000800. DOI: https://doi.org/10.1099/mgen.0.000800
- Chen L., Yang J., Yu J., et al. VFDB: a reference database for bacterial virulence factors. Nucleic Acids Res. 2005;33(Issue suppl. 1):D325–8. DOI: https://doi.org/10.1093/nar/gki008
- Alcock B.P., Huynh W., Chalil R., et al. CARD 2023: expanded curation, support for machine learning, and resistome prediction at the Comprehensive Antibiotic Resistance Database. Nucleic Acids Res. 2023;51(D1):D690–9. DOI: https://doi.org/10.1093/nar/gkac920
- Olson R.D., Assaf R., Brettin T., et al. Introducing the Bacterial and Viral Bioinformatics Resource Center (BV-BRC): a resource combining PATRIC, IRD and ViPR. Nucleic Acids Res. 2023;51(D1):D678–89. DOI: https://doi.org/10.1093/nar/gkac1003
- Carattoli A., Zankari E., García-Fernández A., et al. In silico detection and typing of plasmids using PlasmidFinder and plasmid multilocus sequence typing. Antimicrob. Agents Chemother. 2014;58(7):3895–903. DOI: https://doi.org/10.1128/AAC.02412-14
- Follador R., Heinz E., Wyres K.L., et al. The diversity of Klebsiella pneumoniae surface polysaccharides. Microb. Genom. 2016;2(8):e000073. DOI: https://doi.org/10.1099/mgen.0.000073
- Pan Y.J., Lin T.L., Chen C.T., et al. Genetic analysis of capsular polysaccharide synthesis gene clusters in 79 capsular types of Klebsiella spp. Sci. Rep. 2015;5:15573. DOI: https://doi.org/10.1038/srep15573
- Liu C., Du P., Xiao N., et al. Hypervirulent Klebsiella pneumoniae is emerging as an increasingly prevalent K. pneumoniae pathotype responsible for nosocomial and healthcare-associated infections in Beijing, China. Virulence. 2020;11(1):1215–24. DOI: https://doi.org/10.1080/21505594.2020.1809322
- Olaitan A.O., Morand S., Rolain J.M. Mechanisms of polymyxin resistance: acquired and intrinsic resistance in bacteria. Front. Microbiol. 2014;5:643. DOI: https://doi.org/10.3389/fmicb.2014.00643
- Palzkill T. Metallo-β-lactamase structure and function. Ann. N.Y. Acad. Sci. 2013;1277:91–104. DOI: https://doi.org/10.1111/j.1749-6632.2012.06796.x
- Wu F., Ying Y., Yin M., et al. Molecular characterization of a multidrug-resistant Klebsiella pneumoniae strain R46 isolated from a rabbit. Int. J. Genomics. 2019;2019:5459190. DOI: https://doi.org/10.1155/2019/5459190
- Fernandes S., São-José C. More than a hole: the holin lethal function may be required to fully sensitize bacteria to the lytic action of canonical endolysins. Mol. Microbiol. 2016;102(1):92–106. DOI: https://doi.org/10.1111/mmi.13448
- Balcazar J.L. Bacteriophages as vehicles for antibiotic resistance genes in the environment. PLoS Pathog. 2014;10(7):e1004219. DOI: https://doi.org/10.1371/journal.ppat.1004219
Дополнительные файлы