Устойчивость к противомикробным препаратам пищевых изолятов Salmonella enterica на территории Республики Беларусь
- Авторы: Куликова Н.Г.1, Чернышков А.В.1, Михайлова Ю.В.1, Зенькович А.Л.2, Довнар Д.А.2, Марейко А.М.2, Битюмина Л.А.1, Шеленков А.А.1, Егорова А.Е.1, Саенко С.С.1, Манзенюк И.Н.1
-
Учреждения:
- Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
- Республиканский центр гигиены, эпидемиологии и общественного здоровья
- Выпуск: Том 100, № 2 (2023)
- Страницы: 153-167
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/1301
- DOI: https://doi.org/10.36233/0372-9311-343
- EDN: https://elibrary.ru/mqqmxo
- ID: 1301
Цитировать
Аннотация
Введение. Устойчивость к противомикробным препаратам является глобальной проблемой здравоохранения. Salmonella spp., которые могут передаваться человеку через контаминированную пищевую продукцию, признаны важными патогенами пищевого происхождения во всём мире.
Материалы и методы. Исследования противомикробной резистентности 358 изолятов микроорганизмов из пищевых продуктов и воды, изолированных на территории Республики Беларусь в 2018–2021 гг., проводились путём изучения фенотипических и генотипических характеристик антибиотикорезистентности микроорганизмов. Таксономическое положение бактерий было идентифицировано методом MALDI-TOF масс-спектрометрии. Фенотипическую чувствительность бактерий к антимикробным препаратам определяли методом минимальной подавляющей концентрации с помощью автоматизированного бактериологического анализатора «Sensititre» и диско-диффузионным методом к 45 противомикробным препаратам. Гены устойчивости к противомикробным препаратам у мультирезистентных изолятов сальмонелл определяли с помощью полногеномного секвенирования.
Результаты. Анализ фенотипической чувствительности бактерий in vitro показал высокую чувствительность к фторхинолонам (97,2%), цефалоспоринам 3-го поколения (93,9%), карбапенемам (98,0%), ампициллину (81,8%), аминогликозидам (97,5%), тетрациклинам (87,5%), хлорамфениколу (93,8%), триметоприм/сульфаметоксазолу (ко-тримоксазолу) (95,3%) и колистину (85,2%). Показано, что механизм резистентности к антибиотикам у S. enterica был ассоциирован с наличием генов blaTEM-1B (82%), blaTEM-1C (7,7%), blaSHV-12 (2,6%), blaDHA-1 (2,6%), blaCMY-2 (7,7%), qnrB2 (9,1%), qnrB4 (9,1%), qnrB5 (9,1%), qnrB19 (72,7%), aac(6’)-Ib-cr (9,1%), aac(6’)-Iaa (100%), aadA1 (13,2%), aadA2 (8,8%), tetB (74,3%), tetA (25,7%), tetM (2,9%), tetD (28,6%), mcr-9 (1,5%).
Заключение. Все изоляты микроорганизмов были фенотипически высокочувствительны к препаратам 1-й линии в терапии сальмонеллёза: фторхинолонам и цефалоспоринам 3-го поколения. Результаты полногеномного секвенирования мультирезистентных изолятов сальмонелл (19,0%) выявили гены устойчивости к 9 группам антибиотиков: аминогликозидам (100%), бета-лактамам (57,4%), фторхинолонам (16,2%), тетрациклинам (51,5%), макролидам (1,5%), фениколам (30,4%), триметоприму (13,0%), сульфаниламидам (47,8%) и колистину (1,4%). Таким образом, для контроля устойчивости к противомикробным препаратам среди сальмонелл пищевого происхождения решающее значение имеет эпидемиологический надзор за их распространением в цепи пищевых продуктов.
Полный текст
Введение
Ведущую позицию по бактериальной инвазии желудочно-кишечного тракта среди всех пищевых патогенов занимают различные серовары Salmonella enterica [1]. Сальмонеллёзная инвазия у людей представляет большую опасность из-за способности формировать длительное бактерионосительство и вызывать осложнения [2]. Вследствие высокой экологической пластичности микроорганизмы рода S. enterica легко находят экологические ниши, адаптируются к различным условиям и могут сохранять жизнеспособность в сухих и замороженных пищевых продуктах [1, 2], а также адаптироваться к условиям массового применения антимикробных препаратов в здравоохранении и сельском хозяйстве, способствуя тем самым распространению резистентности к противомикробным препаратам.
Механизмы лекарственной устойчивости микроорганизмов зависят от различных фермент-опосредованных факторов [3]. Учитывая способность Salmonella spp. выступать в роли вектора передачи генов резистентности другим микроорганизмам, важное значение для контроля распространения антибиотикоустойчивости имеет изучение как фенотипического, так и генотипического профилей устойчивости сальмонелл.
Материалы и методы
Коллекция микроорганизмов
Материалом для исследования служили культуры S. enterica (n = 358), изолированные на территории Республики Беларусь в 2018–2021 гг. Выделение и первичная идентификация бактериальных изолятов проводились в Республиканском центре гигиены, эпидемиологии и общественного здоровья (Минск).
Источниками выделения изолятов микроорганизмов служили птицепродукты (n = 113), мясная (n = 52), рыбная (n = 1), молочная (n = 2), кондитерская (n = 3), кулинарная (n = 158) продукция, сточные воды и смывы с рабочих поверхностей (n = 29). Окончательная видовая идентификация изолятов микроорганизмов и определение их чувствительности к противомикробным препаратам проводились в Референс-центре Роспотребнадзора по мониторингу остаточного количества антибиотиков и антибиотикорезистентности бактерий в продовольственном сырье и пищевых продуктах в ЦНИИ Эпидемиологии Роспотребнадзора (Москва).
Видовая идентификация и хранение изолятов микроорганизмов
Все исследованные изоляты микроорганизмов были идентифицированы до рода методом матрично-активированной лазерной ионизации — времяпролетной масс-спектрометрии (MALDI-TOF MS) с применением системы «Microflex LT» и программного обеспечения «MALDI Biotyper Compass v.4.1.80» («Bruker Daltonics»). В качестве критерия надёжной видовой идентификации на MALDI-TOF MS использовали рекомендуемые значения Score ≥ 2,0. Серотипирование сальмонелл проводили при помощи реакции агглютинации с сальмонеллёзными сыворотками («ПЕТСАЛ») согласно схеме Кауффмана–Уайта. Хранение изолятов бактерий осуществляли при –70ºС в бульоне Мюллера–Хинтона с добавлением 10% глицерина [4].
Определение чувствительности в отношении противомикробных препаратов
Профили чувствительности к противомикробным препаратам пищевых изолятов микроорганизмов, выделенных в 2018–2019 гг., определяли диско-диффузионным методом в отношении следующих антибиотиков: ампициллина, цефотаксима, цефтазидима, меропенема, ципрофлоксацина, левофлоксацина, амикацина, гентамицина, хлорамфеникола и ко-тримоксазола. Клинические категории чувствительности изолятов микроорганизмов в отношении противомикробных препаратов определяли на основании пограничных значений минимальной подавляющей концентрации, установленных EUCAST (версии 8.0, 2018 и 9.0, 2019 соответственно).
Профили чувствительности к противомикробным препаратам пищевых изолятов микроорганизмов, выделенных в 2020–2021 гг., проводили методом микроразведений в бульоне Мюллера–Хинтона с определением минимальной подавляющей концентрации с помощью полуавтоматического бактериологического анализатора «Sensititre» («TREK Diagnostics Systems»). Инокуляцию микроорганизмов проводили c использованием 96-луночных микропланшетов с антибиотиками для грамотрицательных микроорганизмов RUGNF и GN4F. Анализ результатов определения чувствительности изолятов микроорганизмов, выделенных из пищевых продуктов и продовольственного сырья, в отношении противомикробных препаратов проводили с помощью программного обеспечения «SWIN» до категории согласно стандарту интерпретации CLSI (30-е издание, 2020) и/или EUCAST (версии 10.0, 2020 и 11.0, 2021 соответственно). Для контроля качества определения чувствительности использовали культуры Escherichia сoli ATCC25922 и E. coli ATCC35218.
Определение генетических детерминант резистентности
Генетические детерминанты резистентности определяли у мультирезистентных изолятов сальмонелл с помощью полногеномного секвенирования. Экстракцию ДНК выполняли с использованием набора реагентов «РИБО-преп» (ЦНИИ Эпидемиологии). Приготовление образцов ДНК для дальнейшего секвенирования осуществляли с использованием «Illumina Nextera DNA Library Prep Kit» и «Illumina Nextera Index Kit». Секвенирование проводили на приборе «Illumina HiSeq1500» («Illumina») с использованием наборов реагентов «Illumina HiSeq PE Rapid Cluster Kit v2» и наборов «Illumina HiSeq Rapid SBS Kit v2».
Биоинформатический анализ
Сборки геномов на основе коротких прочтений были получены с помощью программы «SPAdes v. 3.12» [5] с параметрами по умолчанию. Оценка качества сборки, проверка организмов и начальная аннотация выполнялись с использованием программного комплекса, описанного ранее [6]. Гены устойчивости к антибиотикам in silico определяли при помощи базы данных «Resfinder 4.0» [7] с параметрами по умолчанию, проведено типирование изолятов микроорганизмов с использованием схемы мультилокусного типирования последовательностей (MLST) с помощью веб-сайта Pasteur MLST1, по состоянию на 20.10.2021).
Статистическая обработка результатов
Статистическую обработку результатов исследования проводили с использованием стандартных методов описательной статистики с помощью программы «Microsoft Office Excel 2010». Статистическую значимость различий доли резистентных культур оценивали с помощью t-критерия Стьюдента при уровне значимости α < 0,05.
Результаты
Всего за 2018–2021 гг. было изучено 358 изолятов S. enterica, выделенных из продуктов питания и продовольственного сырья на территории Республики Беларусь. Наибольшее количество культур для исследований поступило в Референс-центр Роспотребнадзора в 2018 г. (n = 121; 33,8 ± 0,29%), наименьшее — в 2021 г. (n = 43; 12,0 ± 0,14%). В 2019 и 2020 гг. поступило 104 (29,1 ± 0,27%) и 90 (25,1 ± 0,24%) изолятов микроорганизмов соответственно.
Большинство культур было выделено из мясной продукции (n = 52), птицепродуктов (n = 113) и кулинарной продукции, приготовленной из переработанного мяса свинины и домашней птицы (n = 158) (табл. 1). Наименьшее количество сальмонелл в наших исследованиях было выделено из кондитерской, молочной и рыбной продукции. Помимо пищевой продукции сальмонеллы были выделены из питьевой воды, сточных вод и смывов с рабочих поверхностей, которые были отнесены к продукции «другое» (n = 29).
Таблица 1. Обсеменённость пищевой продукции культурами рода Salmonella
Вид продукции Source | Количество изолятов Number of isolates | Доля изолятов, % Number of isolates, % |
Кулинарная продукция Cookery food | 158 | 44,1 ± 0,28 |
Птицепродукты Poultry | 113 | 31,6 ± 0,24 |
Мясная продукция Meat | 52 | 14,5 ± 0,14 |
Кондитерская продукция Confectionery | 3 | 0,8 ± 0,06 |
Молочная продукция Dairy | 2 | 0,6 ± 0,05 |
Рыбная продукция Seafood | 1 | 0,3 ± 0,04 |
Другое Others | 29 | 8,1 ± 0,1 |
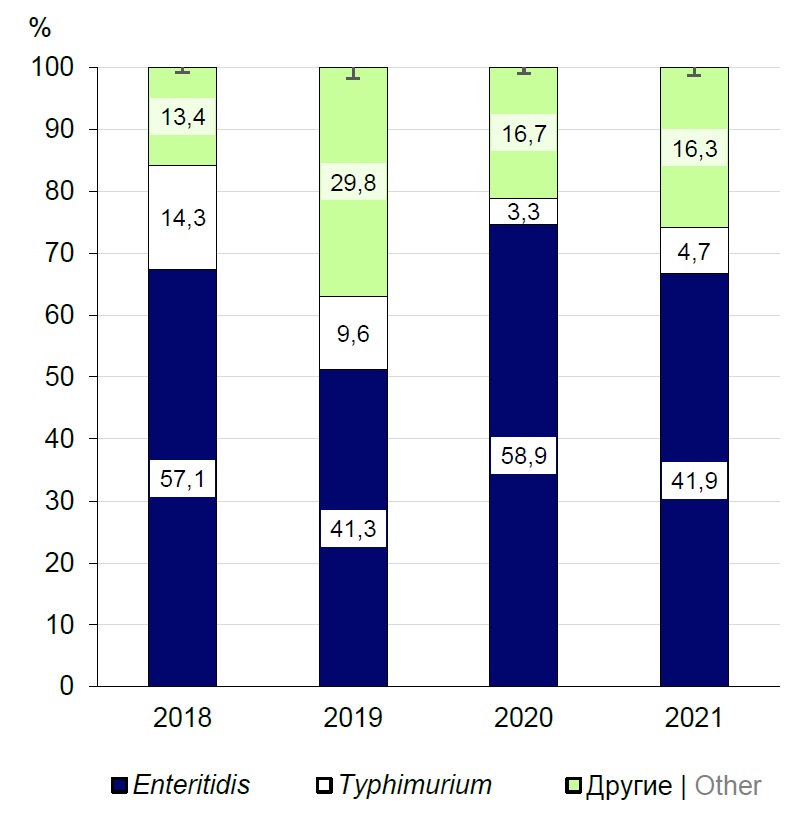
За время исследований было выделено 28 серотипов S. enterica. Доминирующими были изоляты серотипа Enteritidis (n = 182; 50,80 ± 0,20%): в 2018 г. их доля составила 57,10 ± 0,27% (n = 68), в 2019 г. — 41,30 ± 0,22% (n = 43), в 2020 г. — 58,9 ± 0,27% (n = 53), в 2021 г. — 41,90 ± 0,12% (n = 18) (рис. 1). От 3,30 ± 0,12% (n = 3) в 2020 г. до 14,30 ± 0,56% (n = 17) в 2018 г. сальмонелл принадлежало серотипу Typhimurium (n = 61; 17,00 ± 0,15%). Все остальные серотипы составляли наименьшую долю (от 0,30 ± 0,01 до 2,50 ± 0,06%), поэтому были отнесены в группу «другие», которая составляла от 13,40 ± 0,16 до 29,80 ± 0,25% культур. Культуры в данной группе принадлежали серотипам Agona, Blegdam, Brandenburg, Bredeney, Chester, Derby, Dublin, Essen, Fyris, Give, Goettingen, Goma, Infantis, Jerusalem, Kapemba, Kottbus, London, Mbandaka, Munchen, Panama, Saintpaul, Sandiego, Tsevie, Virchow.
Рис. 1. Распространённость серотипов S. enterica пищевого происхождения, выделенных в Республике Беларусь.
Проанализированные данные фенотипической чувствительности изолятов сальмонелл к 45 антибактериальным препаратам за 2018–2021 гг. показали высокую чувствительность бактерий к ним (76,90 ± 0,06%). Множественная устойчивость к антибиотикам (multidrug resistance, MDR) была отмечена у 19,00 ± 0,05% (n = 68) культур.
Основными препаратами терапии тяжёлых форм сальмонеллёза являются антибиотики фторхинолоновой группы, у которых отсутствует перекрёстная резистентность с другими классами антибиотиков из-за их антимикробной активности, обусловленной ингибированием ДНК-гиразы или топоизомеразы IV [8]. При анализе фенотипической чувствительности сальмонелл, выделенных из пищевых продуктов и продовольственного сырья на территории Республики Беларусь, была отмечена довольно высокая чувствительность бактерий к данной группе препаратов (от 88,40 ± 0,31 до 100%). Однако в 2020–2021 гг. имеется тенденция к постепенному ежегодному увеличению доли резистентных изолятов S. enterica: с 0% в 2018 и 2019 гг. до 5,6 ± 0,1% и 11,60 ± 0,31% в 2020 и 2021 гг. соответственно (рис. 2).
Рис. 2. Профиль фенотипической чувствительности изолятов S. enterica к фторхинолонам.
Анализ фенотипической чувствительности сальмонелл показал тенденцию к снижению активности цефалоспоринов 3-го поколения, несмотря на высокий процент фенотипически чувствительных культур: с 100% в 2018 г. до 83,70 ± 0,14% в 2021 г. (рис. 3). Вместе с тем на протяжении всего периода исследований наблюдался ежегодный рост фенотипически резистентных культур сальмонелл.
Рис. 3. Профиль фенотипической чувствительности изолятов S. enterica к цефалоспоринам III поколения.
За весь период исследований S. enterica были фенотипически высокочувствительными к резервным препаратам терапии сальмонеллеза — ампициллину и карбапенемам (имипенему и меропенему). В отношении ампициллина наблюдался общий тренд постепенного нарастания резистентных культур с 14,9 ± 0,1% в 2018 г. до 23,30 ± 0,55% в 2021 г.; в отношении карбапенемов доля устойчивых культур в 2020 г. увеличилась до 5,60 ± 0,11% по сравнению с 2018 и 2019 гг., затем незначительно снизилась (до 4,70 ± 0,14%) в 2021 г.
Антибиотики из группы аминогликозидов имеют основное клиническое значение в терапии нозокомиальных инфекций, вызванных аэробными грамотрицательными бактериями. Изучение фенотипической чувствительности к аминогликозидам культур S. enterica, выделенных из пищевых продуктов на территории Республики Беларусь, показало высокую фенотипическую чувствительность к аминогликозидам на протяжении всего периода мониторинга: с 95,30 ± 0,06 до 100,0%. Однако в 2020–2021 гг. появилась тенденция к росту доли резистентных культур до 3,30 ± 0,07 и 4,70 ± 0,15% соответственно (рис. 4).
Рис. 4. Профиль фенотипической чувствительности изолятов S. enterica к аминогликозидным антибиотикам.
В 2020 и 2021 гг. была проведена оценка чувствительности выделенных культур Salmonella к колистину и тетрациклинам как препаратам резерва в отношении микроорганизмов с множественной устойчивостью. Колистин остаётся единственным препаратом последнего резерва, подходящим для лечения опасных для жизни инфекций, вызываемых карбапенем-устойчивыми энтеробактериями. В ряде стран и регионов уже выявлены колистин-резистентные бактерии, вызывающие инфекции, против которых в настоящее время не существует эффективных антибиотиков [9]. В проведённом нами исследовании была выявлена динамика к увеличению доли фенотипически колистин-резистентных изолятов микроорганизмов в 2,3 раза (с 10,10 ± 0,18% в 2020 г. до 23,30 ± 0,58% в 2021 г.) и тетрациклин-резистентных культур — в 7,2 раза (с 3,9 ± 0,1% в 2020 г. до 27,90± 0,65% в 2021 г.; рис. 5). Кроме того, антибиотиками резерва с широким спектром действия являются ко-тримоксазол и хлорамфеникол, к которым на протяжении всего периода наблюдений сохранялась низкая доля резистентных культур: от 4,8 ± 0,1 до 6,70 ± 0,13% и от 3,80 ± 0,07 до 7,4 ± 0,12% соответственно.
Рис. 5. Динамика изменения доли фенотипически резистентных культур S. enterica к колистину и тетрациклину в 2020–2021 гг.
Тяжесть протекания сальмонеллёзной инфекции зависит от многих факторов, включая наличие детерминант устойчивости к противомикробным препаратам, присутствующих у бактерий [10]. На территории Беларуси в 2018–2021 гг. было выявлено 68 (19,0 ± 0,2%) мультирезистентных изолятов сальмонелл, у которых были изучены генетические маркеры резистентности. Основным механизмом устойчивости к бета-лактамным антибиотикам у Salmonella spp. является приобретение генов bla, которые кодируют инактивирующие антибиотик ферменты [11]. Несмотря на небольшую долю фенотипически резистентных к бета-лактамным антибиотикам культур, генотипический профиль резистентности изолятов микроорганизмов выявил высокую долю продуцентов бета-лактамаз классов А и С (n = 39; 57,4 ± 0,2%). Большинство из выделенных изолятов микроорганизмов содержали бета-лактамазы расширенного спектра (БЛРС) blaTEM-1B (n = 32; 82,10 ± 0,16%), blaTEM-1C (n = 3; 7,70 ± 0,26%), blaSHV-12 (n = 1; 2,60 ± 0,11%), blaDHA-1 (n = 1; 2,60 ± 0,11%); также были выявлены изоляты серотипа Enteriditis, содержащие цефалоспориназы blaCMY-2 (n = 3; 7,70 ± 0,26%; табл. 2).
Таблица 2. Генотипический профиль резистентности изолятов S. enterica в отношении бета-лактамных антибиотиков
Год Year | Изолят Isolate | Серотип Serotype | MLST | Детерминанты резистентности | Resistance genes | ||||
blaTEM-1B | blaTEM-1C | blaCMY-2 | blaDHA-1 | blaSHV-12 | ||||
2018 | Crie F21 | Enteritidis | ST11 | + | – | – | – | – |
2018 | Crie F28 | Tythimurium | ST34 | + | – | – | – | – |
2018 | Crie F47 | Enteritidis | ST11 | – | + | – | – | – |
2018 | Crie F34 | Mendoza | ST490 | + | – | – | – | – |
2018 | Crie F50 | Tythimurium | ST34 | + | – | – | – | – |
2018 | Crie F40 | Tythimurium | ST34 | + | – | – | – | – |
2018 | Crie F51 | Enteritidis | ST11 | – | + | – | – | – |
2018 | Crie F297 | Enteritidis | ST11 | + | – | – | – | – |
2018 | Crie F46 | Tythimurium | ST9644 | + | – | – | – | – |
2018 | Crie F36 | Tythimurium | ST34 | + | – | – | – | – |
2018 | Crie F37 | Tythimurium | ST34 | + | – | – | – | – |
2018 | Crie F303 | Enteritidis | ST11 | + | – | – | – | – |
2019 | Crie F146 | Enteritidis | ST11 | + | – | – | – | – |
2019 | Crie F296 | Tythimurium | ST19 | + | – | – | – | – |
2019 | Crie F149 | Tythimurium | ST34 | + | – | – | – | – |
2019 | Crie F158 | Brandenburg | ST9644 | + | – | – | – | – |
2019 | Crie F298 | Mendoza | ST490 | + | – | – | – | – |
2019 | Crie F159 | Enteritidis | ST11 | – | + | – | – | – |
2019 | Crie F162 | Enteritidis | ST11 | + | – | – | – | – |
2019 | Crie F163 | Enteritidis | ST11 | + | – | – | – | – |
2019 | Crie F164 | Tythimurium | ST34 | + | – | – | – | – |
2019 | Crie F165 | Tythimurium | ST34 | + | – | – | – | – |
2019 | Crie F167 | Tythimurium | ST34 | + | – | – | – | – |
2019 | Crie F168 | Tythimurium | ST34 | + | – | – | – | – |
2019 | Crie F302 | Enteritidis | ST11 | – | – | + | – | – |
2019 | Crie F353 | Tythimurium | ST34 | + | – | – | – | – |
2020 | Crie F919 | Tythimurium | ST34 | + | – | – | – | – |
2020 | Crie F920 | Tythimurium | ST34 | + | – | – | – | – |
2020 | Crie F923 | Enteritidis | ST11 | – | – | + | – | – |
2020 | Crie F926 | Enteritidis | ST11 | – | – | + | – | – |
2021 | Crie F1149 | Tythimurium | ST34 | + | – | – | – | – |
2021 | Crie F1151 | Tythimurium | ST34 | – | – | – | + | + |
2021 | Crie F1153 | Tythimurium | ST34 | + | – | – | – | – |
2021 | Crie F1154 | Tythimurium | ST34 | + | – | – | – | – |
2021 | Crie F1155 | Tythimurium | ST34 | + | – | – | – | – |
2021 | Crie F1156 | Tythimurium | ST34 | + | – | – | – | – |
2021 | Crie F1157 | Tythimurium | ST34 | + | – | – | – | – |
2021 | Crie F1159 | Tythimurium | ST34 | + | – | – | – | – |
Анализ генотипического профиля чувствительности MDR сальмонелл показал наличие детерминант устойчивости к фторхинолонам у 11 изолятов (16,20 ± 0,33%), кодированных генами qnrB2 (n = 1; 9,10 ± 0,69%), qnrB4 (n = 1; 9,10 ± 0,69%), qnrB5 (n = 1; 9,10 ± 0,69%), qnrB19 (n = 8; 72,70 ± 0,12%) и ферментом аминогликозидацетитрансферазой aac(6’)-Ib-cr (n = 1; 9,10 ± 0,69%), обусловливающую одновременную инактивацию фторхинолонов и аминогликгозидов (табл. 3).
Таблица 3. Генотипический профиль резистентности S. enterica к фторхинолонам
Год Year | Изолят Isolate | Серотип Serotype | MLST | Детерминанты резистентности | Resistance genes | ||||
qnrB2 | qnrB4 | qnrB5 | qnrB19 | aac(6')-Ib-cr | ||||
2018 | Crie F46 | Tythimurium | ST9644 | – | – | – | + | – |
2019 | Crie F298 | Mendoza | ST490 | + | – | – | – | – |
2019 | Crie F353 | Tythimurium | ST34 | – | – | – | + | – |
2020 | Crie F920 | Tythimurium | ST34 | – | – | – | + | – |
2020 | Crie F921 | Infantis | ST32 | – | – | – | + | – |
2020 | Crie F922 | Enteritidis | ST11 | – | – | – | + | – |
2020 | Crie F925 | Enteritidis | ST11 | – | – | – | + | – |
2020 | Crie F926 | Enteritidis | ST11 | – | – | – | + | – |
2021 | Crie F1149 | Tythimurium | ST34 | – | – | + | – | – |
2021 | Crie F1151 | Tythimurium | ST34 | – | + | – | – | + |
2021 | Crie F1159 | Tythimurium | ST34 | – | – | – | + | – |
Несмотря на высокий процент фенотипически чувствительных культур к аминогликозидам, согласно данным полногеномного секвенирования, детерминанты резистентности к данной группе препаратов присутствовали у всех изученных MDR-культур, в том числе у сальмонелл, которые проявили фенотипическую чувствительность к аминогликозидам (n = 61; 89,70 ± 0,08%). Доминирующим геном резистентности, обнаруженным у всех изученных изолятов микроорганизмов, был aac(6’)-Iaa (n = 68; 100%). Основными генетическими маркерами резистентности к аминогликозидам, выявленными в наших исследованиях, являлись гены aadA1 (n = 9; 13,20 ± 0,28%) и aadA2 (n = 6; 8,8 ± 0,2%).
Анализ результатов проведённых нами генотипических исследований MDR-культур сальмонелл выявил у 29,4 ± 0,51% (n = 20) гены плазмидных эффлюкс-помп cmlA1 (n = 4; 20,00 ± 0,83%) и floR (n = 11; 55,00 ± 1,13%), обусловливающих резистентность к фениколам, а также гены инактивации хлорамфеникола посредством фермента CHL-ацетилтрансферазы — catA1 (n = 5; 25,00 ± 0,92%) и catA2 (n = 1; 5,00 ± 0,25%; табл. 4).
Таблица 4. Генотипический профиль резистентности S. enterica к хлорамфениколу
Год Year | Изолят Isolate | Серотип Serotype | MLST | Детерминанты резистентности | Resistance genes | |||
cmlA1 | floR | catA1 | catA2 | ||||
2018 | Crie F21 | Enteritidis | ST11 | + | – | – | – |
2018 | Crie F28 | Tythimurium | ST34 | – | + | – | – |
2018 | Crie F29 | Enteritidis | ST11 | – | – | + | – |
2018 | Crie F40 | Tythimurium | ST34 | – | + | – | |
2018 | Crie F299 | Enteritidis | ST11 | – | – | + | – |
2018 | Crie F36 | Tythimurium | ST34 | – | + | – | – |
2018 | Crie F37 | Tythimurium | ST34 | – | + | – | – |
2018 | Crie F303 | Enteritidis | ST11 | + | – | – | – |
2019 | Crie F146 | Enteritidis | ST11 | + | – | – | – |
2019 | Crie F149 | Tythimurium | ST34 | – | + | – | – |
2019 | Crie F298 | Mendoza | ST490 | – | + | – | – |
2019 | Crie F352 | Enteritidis | ST11 | – | – | + | – |
2019 | Crie F164 | Tythimurium | ST34 | – | + | – | – |
2019 | Crie F165 | Tythimurium | ST34 | – | + | – | – |
2019 | Crie F168 | Tythimurium | ST34 | – | + | – | – |
2019 | Crie F170 | Enteritidis | ST11 | – | – | + | – |
2019 | Crie F171 | Tythimurium | ST34 | – | – | + | – |
2020 | Crie F919 | Tythimurium | ST34 | + | + | – | – |
2020 | Crie F920 | Tythimurium | ST34 | – | + | – | – |
2021 | Crie F1151 | Tythimurium | ST34 | – | – | – | + |
Гены резистентности класса mcr, обусловливающие резистентность к колистину, были выявлены только у одной MDR-культуры Crie F1151, которая фенотипически была чувствительной к колистину. У фенотипически устойчивых к колистину культур гены mcr не выявлены. Генетические детерминанты резистентности к тетрациклинам были выявлены у 51,5 ± 0,2% (n = 35) сальмонелл (табл. 5). Механизмы устойчивости были обусловлены наличием генов, кодирующих эффлюкс-помпы цитоплазматической мембраны: tetB (n = 26; 74,30 ± 0,65%), tetA (n = 9; 25,70 ± 0,67%), tetD (n = 10; 28,60 ± 0,71%); также у изученных культур присутствовал ген резистентности к тетрациклину tetM (n = 1; 2,90 ± 0,15%), обусловливающий механизмы защиты мишени.
Таблица 5. Генотипический профиль резистентности S. enterica к тетрациклинам
Год Year | Изолят Isolate | Серотип Serotype | MLST | Детерминанты резистентности | Resistance genes | |||
tetB | tetA | tetM | tetD | ||||
2018 | Crie F28 | Tythimurium | ST34 | + | – | – | – |
2018 | Crie F29 | Enteritidis | ST11 | – | + | – | – |
2018 | Crie F34 | Mendoza | ST490 | + | – | – | – |
2018 | Crie F50 | Tythimurium | ST34 | + | – | – | – |
2018 | Crie F40 | Tythimurium | ST34 | + | – | – | – |
2019 | Crie F296 | Tythimurium | ST19 | – | + | – | – |
2019 | Crie F147 | Infantis | ST32 | – | + | – | – |
2019 | Crie F149 | Tythimurium | ST34 | + | – | – | – |
2019 | Crie F158 | Brandenburg | ST9644 | + | – | – | – |
2019 | Crie F298 | Mendoza | ST490 | + | – | – | – |
2019 | Crie F352 | Enteritidis | ST11 | – | + | – | – |
2019 | Crie F164 | Tythimurium | ST34 | + | – | – | – |
2019 | Crie F165 | Tythimurium | ST34 | + | – | – | – |
2019 | Crie F167 | Tythimurium | ST34 | + | – | – | – |
2019 | Crie F168 | Tythimurium | ST34 | + | – | – | – |
2019 | Crie F170 | Enteritidis | ST11 | – | + | – | – |
2019 | Crie F171 | Enteritidis | ST11 | – | + | – | – |
2019 | Crie F353 | Tythimurium | ST34 | + | – | – | – |
2018 | Crie F46 | Tythimurium | ST9644 | + | – | – | – |
2018 | Crie F299 | Enteritidis | ST11 | – | + | – | – |
2018 | Crie F36 | Tythimurium | ST34 | + | – | – | – |
2018 | Crie F37 | Tythimurium | ST34 | + | – | – | – |
2020 | Crie F919 | Tythimurium | ST34 | + | – | + | – |
2020 | Crie F920 | Tythimurium | ST34 | + | – | – | – |
2020 | Crie F921 | Infantis | ST32 | – | + | – | – |
2021 | Crie F1148 | Virchow | ST8662 | – | + | – | – |
2021 | Crie F1149 | Tythimurium | ST34 | + | – | – | – |
2021 | Crie F1150 | Tythimurium | ST34 | + | – | – | – |
2021 | Crie F1151 | Tythimurium | ST34 | + | – | – | + |
2021 | Crie F1153 | Tythimurium | ST34 | + | – | – | – |
2021 | Crie F1154 | Tythimurium | ST34 | + | – | – | – |
2021 | Crie F1155 | Tythimurium | ST34 | + | – | – | – |
2021 | Crie F1156 | Tythimurium | ST34 | + | – | – | – |
2021 | Crie F1157 | Tythimurium | ST34 | + | – | – | – |
2021 | Crie F1159 | Tythimurium | ST34 | + | – | – | – |
Детерминанты резистентности к ко-тримоксазолу были выявлены у 50,0 ± 0,2% (n = 34) MDR-культур, генотипический профиль резистентности которых был представлен генами синтеза дегидрофолат-редуктазы dfrA1 (n = 2,00 ± 0,18; 5,9%), dfrA8 (n = 2,00 ± 0,18; 5,9%), dfrA12 (n = 3,00 ± 0,27; 8,8%) и dfrA14 (n = 2,00 ± 0,18; 5,9%), а также генами (n = 33; 48,50 ± 0,59%), продуцирующими невосприимчивые к сульфонамидам дигидроптероат-синтазы sul1 (n = 6; 18,2 ± 0,59%), sul2 (n = 23; 69,7 ± 0,74%) и sul3 (n = 4; 12,1 ± 0,36%; табл. 6).
Таблица 6. Генотипический профиль резистентности S. enterica к ко-тримоксазолу
Год Year | Изолят Isolate | Серотип Serotype | ST | Детерминанты резистентности | Resistance genes | ||||||
sul3 | sul2 | sul1 | dfrA12 | dfrA8 | dfrA14 | dfrA1 | ||||
2018 | Crie F21 | Enteritidis | ST11 | + | – | – | – | – | – | – |
2018 | Crie F28 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2018 | Crie F34 | Mendoza | ST490 | – | – | + | + | – | – | – |
2018 | Crie F50 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2018 | Crie F40 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2018 | Crie F46 | Tythimurium | ST9644 | – | + | – | – | – | – | – |
2018 | Crie F36 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2018 | Crie F37 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2018 | Crie F303 | Enteritidis | ST11 | + | – | – | – | + | – | – |
2019 | Crie F146 | Enteritidis | ST11 | + | – | – | – | – | – | – |
2019 | Crie F296 | Tythimurium | ST19 | + | – | – | – | – | – | – |
2019 | Crie F147 | Infantis | ST32 | – | – | + | – | – | – | – |
2019 | Crie F149 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2019 | Crie F158 | Brandenburg | ST9644 | – | + | – | – | – | – | – |
2019 | Crie F298 | Mendoza | ST490 | – | – | + | + | – | – | – |
2019 | Crie F164 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2019 | Crie F165 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2019 | Crie F166 | Enteritidis | ST11 | – | – | – | – | + | – | – |
2019 | Crie F167 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2019 | Crie F168 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2019 | Crie F353 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2020 | Crie F919 | Tythimurium | ST34 | – | + | – | + | – | – | – |
2020 | Crie F920 | Tythimurium | ST34 | – | + | – | – | – | + | – |
2020 | Crie F921 | Infantis | ST32 | – | – | + | – | – | + | – |
2021 | Crie F1148 | Virchow | 8662 | – | – | + | – | – | – | + |
2021 | Crie F1149 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2021 | Crie F1150 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2021 | Crie F1151 | Tythimurium | ST34 | – | – | + | – | – | – | + |
2021 | Crie F1153 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2021 | Crie F1154 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2021 | Crie F1155 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2021 | Crie F1156 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2021 | Crie F1157 | Tythimurium | ST34 | – | + | – | – | – | – | – |
2021 | Crie F1159 | Tythimurium | ST34 | – | + | – | – | – | – | – |
Следует отметить, что несмотря на наличие детерминант антибиотикорезистентности, 10 культур были полностью чувствительными ко всем изученным антибиотикам — Crie F146, Crie F149, Crie F158, Crie F159, Crie F162, Crie F163, Crie F164, Crie F165, Crie F167 и Crie F168. Кроме этого, в нашем исследовании не было выявлено карбапенемаз классов А (KPC) и В (GIM, VIM, IMP, NDM, SPM и FOX).
Практически все культуры Salmonella, продуцирующие БЛРС или AmpC (n = 20; 51,30 ± 0,27%), характеризовались полной фенотипической чувствительностью к другим, не-бета-лактамным классам антибиотиков, включая фторхинолоны; 46,20 ± 0,27% (n = 18) изолятов микроорганизмов были резистентными к 1-2 нелактамным классам антибиотиков.
При анализе MLST in silico выявлены 10 различных сиквенс-типов MDR-изолятов S. enterica: ST11 серовара Enteritidis (n = 31; 47,0 ± 0,57%), ST34 и ST19 серовара Tythimurium (n = 21; 33,30 ± 0,51% и n = 3; 4,50 ± 0,10% соответственно), ST32 серовара Infantis (n = 4; 6,10 ± 0,13%), ST490 серовара Mendoza (n = 3; 4,50 ± 0,10%), ST897 серовара Bredeney (n = 1; 1,50 ± 0,03%), ST8662 серовара Virchow (n = 1; 1,50 ± 0,03%), ST1992 серовара London (n = 1; 1,00 ± 0,03%), ST1986 серовара Stanleyville (n = 1; 1,00 ± 0,03%). У культур сальмонелл Crie F46 и Crie F158 был выявлен новый сиквенс-тип ST9644. Доминирующие сиквенс-типы ST11, ST34, ST32, ST490 и ST19 были связаны со множественной резистентностью культур, которые содержали детерминанты резистентности к 7 классам антибиотиков (табл. 2–6), в то время как сиквенс-типы ST897, ST1992 и ST1986 содержали гены, обусловливающие резистентность только к аминогликозидам.
Культура Crie F1151 была выделена из кулинарной продукции и таксономически относилась к сальмонеллам серотипа Tythimurium. Культура отличалась фенотипической мультирезистентностью к пенициллинам, цефалоспоринам, азтреонаму, фторхинолонам, аминогликозидам, триметоприм/сульфаметоксазолу и тетрациклинам, кодируемым соответствующими детерминантами резистентности: aac(6')-IIc, aac(6')-Iaa, aadA1, aph(3')-Ia, blaDHA-1, blaSHV-12, blaTEM-1B, aac(6')-Ib-cr, catA2, qnrB4, sul1, dfrA1 и tetB, tetD соответственно. Генотипический профиль резистентности культуры S. tyhimurium Crie F1151 также содержал детерминанты, обусловливающие резистентность к макролидам ereA. Данный изолят сальмонеллы был единственным, у которого была выявлена детерминанта mcr-9, несмотря на фенотипическую чувствительность к колистину, и aac(6')-Ib-cr, обусловливающая одновременную резистентность к фторхинолонам и аминогликозидам.
Обсуждение
Сальмонеллёз является часто регистрируемой желудочно-кишечной инфекцией у людей и важной причиной вспышек пищевых отравлений во всём мире. В Европейском союзе (ЕС) в 2019 г. число подтверждённых случаев сальмонеллёза составило 87 923 человека; в 2020 г. — 57 702 человека, что стало самым низким зарегистрированным числом с 2007 г. из-за последствий выхода Великобритании из ЕС и пандемии COVID-19 [9].
Нами были изучены культуры S. enterica, выделенные из различных пищевых продуктов на территории Республики Беларусь в 2018–2021 гг., с целью оценки их профилей чувствительности к антибиотикам. Наши данные показали, что самыми распространёнными источниками сальмонелл были мясо свинины и птицепродукты, в том числе обработанные. При этом в отношении птицепродуктов можно отметить, что распространённость резистентных сальмонелл значительно увеличилась за период исследований — с 19,8% в 2018 г. до 65,1% в 2021 г. Наблюдаемая в наших исследованиях распространённость сальмонелл в основном в продукции из мяса свинины, курицы и индейки была сравнима с зарегистрированными в США, Египте и Колумбии [12–14]. Преобладающими серотипами за все годы исследований были Enteritidis (50,8%) и Typhimurium (9,0%). Доминирование этих серотипов в мясной продукции также выявлено в исследованиях из Индии и Саудовской Аравии, где на долю серотипов Enteritidis и Typhimurium приходилось более 95% изолятов [3, 11]. Согласно данным доклада EC о зоонозах «Единое здоровье» (2020), в европейском регионе также доминировал серотип Enteritidis [15].
Анализ фенотипической резистентности культур, выделенных на территории Республики Беларусь, показал высокую чувствительностью к препаратам фторхинолоновой группы — от 88,4 до 100%. Однако на протяжении периода мониторинга наблюдался рост резистентных культур до 11,6%. В США на протяжении 2018–2021 гг., по данным Центров по контролю и профилактике заболеваний в США2, также наблюдался общий тренд растущей резистентности сальмонелл к действию ципрофлоксацина на 8,5%. Высокая активность фторхинолонов также показана в отношении сальмонелл, выделенных из мяса свинины в Таиланде: чувствительными были 76% изученных культур. У S. еnteritidis, наиболее распространённого типа сальмонелл у людей, наблюдались тенденции к увеличению резистентности к антибиотикам класса фторхинолонов. По данным Европейского центра профилактики и контроля заболеваний, у животных устойчивость S. еnteritidis к этим антибиотикам была от умеренной до высокой [16].
Уровень фенотипической устойчивости к цефалоспоринам 3-го поколения сальмонелл, выделенных на территории Республики Беларусь, был невысоким: до 16,3%. За 2018–2021 гг. наблюдался рост резистентных к цефалоспоринам 3-го поколения культур в 5,6 раза: с 2,9% в 2019 г. до 16,3% в 2021 г. Согласно данным Центров по контролю и профилактике заболеваний в США3, доля резистентных культур к цефалоспоринам в выбранный период времени также была небольшой: 2,3% в 2018 г., 1,7% в 2019 г. и стабильно держалась на отметке 2% в 2020 и 2021 гг. В отчёте, опубликованном Европейским центром профилактики и контроля заболеваний и Европейским агентством по безопасности пищевых продуктов, указано, что в 2019 г. доля цефотаксим-резистентных и цефтазидим-резистентных культур в европейском регионе сохранялась на низких уровнях — 1,8 и 1,2% соответственно [17].
Распространение MDR-сальмонелл представляет серьёзную проблему для здоровья, поскольку онb вызыва.т более длительные госпитализации, продолжительные болезни и более высокий уровень смертности, чем чувствительные изоляты сальмонелл [17, 18]. Европейский центр профилактики и контроля заболеваний в докладе в 2021 г. отметил выросшую до критических отметок долю MDR- изолятов S. enterica из мяса свинины и её полуфабрикатов: до 56,5% [17]. По оценкам Всемирной организации здравоохранения, из 100 тыс. случаев сальмонеллеза каждый год большое количество вызвано MDR-изолятами S. enterica [19], причём большинство из них произошло в результате употребления заражённых продуктов животного происхождения, особенно говядины, свинины и продуктов из птицы [20, 21]. Среди изученных изолятов сальмонелл, выделенных на территории Республики Беларусь, 19% культур показали профиль MDR с устойчивостью к 3 и более классам антимикробных препаратов, что сравнимо с данными исследований египетских учёных [22]. В США в 2008–2017 гг. устойчивость к 3 и более препаратам проявляли 28,0% изученных бактерий, изолированных из птицепродуктов [23]. В исследованиях китайских коллег MDR-устойчивость показывали 95,33% сальмонелл, выделенных из мяса свинины [24]; таиландскими учёными также была отмечена мультирезистентность 23,2% сальмонелл, выделенных из мяса уток [25].
Анализ MLST in silico MDR-изолятов сальмонелл, выделенных на территории Республики Беларусь, выявил 5 сиквенс-типов S.enterica, связанных с множественной устойчивостью культур сальмонелл. Доминирующими сиквенс-типами были ST11 серовара Enteritidis (47,0 ± 0,57%), ST34 (33,3 ± 0,51%) и ST19 (4,5 ± 0,10%) серовара Tythimurium. Данные сиквенс-типы были также распространёнными в Китае и Ираке, где среди сальмонелл серовара Tythimurium преобладал ST19 [26–28]. В ЕС превалирующим сиквенс-типом был ST11 серовара Enteritidis, в то время как на территории России доминирующим является серотип Infantis ST32 [29].
Проведённые нами генотипические исследования культур, выделенных на территории Республики Беларусь, выявили 5 генов, обусловливающих резистентность к бета-лактамным антибиотикам: blaTEM-1B, blaTEM-1C, blaDHA-1, blaSHV-12, а также цефалоспориназы класса blaCMY-2. Вероятнее всего, ежегодный рост доли фенотипически резистентных культур связан с активацией генов резистентности БЛРС, т.к. исследования показали, что 56,5% всех исследованных MDR-изолятов продуцировали БЛРС. Ген blaSHV идентифицирован в основном у представителей семейства Enterobacteriaceae, изолированных из различных экосистем: человека, животных и окружающей среды [30, 31]. Вероятно, продуцируемые хромосомной пенициллиназой Klebsiella pneumoniae, SHV бета-лактамазы в настоящее время включают большое количество аллельных вариантов: БЛРС, не-БЛРС и несколько неклассифицированных вариантов, ввиду чего они были выделены в наших исследованиях [32].
Фенотипическая характеристика большинства MDR-культур (n = 59; 85,5%) соответствовала молекулярному механизму резистентности и была обусловлена спектром ферментативной активности бета-лактамаз. Полученные данные полного совпадения фенотипических и генотипических признаков устойчивости культур из Беларуси к цефалоспоринам 3-го поколения подтвердили диагностическую ценность индикаторных препаратов цефтазидима, цефтриаксона, цефоперазона и цефотаксима. При трехлетнем мониторинге европейскими лабораториями за сальмонеллами, выделенными из различных категорий пищевых продуктов, была также показана диагностическая ценность для обнаружения цефалоспориназ индикаторных препаратов цефтриаксона, цефтазидима и цефотаксима [9]. Резкое увеличение доли фенотипически резистентных культур к тетрациклину (с 3,9 до 23,3%) наряду с высоким процентом культур (52,2%), содержащих детерминанты резистентности, могут свидетельствовать о чрезмерном употреблении антибиотиков из группы тетрациклинов в сельском хозяйстве, за счёт чего происходят накопление и передача генетического материала антибиотикорезистентности между патогенными бактериями. Наличие детерминант антибиотикорезистентности у чувствительных культур, а также наличие генов ereA и mcr-9 у S. typhimurium Crie F-1151 может служить подтверждением потенциала сальмонелл выступать в качестве вектора передачи генов антибиотикорезистентности другим микроорганизмам. По данным литературы, детерминанты mcr-9 наряду с детерминантами mcr-1 считаются наиболее распространёнными в мире [33]. По данным Национальной базы данных организмов, устойчивыми к антибиотикам лидерами по количеству mcr-9-позитивных изолятов являются США. В европейском регионе и на территории России, а также в Китае доминирующими являются детерминанты mcr-9 и mcr-1 [33].
Полученные данные, безусловно, вызывают тревогу, поскольку все исследуемые нами изоляты микроорганизмов были получены из образцов продуктов питания, предназначенных для употребления в пищу человеком. Несмотря на снижение рисков заболеваний пищевого происхождения при термической обработке продуктов питания, гены антибиотикорезистентности сохраняются и при попадании в организм могут передаваться микробиоценозу кишечника, передавая резистентность другим микроорганизмам [34]. Таким образом, наши данные соответствуют последним рекомендациям Европейского агентства по безопасности продуктов питания о необходимости изучения фенотипической и генотипической характеристики изолятов микроорганизмов пищевого происхождения при мониторинге и эпиднадзоре за устойчивостью к антибиотикам, особенно в подходе «Единое здоровье», который признает взаимосвязь здоровья человека, животных и окружающей среды.
Заключение
Нерациональное использование антибиотиков в медицине и ветеринарии во многом способствовало возникновению и распространению резистентных изолятов нетифоидных сальмонелл.
Полученные в результате наших исследований данные о медленно растущей фенотипической резистентности к антибиотикам 1-й линии (цефалоспоринам и фторхинолонам) и наличии плазмид- опосредованных детерминант резистентности говорят о возможном серьёзном ограничении выбора эффективных противомикробных препаратов в будущем. По этой причине крайне важно отслеживать фенотипы и генотипы устойчивости к противомикробным препаратам, а также пути передачи культур S. enterica через пищевую цепь.
Наличие большого разнообразия детерминант резистентности при довольно высокой фенотипической чувствительности культур позволяет предположить, что источники выделения изолятов микроорганизмов могли подвергаться воздействию антибиотиков и/или приобретать детерминанты резистентности от других микроорганизмов.
Проведённые исследования свидетельствуют о необходимости проведения дальнейшего мониторинга распространённости устойчивых к антибиотикам бактерий пищевого происхождения на территории Республики Беларусь, особенно с точки зрения подхода «Единое здоровье».
1 URL: https://bigsdb.pasteur.fr/
2 NARMS Now: Human Data.
URL: https://wwwn.cdc.gov/narmsnow/
3 NARMS Now: Human Data.
URL: https://wwwn.cdc.gov/narmsnow/
Об авторах
Нина Георгиевна Куликова
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Автор, ответственный за переписку.
Email: kulikova_ng@cmd.su
ORCID iD: 0000-0002-1716-6969
к.б.н., с.н.с. направления клинической микробиологии лаб. клинической микробиологии и микробной экологии человека
Россия, МоскваАлексей Валерьевич Чернышков
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: chernyshkov@cmd.su
ORCID iD: 0000-0002-7049-4195
к.м.н., с.н.с. направления клинической микробиологии лаб. клинической микробиологии и микробной экологии человека
Россия, МоскваЮлия Владимировна Михайлова
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: mihailova@cmd.su
ORCID iD: 0000-0002-5646-538X
к.б.н., зав. лаб. молекулярных механизмов антибиотикорезистентности
Россия, МоскваАлександр Леонидович Зенькович
Республиканский центр гигиены, эпидемиологии и общественного здоровья
Email: kulikova_ng@cmd.su
зам. главного врача по лабораторному делу
Белоруссия, МинскДарья Александровна Довнар
Республиканский центр гигиены, эпидемиологии и общественного здоровья
Email: kulikova_ng@cmd.su
врач-бактериолог микробиологической референс-лаборатории
Белоруссия, МинскАлла Михайловна Марейко
Республиканский центр гигиены, эпидемиологии и общественного здоровья
Email: kulikova_ng@cmd.su
врач-бактериолог микробиологической референс-лаборатории
Белоруссия, МинскЛюция Айткалиевна Битюмина
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: bitumina@cmd.su
ORCID iD: 0000-0002-5378-0827
врач-бактериолог направления клинической микробиологии лаб. клинической микробиологии и микробной экологии человека
Россия, МоскваАндрей Александрович Шеленков
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: shelenkov@cmd.su
ORCID iD: 0000-0002-7409-077X
к.ф-м.н., с.н.с. лаб. молекулярных механизмов антибиотикорезистентности
Россия, МоскваАнна Евгеньевна Егорова
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: a.egorova@cmd.su
ORCID iD: 0000-0003-0486-1353
н.с. лаб. молекулярных механизмов антибиотикорезистентности
Россия, МоскваСтепан Сергеевич Саенко
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: saenko@cmd.su
ORCID iD: 0000-0002-4925-1308
м.н.с. лаб. молекулярных механизмов антибиотикорезистентности
Россия, МоскваИгорь Николаевич Манзенюк
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: manzeniuk@cmd.su
ORCID iD: 0000-0002-1146-1430
к.м.н., помощник директора по научной работе
Россия, МоскваСписок литературы
- Павлова А.С., Бочарова Ю.А., Кулешов К.В. и др. Молекулярные детерминанты резистентности Salmonella enterica к антибиотикам. Журнал микробиологии, эпидемиологии и иммунобиологии. 2021;98(6):721–30. Pavlova A.S., Bocharova Yu.A., Kuleshov K.V., et al. Molecular determinants of antibiotic resistance in Salmonella enterica antibiotic resistance. Journal of microbiology, epidemiology and immunobiology. 2021;98(6):721–30. DOI: https://doi.org/10.36233/0372-9311-140. EDN: https://elibrary.ru/zqrygu
- Решетнева И.Т., Перьянова О.В., Дмитриева Г.М., Остапова Т.С. Антибиотикорезистентность сальмонелл, выделенных на территории Красноярского края. Гигиена и санитария. 2015;94(2):35–8. Reshetneva I.T., Per'yanova O.V., Dmitrieva G.M., Ostapova T.S. Antibiotic resistance of Salmonella spp. isolated in the territory of the Krasnoyarsk region. Hygiene and Sanitation. 2015;94(2):35–8. EDN: https://elibrary.ru/tphjlx
- Al-Ansari M.M., Aljubali M.M., Somily A.M., et al. Isolation and molecular characterization of multidrug-resistant Salmonella enterica serovars. J. Infect. Public Health. 2021;14(12):1767–76. DOI: https://doi.org/10.1016/j.jiph.2021.10.011
- Похиленко В.Д., Баранов А.М., Детушев К.В. Методы длительного хранения коллекционных культур микроорганизмов и тенденции развития. Известия высших учебных заведений. Поволжский регион. Медицинские науки. 2009;(4):99–121. Pokhilenko V.D., Baranov A.M., Detushev K.V. Methods of long-term storage of collection cultures of microorganisms and development trends. Izvestiya vysshikh uchebnykh zavedeniy. Povolzhskiy region. Meditsinskie nauki. 2009;(4):99–121. EDN: https://elibrary.ru/lakpfx
- Bankevich A., Nurk S., Antipov D., et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. J. Comput. Biol. 2012;19(5):455–77. DOI: https://doi.org/10.1089/cmb.2012.0021
- Shelenkov A., Mikhaylova Y., Yanushevich Y., et al. Molecular typing, characterization of antimicrobial resistance, virulence profiling and analysis of whole-genome sequence of clinical Klebsiella pneumoniae isolates. Antibiotics (Basel). 2020;9(5):261. DOI: https://doi.org/10.3390/antibiotics9050261
- Zankari E., Hasman H., Cosentino S., et al. Identification of acquired antimicrobial resistance genes. J. Antimicrob. Chemother. 2012;67(11):2640–4. DOI: https://doi.org/10.1093/jac/dks261
- Яковлев В.П. Антимикробные препараты группы фторхинолонов. Consilium Medicum. 2006;8(1):35–41. Yakovlev V.P. Antimicrobial preparations of fluoroquinolones group. Consilium Medicum. 2006;8(1):35–41. EDN: https://elibrary.ru/rglbxl
- Gambino D., Gargano V., Butera G., et al. Food is reservoir of MDR Salmonella: prevalence of ESBLs profiles and resistance genes in strains isolated from food. Microorganisms. 2022;10(4):780. DOI: https://doi.org/10.3390/microorganisms10040780
- Ngoi S.T., Teh C.S., Chai L.C., Thong K.L. Overview of molecular typing tools for the characterization of Salmonella enterica in Malaysia. Biomed. Environ. Sci. 2015;28(10):751–64. DOI: https://doi.org/10.3967/bes2015.105
- Kumar Y., Singh V., Kumar G., et al. Serovar diversity of Salmonella among poultry. Indian J. Med. Res. 2019;150(1):92–5. DOI: https://doi.org/10.4103/ijmr.IJMR_1798_17
- Mamber S.W., Mohr T., Barlow K., et al. Occurrence of Salmonella in ready-to-eat meat and poultry product samples from U.S. Department of Agriculture-Regulated Producing Establishments. II. Salmonella in ready-to-eat pork barbecue products, from 2005 to 2012. J. Food Prot. 2018;81(10):1737–42. DOI: https://doi.org/10.4315/0362-028X.JFP-18-026
- Rodríguez R., Fandiño C., Donado P., et al. Characterization of Salmonella from commercial egg-laying hen farms in a central region of Colombia. Avian Dis. 2015;59(1):57–63. DOI: https://doi.org/10.1637/10873-052714-reg
- Elkenany R., Elsayed M.M., Zakaria A.I., et al. Antimicrobial resistance profiles and virulence genotyping of Salmonella enterica serovars recovered from broiler chickens and chicken carcasses in Egypt. BMC Vet. Res. 2019;15(1):124. DOI: https://doi.org/10.1186/s12917-019-1867-z
- European Food Safety Authority; European Centre for Disease Prevention and Control. The European Union One Health 2020 Zoonoses Report. EFSA J. 2021;19(12):e06971. DOI: https://doi.org/10.2903/j.efsa.2021.6971
- European Food Safety Authority; European Centre for Disease Prevention and Control. The European Union Summary Report on Antimicrobial Resistance in zoonotic and indicator bacteria from humans, animals and food in 2019-2020. EFSA J. 2022;20(3):e07209. DOI: https://doi.org/10.2903/j.efsa.2022.7209
- European Food Safety Authority; European Centre for Disease Prevention and Control. The European Union Summary Report on Antimicrobial Resistance in zoonotic and indicator bacteria from humans, animals and food in 2018/2019. EFSA J. 2021;19(4):e06490. DOI: https://doi.org/10.2903/j.efsa.2021.6490
- Proietti P.C., Stefanetti V., Musa L., et al. Genetic profiles and antimicrobial resistance patterns of Salmonella infantis strains isolated in Italy in the food chain of broiler meat production. Antibiotics (Basel). 2020;9(11):814. DOI: https://doi.org/10.3390/antibiotics9110814
- WHO. Antimicrobial Resistance: Global Report on Surveillance. Geneva; 2014.
- Castro-Vargas R.E., Herrera-Sánchez M.P., Rodríguez-Hernández R., Rondón-Barragán I.S. Antibiotic resistance in Salmonella spp. isolated from poultry: a global overview. Vet. World. 2020;13(10):2070–84. DOI: https://doi.org/10.14202/vetworld.2020.2070-2084
- Campos J., Mourão J., Peixe L., Antunes P. Non-typhoidal Salmonella in the pig production chain: a comprehensive analysis of its impact on human health. Pathogens. 2019;8(1):19. DOI: https://doi.org/10.3390/pathogens8010019
- Lamas A., Fernandez-No I.C., Miranda J.M., et al. Prevalence, molecular characterization and antimicrobial resistance of Salmonella serovars isolated from northwestern Spanish broiler flocks (2011–2015). Poult. Sci. 2016;95(9):2097–105. DOI: https://doi.org/10.3382/ps/pew150
- Yin X., M'ikanatha N.M., Nyirabahizi E., et al. Antimicrobial resistance in non-Typhoidal Salmonella from retail poultry meat by antibiotic usage-related production claims – United States, 2008–2017. Int. J. Food Microbiol. 2021;342:109044. DOI: https://doi.org/10.1016/j.ijfoodmicro.2021.109044
- Xu Z., Chen X., Tan W., et al. Prevalence and antimicrobial resistance of Salmonella and Staphylococcus aureus in fattening pigs in Hubei Province, China. Microb. Drug Resist. 2021;27(11):1594–602. DOI: https://doi.org/10.1089/mdr.2020.0585
- Sinwat N., Witoonsatian K., Chumsing S., et al. Antimicrobial resistance phenotypes and genotypes of Salmonella spp. isolated from commercial duck meat production in Thailand and their minimal inhibitory concentration of disinfectants. Microb. Drug Resist. 2021;27(12):1733–41. DOI: https://doi.org/10.1089/mdr.2020.0230
- Yang J., Zhang Z., Zhou X., et al. Prevalence and characterization of antimicrobial resistance in Salmonella enterica isolates from retail foods in Shanghai, China. Foodborne Pathog. Dis. 2020;17(1):35–43. DOI: https://doi.org/10.1089/fpd.2019.2671
- Zhao X., Hu M., Zhang Q., et al. Characterization of integrons and antimicrobial resistance in Salmonella from broilers in Shandong, China. Poult. Sci. 2020;99(12):7046–54. DOI: https://doi.org/10.1016/j.psj.2020.09.071
- Harb A., Habib I., Mezal E.H., et al. Occurrence, antimicrobial resistance and whole-genome sequencing analysis of Salmonella isolates from chicken carcasses imported into Iraq from four different countries. Int. J. Food Microbiol. 2018; 284:84–90. DOI: https://doi.org/10.1016/j.ijfoodmicro.2018.07.007
- Egorova A., Mikhaylova Y., Saenko S., et al. Comparative whole-genome analysis of Russian foodborne multidrug-resistant Salmonella infantis isolates. Microorganisms. 2021;10(1):89. DOI: https://doi.org/10.3390/microorganisms10010089
- Alcalá L., Alonso C.A., Simón C., et al. Wild birds, frequent carriers of extended-spectrum β-lactamase (ESBL) producing Escherichia coli of CTX-M and SHV-12 types. Microb. Ecol. 2016;72(4):861–9. DOI: https://doi.org/10.1007/s00248-015-0718-0
- Liakopoulos A., Mevius D., Ceccarelli D. A review of SHV extended-spectrum β-lactamases: neglected yet ubiquitous. Front. Microbiol. 2016;7:1374. DOI: https://doi.org/10.3389/fmicb.2016.01374
- Bush K. Past and present perspectives on β-lactamases. Antimicrob. Agents Chemother. 2018;62(10):e01076–18. DOI: https://doi.org/10.1128/AAC.01076-18
- Ling Z., Yin W., Shen Z., et al. Epidemiology of mobile colistin resistance genes MCR-1 to MCR-9. J. Antimicrob. Chemother. 2020;75(11):3087–95. DOI: https://doi.org/10.1093/jac/dkaa205
- Groussin M., Poyet M., Sistiaga A., et al. Elevated rates of horizontal gene transfer in the industrialized human microbiome. Cell. 2021;184(8):2053–67.e18. DOI: https://doi.org/10.1016/j.cell.2021.02.052
Дополнительные файлы