Multiplex PCR screening for virulence genes of Klebsiella pneumoniae isolated from microbiota of diseased and healthy people
- Authors: Pay G.V.1, Rakitina D.V.1, Sukhina M.A.1,2, Yudin S.M.1, Makarov V.V.1, Maniya T.R.1, Zagaynova A.V.1
-
Affiliations:
- Centre for Strategic Planning and Management of Biomedical Health Risk of the Federal Medical Biological Agency
- State Scientific Center of Coloproctology
- Issue: Vol 99, No 4 (2022)
- Pages: 436-444
- Section: ORIGINAL RESEARCHES
- URL: https://microbiol.crie.ru/jour/article/view/1312
- DOI: https://doi.org/10.36233/0372-9311-237
- ID: 1312
Cite item
Full Text
Abstract
Introduction. Klebsiella pneumoniae in human microbiota may appear as a part of commensal microbiota, and as hypervirulent pathogen, for example, hypermucoid pathotype. This pathotype is characterized by certain genetic determinants, leading to the possibility of detecting the pathogenic potential of isolates by PCR.
Aim of the study: to evaluate and compare pathogenic potential of K. pneumoniae isolates from practically healthy people, patients with inflammatory bowel disease (IBD) and extraintestinal infections (ExII).
Materials and methods. Testing was performed with the set of nucleotides for multiplex PCR analysis targeting eight potentially virulent genes with the following functions: ferrum uptake (ybsT, kfu, iutA), adhesion and invasion (mrkD), hypermucoid phenotype and virulent serotypes (mrkD, magA, rmpA, k2) and metabolism of allantoin (allS). PCR assay was used to screen Klebsiella pneumoniae isolates from feces of patients with IBD (69 isolates) and of practically healthy people (68 isolates), and multiresistant isolates from biological material (blood, urine, surgical wounds, bronchoalveolar lavage) of patients with extraintestinal infections (mrExII, 25 isolates).
Results. Results of the testing demonstrated association of four of targeted determinants with the patients diagnoses. YbtS gene was significantly more often found in isolates from IBD (р = 0.024) and mrExII (p < 0.001) groups. RmpA gene was significantly more often detected in IBD group (р = 0.038). Extraintestinal infectious isolates were significantly (р ≤ 0.001) enriched with mrkD and allS genes (р = 0.032).
Conclusion. The most potentially virulent group was isolated from patients with extraintestinal infections, the least virulent — isolates from feces of practically healthy people. The most frequently detected virulence genes were involved in adhesion and hypermucoid phenotype formation.
Full Text
ВВЕДЕНИЕ
Klebsiella pneumoniae является представителем нормальной резидентной микрофлоры верхних дыхательных путей и желудочно-кишечного тракта у «практически» здоровых людей [1]. С другой стороны, K. pneumoniae является клинически значимым условно-патогенным микроорганизмом и может попадать в организм человека как из внешней среды (в том числе с водой), так и в условиях внутрибольничного инфицирования [1–3]. K. pneumoniae способна поражать различные системы и органы при пневмониях, менингитах, инфекциях мочеполовых путей, абсцессах и инфекциях ран [4][5]. Описан ряд случаев, когда K. pneumonia вызывала эпидемические вспышки [6][7]. В последнее время предполагается участие K. pneumoniae в патогенезе различных воспалительных заболеваний кишечника (ВЗК) [8][9].
Поскольку K. pneumoniae может быть как частью нормальной микробиоты кишечника, так и инфекционным агентом, для диагностики и лечения встаёт вопрос об участии в патогенезе заболевания конкретного изолята, выделенного от пациента. Патогенность K. pneumoniae может быть обусловлена различными факторами, обеспечивающими защиту от атаки иммунной системы организма, способность к адгезии и инвазии в клетки хозяина, выработку токсинов, а также устойчивость к антибиотикам [10–12]. Система поглощения железа, включающая секрецию сидерофоров (белков и пептидов, ответственных за захват и импорт ионов железа), также способна вносить вклад в патогенез, индуцируя воспаление и усиленный бактериальный рост [13].
В последние годы выделяют определённый патотип гипервирулентных бактерий K. pneumoniae (hvKp). Изоляты клебсиелл, относящиеся к hvKp, способны вызывать кишечные и внекишечные поражения (затрагивающие печень, лёгкие, мочеполовые пути и т.д.) и распространяются как в обществе, так и в качестве госпитальных инфекций [10][14].
Определяющим фенотипическим признаком hvKp является гипермукоидный фенотип. Он легко выявляется «нитевым тестом»: колония бактерий на агаровой чашке может быть растянута инокуляционной петлей как минимум на 5 мм [15].
Другими признаками hvKp считаются устойчивость к антибиотикам [16], повышенный биосинтез полисахаридов бактериальной капсулы [17] и специфические серотипы бактериальной капсулы, а также повышенная продукция определённых сидерофоров [18]. Все эти признаки связаны с определёнными генами, находящимися как в плазмидах вирулентности, так и в мобильных элементах, интегрированных в геном бактерии [17]. Таким образом, по крайней мере предварительно, вирулентность бактерии может быть оценена не микробиологическими, биохимическими методами и тестами на животных, а методом генетического анализа с применением полимеразной цепной реакции (ПЦР). Выявление корректных генов-мишеней у микроорганизмов необходимо для разработки диагностических наборов, планируемых для использования в клинических лабораториях, при эпидемиологическом мониторинге и в научных исследованиях.
Цель работы — оценить патогенный потенциал изолятов K. pneumoniae, изолированных от «практически» здоровых лиц, пациентов с ВЗК, и штаммов, выделенных из биологического материала пациентов с внекишечными инфекциями (ВКИ) посредством детекции генов, связанных с вирулентностью.
МАТЕРИАЛЫ И МЕТОДЫ
Исследование проводили с применением набора олигонуклеотидов для мультиплексного анализа, предложенного F. Compain и соавт. [19], — 8 генов, предположительно ассоциированных с вирулентностью, с функцией захвата железа (3 гена), адгезии и инвазии (3 гена), вирулентного серотипа и метаболизма аллантоина (продукта расщепления пуринов и мочевой кислоты).
ИЗОЛЯТЫ И ПАЦИЕНТЫ
Бактериальные изоляты получены из музейной коллекции Центра стратегического планирования ФМБА России. Изоляты K. pneumoniae были выделены культуральным методом на плотных дифференциальных средах Эндо («HiMedia»), Агар BСР («Conda»), Chromocult Conform Agar («Merck») из кала 69 пациентов с ВЗК и 68 «практически» здоровых людей (без жалоб на проблемы с желудочно-кишечным трактом). Изучены также 25 штаммов из раневых поверхностей, крови, мочи и бронхоальвеолярного лаважа от пациентов с мультирезистентными ВКИ (мрВКИ) со множественной устойчивостью к антибиотикам, предоставленные НМНИЦ колопроктологии им. А.Н. Рыжих.
Видовую идентификацию изолятов проводили методом матрично-активированной лазерной десорбции/ионизации с времяпролётной масс-спектрометрией (MALDI-TOF MS) на анализаторе «Microoflex MALDI-TOF MS» («Bruker Daltonics GmbH & Co. KG») с использованием базы бактериальных белковых спектров и программного обеспечения «MALDI Biotyper» («Bruker Daltonics GmbH & Co. KG»). Идентификацию проводили согласно протоколу производителя, достоверным считали результат c Z-score > 2.
Критерии включения пациентов для группы ВЗК:
- возраст старше 18 лет;
- диагностированное гастроэнтерологом ВЗК (язвенный колит или болезнь Крона);
- отсутствие антибиотикотерапии в течение последних 3 мес.
Критерии включения пациентов для группы практически здоровых людей:
- возраст старше 18 лет;
- отсутствие жалоб на здоровье в области желудочно-кишечного тракта;
- нормальный стул;
- отсутствие антибиотикотерапии в течение последних 3 мес.
У всех пациентов было получено добровольное информированное согласие на сбор материала и проведение исследования. Протокол исследования одобрено Локальным независимым этическим комитетом (протокол № 98А заседания Локального независимого этического комитета ФГБУ «ГНЦК им. А.Н. Рыжих» от 16.07.2018).
Все выделенные изоляты K. pneumoniae хранятся в рабочей коллекции ЦСП ФМБА в среде для длительного хранения живых бактериальных клеток при –70ºС1.
ВЫДЕЛЕНИЕ ДНК
Культивированные бактериальные клетки промывали стерильным физиологическим раствором, собирали центрифугированием, ресуспендировали в стерильном физрастворе и лизировали прогреванием при 70ºС в течение 15 мин. Клеточный дебрис осаждали центрифугированием в течение 10 мин при 6000 об/мин, а надосадочную жидкость, содержащую бактериальную ДНК, использовали для постановки ПЦР.
ПЦР-АНАЛИЗ
ПЦР-анализ проводили на приборе «Bio-Rad Thermal Cycler T100» («Bio-Rad Laboratories, Inc.») с использованием синтетических олигонуклеотидов, предложенных F. Compain и соавт. [19], для мультиплексного анализа (табл. 1) и реактивов набора для ПЦР с Taq-полимеразой (PK114, «Евроген») согласно протоколу производителя.
Таблица 1. Используемые в работе последовательности для синтетических олигонуклеотидов
Table 1. Specific oligonucleotides sequences used in PCR assay
Условия реакции: 15 мин при 95ºС, 30 циклов (95ºС 30 с, 60ºС 90 с, 72ºС 60 с), финальная стадия элонгации 72ºС 10 мин [19].
Продукты реакции детектировали методом электрофореза в 1,2% агарозном геле на трис-борат-ЭДТА буфере с окрашиванием бромистым этидием. Детекцию производили с помощью системы «BioRad Gel-detection» («Bio-Rad Laboratories, Inc.»).
СТАТИСТИЧЕСКИЙ АНАЛИЗ
Анализ статистической достоверности различий представленности генов K. рneumoniae, связанных с вирулентностью, между группами изолятов проводили с помощью программного обеспечения «Statistica» («Statsoft») методом критерия согласия Пирсона (χ2) [20].
РЕЗУЛЬТАТЫ
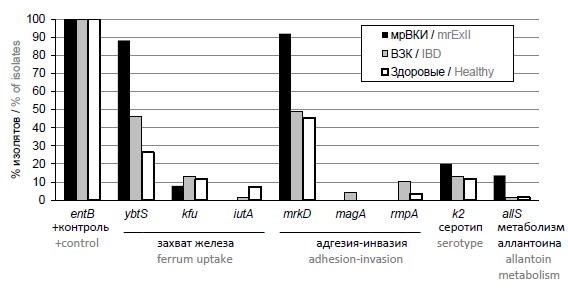
Результаты исследования наличия генов-мишеней у выборки из 69 изолятов от больных ВЗК, 68 изолята из кала «практически» здоровых людей и 25 изолятов от пациентов с ВКИ приведены на рисунке.
Доля изолятов, выделенных из кала «практически» здоровых людей, больных ВЗК и ВКИ, содержащих потенциально опасные гены K. рneumoniae.Ген entB использовался как положительный контроль прохождения реакции и выявлялся во всех исследуемых образцах.
Статистические различия между группами изолятов, выделенных от больных с ВЗК, ВКИ, обусловленными K. pneumoniae и группой изолятов, выделенных от «практически» здоровых лиц, представлены в табл. 2.
Таблица 2. Результаты статистического анализа данных методом критерия согласия Пирсона (χ2)
Table 2. Statistical evaluation of PCR assay results by Pearson's χ2 test
Примечание. Для гена kfu не выявлено статистически значимых отличий между исследуемыми группами.
Note. For kfu gene no statistical difference was found between the studied groups.
Ген сидерофора ybtS обнаруживался в изучаемых группах с достоверно различной частотой — у 88% изолятов мрВКИ, у 46,4% ВЗК и ещё реже у изолятов от «практически» здоровых — 26,5%. Отвечающий за транспорт железа ген kfu выявлялся во всех исследуемых группах без статистически значимых отличий (8–13% изолятов). Сидерофор iutA не выявлен у мрВКИ и найден всего у 1,4% ВЗК, в то же время обнаруживается у 7,4% здоровых изолятов из кала.
Ген mrkD, кодирующий фимбриальный адгезин 3-го типа, обеспечивающий прикрепление клебсиелл к целому ряду тканей человека, выявлен у ~49,3% изолятов ВЗК, у 45,5% здоровых и у 92% изолятов ВКИ.
Ген k2, ассоциированный с К2-серотипом бактериальной капсулы, встречался во всех исследуемых группах изолятов из кала — как здоровых, так и ВЗК и мрВКИ, без статистических различий.
Ген, связанный с гипермукоидным фенотипом K. pneumoniae (rmpA), не обнаружен во внекишечных изолятах, а среди кишечных чаще встречаются у изолятов, выделенных из ВЗК (10,1%). Хромосомный ген magA, ассоциированный с гипермукоидным фенотипом, встречается только у ВЗК-изолятов (4,3% из выборки).
Интересно, что ген allS достоверно чаще (χ2= 4,9; р = 0,032) встречался в группе внекишечных изолятов (13,8% в сравнении с 1,4–1,6 у изолятов, выделенных из кала).
ОБСУЖДЕНИЕ
Из 4 изученных генов сидерофоров только ybtS показал значимые отличия для исследуемых групп. Он достоверно чаще встречался у изолятов, выделенных от больных с ВЗК (р = 0,024) и мрВКИ (p < 0,001), в сравнении с изолятами, выделенными у «практически» здоровых людей. Основная функция сидерофоров заключается в переводе железа, связанного с белками или водонерастворимыми соединениями, в доступную для микроорганизмов ионную форму Fe3+. Большинство аэробных и факультативно-анаэробных микроорганизмов синтезируют хотя бы один сидерофор. В ряде исследований также выявлена связь сидерофоров с вирулентностью микроорганизмов [13].
Ген гипермукоидного фенотипа rmpA достоверно (р = 0,038) чаще встречался у изолятов, выделенных из кала пациентов с ВЗК, отличая эту группу как от группы обследованных «практически» здоровых, так и от пациентов с мрВКИ. Действительно, инвазивные способности hvKp считаются ассоциированными с гипермукоидностью — способностью формировать мукоидную нить повышенной длины (более 5 мм) и вязкости. Ген rmpA регулирует синтез внеклеточных полисахаридов, а ген magA отвечает за полимеризацию капсулы [21, 22]. Обнаружение этих генов только у бактерий, изолированных из кала больных, может быть связано с тем, что гипермукоидный фенотип предоставляет преимущества в случае колонизации на слизистых поверхностях. Ряд исследователей предполагают, что колонизация кишечника — это обязательная первичная стадия инфицирования организма, даже если инфекция проявляется в иных локализациях [23][24]. С кишечной колонизацией связывают также предполагаемые пути эпидемического распространения hvKp [25]. Есть сообщения о более частой встречаемости гена rmpA у кишечных изолятов в сравнении с внекишечными (в том числе из мочи и мазков слизистой оболочки зева) [26]. С другой стороны, имеются исследования, в которых показано наличие связи гена rmpA именно с ВКИ и системной инфекцией [27].
При анализе генов, чаще встречающихся у мультирезистентных штаммов от больных с мрВКИ, выявлены 3 гена, статистически чаще встречающиеся у изолятов данной группы больных в сравнении с «практически» здоровыми: ybtS (p < 0,001), mrkD (р < 0,001) и allS (p < 0,05). Об ассоциированности сидерофоров и генов метаболизма аллантоина с мультирезистентностью гипермукоидных клебсиелл, выделенных в России, сообщалось ранее [28][29].
Ген mrkD [30] функционирует как адгезин 3-го типа, участвуя в образовании биоплёнки на покрытых коллагеном поверхностях, в том числе внеклеточном матриксе клеток бронхиального эпителия. Показана его ассоциация с госпитальными инфекциями, в особенности верхних дыхательных путей [31].
Ген allS (регулирующий метаболизм аллантоина), который достоверно чаще (р = 0,032) встречался в группе внекишечных изолятов, по данным литературы, также более распространён во внекишечных штаммах K. pneumoniae, чем в остальных [32–34]. Он входит в состав оперона, отвечающего за метаболизм аллантоина. Продукты этих генов обеспечивают усвоение азота из экзогенного аллантоина, продукта катаболизма пуринов. Этот оперон обнаруживается и у других инфекционных представителей бактерий семейства Enterobacteriaceae (Salmonella typhimurium [35], Escherichia coli [36]). Генетический анализ данного оперона позволяетпредположить, что он может являться «островком патогенности», мобильным вирулентным элементом генома. Непосредственная функция данных генов в патогенезе не ясна, предполагается, что они важны при распространении инфекции из желудочно-кишечного тракта в печень и иные внекишечные локализации [34].
ЗАКЛЮЧЕНИЕ
Применение метода мультиплексной ПЦР для анализа генов, потенциально отвечающих за вирулентность и патогенность штаммов K. pneumoniae, показало ассоциацию 4 из 8 исследованных генов, связанных с болезненными состояниями пациентов, которые, по-видимому, обеспечивают преимущество перед другими видами микроорганизмов микробиоты человека и могут влиять на развитие патологического процесса. Это может иметь значение как для врачей-клиницистов, так и для врачей-эпидемиологов.
Ген сидерофора ybtS достоверно чаще встречался у изолятов, полученных из биологического материала от больных как ВЗК, так и с мрВКИ. У изолятов, выделенных из кала у пациентов с ВЗК, также достоверно чаще представлен ген гипермукоидного фенотипа rmpA. У внекишечных изолятов наиболее достоверно представлены 2 гена из изученных: адгезин 3-го типа mrkD и ген, отвечающий за метаболизм аллантоина, allS.
Таким образом, при изучении наличия генов, предположительно ассоциированных с патогенностью, у изолятов K. pneumoniae, выделенных из микробиоты больных и «практически» здоровых людей с применением метода мультиплексной ПЦР, получены достоверные результаты, показывающие, что наличие у изолятов K. pneumoniae генов ybtS и rmpA может быть ассоциировано с ВЗК, а наличие у K. pneumoniae генов ybtS, mrkD и allS — с возникновением внекишечных заболеваний, в том числе сепсиса.
1. Патент на изобретение № 2019128097 от 06.09.2019.
About the authors
Galina V. Pay
Centre for Strategic Planning and Management of Biomedical Health Risk of the Federal Medical Biological Agency
Email: tmaniya@cspmz.ru
ORCID iD: 0000-0001-7086-0899
Cand. Sci. (Med.), senior researcher, Laboratory of microbiology and parasitology
Россия, MoscowDaria V. Rakitina
Centre for Strategic Planning and Management of Biomedical Health Risk of the Federal Medical Biological Agency
Email: tmaniya@cspmz.ru
ORCID iD: 0000-0003-3554-7690
Cand. Sci. (Biol.), senior researcher, Laboratory of microbiology and parasitology
Россия, MoscowMarina A. Sukhina
Centre for Strategic Planning and Management of Biomedical Health Risk of the Federal Medical Biological Agency; State Scientific Center of Coloproctology
Email: tmaniya@cspmz.ru
ORCID iD: 0000-0003-4795-0751
Cand. Sci. (Biol.), senior researcher, Laboratory of microbiology and parasitology
Россия, Moscow; MoscowSergey M. Yudin
Centre for Strategic Planning and Management of Biomedical Health Risk of the Federal Medical Biological Agency
Email: tmaniya@cspmz.ru
ORCID iD: 0000-0002-7942-8004
D. Sci. (Med.), Professor, Director
Россия, MoscowValentin V. Makarov
Centre for Strategic Planning and Management of Biomedical Health Risk of the Federal Medical Biological Agency
Email: tmaniya@cspmz.ru
Scopus Author ID: 56273845200
Cand. Sci. (Biol.), Head, Department for analysis and forecasting medical and biological risks
Россия, MoscowTamari R. Maniya
Centre for Strategic Planning and Management of Biomedical Health Risk of the Federal Medical Biological Agency
Author for correspondence.
Email: tmaniya@cspmz.ru
ORCID iD: 0000-0002-6295-661X
researcher, Laboratory of microbiology and parasitology
Россия, MoscowAngelika V. Zagaynova
Centre for Strategic Planning and Management of Biomedical Health Risk of the Federal Medical Biological Agency
Email: tmaniya@cspmz.ru
ORCID iD: 0000-0003-4772-9686
Cand. Sci. (Biol.), Head, Laboratory of microbiology and parasitology
Россия, MoscowReferences
- Bagley S.T. Habitat association of Klebsiella species. Infect. Control. 1985; 6(2): 52-8. https://doi.org/10.1017/S0195941700062603
- Jarvis W.R., Munn V.P., Highsmith A.K., Culver D.H., Hughes J.M. The epidemiology of nosocomial infections caused by Klebsiella pneumoniae. Infect. Control. 1985; 6(2): 68-74. https://doi.org/10.1017/s0195941700062639
- Marchaim D., Chopra T., Pogue J.M., Perez F., Hujer A.M., Rudin S., et al. Outbreak of colistin‐resistant, carbapenem‐ resistant Klebsiella pneumoniae in metropolitan Detroit, Michigan. Antimicrob. Agents Chemother. 2011; 55(2): 593-9. https://doi.org/10.1128/AAC.01020-10
- Komisarova E.V., Volozhantsev N.V. Hypervirulent Klebsiella pneumonia: a new infectious threat. 2019; 17(3): 81-9. https://doi.org/10.20953/1729-9225-2019-3-81-89
- Khaertynov K.S., Anokhin V.A., Davidyuk Y.N., Nicolaeva I.V., Khalioullina S.V., Semyenova D.R., et al. Case of meningitis in a neonate caused by an extended-spectrum-beta-lactamaseproducing strain of hypervirulent Klebsiella pneumoniae. Front. Microbiol. 2017; 8: 1576. https://doi.org/10.3389/fmicb.2017.01576
- Frazee B.W., Hansen S., Lambert L. Invasive infection with hypermucoviscous Klebsiella pneumoniae: multiple cases presenting to a single emergency department in the United States. Ann. Emerg. Med. 2009; 53(5): 639‐42. https://doi.org/10.1016/j.annemergmed.2008.11.007
- McCabe R., Lambert L., Frazee B. Invasive Klebsiella pneumoniae infections, California, USA. Emerg. Infect. Dis. 2010; 16(9): 1490-1. https://doi.org/10.3201/eid1609.100386
- Kaur C.P., Vadivelu J., Chandramathi S. Impact of Klebsiella pneumoniae in lower gastrointestinal tract diseases. J. Dig. Dis. 2018; 19(5): 262-71. https://doi.org/10.1111/1751-2980.12595
- Atarashi K., Suda W., Luo C., Kawaguchi T., Motoo I., Narushima S., et al. Ectopic colonization of oral bacteria in the intestine drives TH1 cell induction and inflammation. Science. 2017; 358(6361): 359-65. https://doi.org/10.1126/science.aan4526
- Choby J.E., Howard‐Anderson J., Weiss D.S. Hypervirulent Klebsiella pneumoniae - clinical and molecular perspectives (Review). J. Intern. Med. 2020; 287(3): 283-300. https://doi.org/10.1111/joim.13007
- Jian-Li W., Yuan-Yuan S., Shou-Yu G., Fei-Fei D., Jia-Yu Y., Xue-Hua W., et al. Serotype and virulence genes of Klebsiella pneumoniae isolated from mink and its pathogenesis in mice and mink. Sci. Rep. 2017; 7(1): 17291. https://doi.org/10.1038/s41598-017-17681-8
- Turton J.F., Payne Z., Coward A., Hopkins K.L., Turton J.A., Doumith M., et al. Virulence genes in isolates of Klebsiella pneumoniae from the UK during 2016, including among carbapenemase gene-positive hypervirulent K1-ST23 and 'nonhypervirulent' types ST147, ST15 and ST383. J. Med. Microbiol. 2018; 67(1): 118-28. https://doi.org/10.1099/jmm.0.000653
- Holden V.I., Breen P., Houle S., Dozois C.M., Bachman M.A. Klebsiella pneumoniae siderophores induce inflammation, bacterial dissemination, and HIF-1α stabilization during pneumonia. mBio. 2016; 7(5): e01397-16. https://doi.org/10.1128/mBio.01397-16
- Лазарева И.В., Старкова П.С., Агеевец В.А., Волкова М.О., Лебедева М.С., Навацкая А.С. и др. Оценка распространения ректального носительства генов вирулентности и карбапенемаз у пациентов, поступивших на плановую госпитализацию. Антибиотики и химиотерапия. 2018; 63(11-12): 18-23.
- Fang C., Chuang Y., Shun C., Chang S., Wang J. A novel virulence gene in Klebsiella pneumoniae strains causing primary liver abscess and septic metastatic complications. J. Exp. Med. 2004; 199(5): 697-705. https://doi.org/10.1084/jem.20030857
- Lee C.R., Lee J.H., Park K.S., Jeon J.H., Kim Y.B., Cha C.J., et al. Antimicrobial resistance of hypervirulent Klebsiella pneumoniae: еpidemiology, hypervirulence-associated determinants, and resistance mechanisms. Front. Cell Infect. Microbiol. 2017; 7: 483. https://doi.org/10.3389/fcimb.2017.00483
- Russo T.A., Olson R., Fang C.T., Stoesser N., Miller M., MacDonald U., et al. Identification of biomarkers for differentiation of hypervirulent Klebsiella pneumoniae from classical K. pneumoniae. J. Clin. Microbiol. 2018; 56(9): e00776-18. https://doi.org/10.1128/JCM.00776-18
- Russo T.A., Olson R., Macdonald U., Metzger D., Maltese L.M., Drake E.J., et al. Aerobactin mediates virulence and accounts for increased siderophore production under iron-limiting conditions by hypervirulent (hypermucoviscous) Klebsiella pneumoniae. Infect. Immun. 2014; 82(6): 2356-67. https://doi.org/10.1128/IAI.01667-13
- Compain F., Babosan A., Brisse S., Genel N., Audo J., Ailloud F., et al. Multiplex PCR for detection of seven virulence factors and K1/K2 capsular serotypes of Klebsiella pneumoniae. J. Clin. Microbiol. 2014; 52(12): 4377-80. https://doi.org/10.1128/JCM.02316-14
- Медицинская статистика. Анализ произвольных таблиц сопряженности с использованием критерия хи-квадрат (онлайн калькулятор). Available at: https://medstatistic.ru/calculators/calchit.html
- Shon A.S., Bajwa R.P., Russo T.A. Hypervirulent (hypermucoviscous) Klebsiella pneumoniae: a new and dangerous breed. Virulence. 2013; 4(2): 107-18. https://doi.org/10.4161/viru.22718
- Lee H.C., Chuang Y.C., Yu W.L., Lee N.Y., Chang C.M., Ko N.Y., et al. Clinical implications of hypermucoviscosity phenotype in Klebsiella pneumoniae isolates: association with invasive syndrome in patients with community-acquired bacteraemia. J. Intern. Med. 2006; 259(6): 606-14. https://doi.org/10.1111/j.1365-2796.2006.01641.x
- Gorrie C.L., Mirceta M., Wick R.R., Edwards D.J., Thomson N.R., Strugnell R.A., et al. Gastrointestinal carriage is a major reservoir of Klebsiella pneumoniae infection in intensive care patients. Clin. Infect. Dis. 2017; 65(2): 208-15. https://doi.org/10.1093/cid/cix270
- Martin R.M., Cao J., Brisse S., Passet V., Wu W., Zhao L., et al. Molecular epidemiology of colonizing and infecting isolates of Klebsiella pneumoniae. mSphere. 2016; 1(5): e00261. https://doi.org/10.1128/mSphere.00261-16
- Fung C.P., Lin Y.T., Lin J.C., Chen T.L., Yeh K.M., Chang F.Y., et al. Klebsiella pneumoniae in gastrointestinal tract and pyogenic liver abscess. Emerg. Infect. Dis. 2012; 18(8): 1322-5. https://doi.org/10.3201/eid1808.111053
- Семенова Д.Р., Николаева И.В., Фиалкина С.В., Хаертынов Х.С., Анохин В.А., Валиуллина И.Р. Частота колонизации «гипервирулентными» штаммами Klebsiella pneumoniae новорожденных и грудных детей с внебольничной и нозокомиальной клебсиеллезной инфекцией. Российский вестник перинатологии и педиатрии. 2020; 65(5): 158-63. https://doi.org/10.21508/1027-4065-2020-65-5-158-163
- Cheng H.Y., Chen Y.S., Wu C.Y., Chang H.Y., Lai Y.C., Peng H.L. RmpA regulation of capsular polysaccharide biosynthesis in Klebsiella pneumoniae CG43. J. Bacteriol. 2010; 192(12): 3144-58. https://doi.org/10.1128/JB.00031-10
- Фурсова Н.К., Асташкин Е.И., Новикова Т.С., Федюкина Г.Н., Ершова О.Н. Мультирезистентные Klebsiella pneumoniae, вызвавшие тяжёлые формы инфекций у пациентов в отделении нейрореанимации. В кн.: Сборник материалов Всероссийская научно-практическая конференция с международным участием «Молекулярная диагностика и биобезопасность - 2020». М.; 2020: 79-80. https://doi.org/10.36233/978-5-9900432-9-9-79
- Kislichkina A.A., Lev A.I., Komisarova E.V., Fursova N.K., Myakinina V.P., Mukhina T.N., et al. Genome sequencing and comparative analysis of three hypermucoviscous Klebsiella pneumoniae strains isolated in Russia. Pathog. Dis. Vol. 2017; 75(4). https://doi.org/10.1093/femspd/ftx024
- Jagnow J., Clegg S. Klebsiella pneumoniae MrkD-mediated biofilm formation on extracellular matrix- and collagen-coated surfaces. Microbiology. 2003; 149(Pt. 9): 2397-405. https://doi.org/10.1099/mic.0.26434-0
- Murphy C.N., Clegg S. Klebsiella pneumoniae and type 3 fimbriae: nosocomial infection, regulation and biofilm formation. Future Microbiol. 2012; 7(8): 991-1002. https://doi.org/10.2217/fmb.12.74
- Yu W.L., Ko W.C., Cheng K.C., Lee C.C., Lai C.C., ChuangY.C. Comparison of prevalence of virulence factors for Klebsiella pneumoniae liver abscesses between isolates with capsular K1/ K2 and non-K1/K2 serotypes. Diagn. Microbiol. Infect. Dis. 2008; 62(2): 1-6. https://doi.org/10.1016/j.diagmicrobio.2008.04.007
- Brisse S., Fevre C., Passet V., Issenhuth-Jeanjean S., Tournebize R., Diancourt L., et al. Virulent clones of Klebsiella pneumoniae: identification and evolutionary scenario based on genomic and phenotypic characterization. PLoS One. 2009; 4(3): e4982. https://doi.org/10.1371/journal.pone.0004982
- Chou H.C., Lee C.Z., Ma L.C., Fang C.T., Chang S.C., Wang J.T. Isolation of a chromosomal region of Klebsiella pneumoniae associated with allantoin metabolism and liver infection. Infect. Immun. 2004; 72(7): 3783-92. https://doi.org/10.1128/IAI.72.7.3783-3792.2004
- Garaizar J., Porwollik S., Echeita A., Rementeria A., Herrera S., Wong R.M., et al. DNA microarray-based typing of an atypical monophasic Salmonella enterica serovar. J. Clin. Microbiol. 2002; 40(6): 2074-8. https://doi.org/10.1128/JCM.40.6.2074-2078.2002
- Rintoul M.R., Cusa E., Baldoma L., Badia J., Reitzer L., Aguilar J. Regulation of the Escherichia coli allantoin regulon: coordinated function of the repressor AllR and the activator AllS. J. Mol. Biol. 2002; 324(4): 599-610. https://doi.org/10.1016/s0022-2836(02)01134-8
Supplementary files