Итоги многоцентрового мониторинга возбудителя инвазивного листериоза в мегаполисе
- Авторы: Воронина О.Л.1, Рыжова Н.Н.1, Кунда М.С.1, Аксенова Е.И.1, Карпова Т.И.1, Мелкумян А.Р.2, Климова Е.А.3, Кареткина Г.Н.3, Посуховский Е.А.3, Груздева О.А.4, Тартаковский И.С.1
-
Учреждения:
- Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Гамалеи Н.Ф.
- Городская клиническая больница имени Ф.И. Иноземцева
- Московский государственный медико-стоматологический университет имени А.И. Евдокимова
- Российская медицинская академия непрерывного профессионального образования
- Выпуск: Том 100, № 3 (2023)
- Страницы: 143-154
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/7321
- DOI: https://doi.org/10.36233/0372-9311-393
- EDN: https://elibrary.ru/nzfizb
- ID: 7321
Цитировать
Аннотация
Введение. Инвазивный листериоз — редкое заболевание, опасное для групп риска и часто приводящее к летальному исходу. Возбудитель — Listeria monocytogenes — убиквитарный сапрофит, ставший актуальным патогеном пищевого происхождения с развитием индустрии полуфабрикатов и продуктов быстрого приготовления.
Цель работы — характеристика изолятов L. monocytogenes в Московском регионе и определение возможных причин восприимчивости к инфекции.
Материалы и методы. Проведён многоцентровой мониторинг L. monocytogenes в Московском регионе с применением бактериологических и геномных методов в описании возбудителя, сбора анамнеза и подробного анализа эпикриза пациентов.
Результаты. В когортах перинатальный листериоз (ПЛ) и менингит-септицемия (МС) инвазивный листериоз встречался во все месяцы года с небольшими подъёмами в марте–апреле и июле–ноябре. В период пандемии COVID-19 в группе МС минимальный возраст пациентов снизился до 31 года, доля умерших выросла в 1,57 раза по сравнению с 2018–2019 гг. В период пандемии наблюдали рост разнообразия генотипов L. monocytogenes и изменения в спектре генотипов возбудителя на этапах пандемии.
За всё время мониторинга охарактеризовали 73 клинических изолята L. monocytogenes, относящихся к 24 генотипам. Семь генотипов принадлежали к первой филогенетической линии (PLI), 14 генотипов — к PLII. В когорте ПЛ была самая высокая доля генотипов PLI (52%). В когорте МС в группе мужчин отметили наибольшее разнообразие генотипов, 6 из которых совпадали с генотипами пищевых изолятов. В выборке изолятов описали 12 новых профилей генов интерналинов. Полногеномное секвенирование показало наличие плазмид в 9 из 58 геномов клинических изолятов. Сопоставление коровых геномов выявило эпидемическую связь между изолятами одного генотипа в отношении ST4, ST21, ST425.
Заключение. Проведённое исследование предоставило подробное описание разнообразия и вирулентности L. monocytogenes, циркулирующих в Московском регионе, что будет способствовать своевременной диагностике и лечению инвазивного листериоза.
Ключевые слова
Полный текст
Введение
Инвазивный листериоз (ИЛ), вызываемый убиквитарным сапрофитом Listeria monocytogenes, является редким заболеванием с высокой долей летальных исходов1. К группам риска по ИЛ относят беременных, эмбрионы, плоды и новорождённых (когорта перинатального листериоза; ПЛ), а также пожилых и иммунокомпрометированных лиц (когорта менингит и/или септицемия; МС). По данным статистики, в Москве в 2020 и 2021 гг. зарегистрировали 18 и 21 случай листериоза соответственно2. Однако, учитывая тяжесть основного диагноза групп риска, мы не можем исключить, что случаи сопутствующего ИЛ являются недоучтёнными из-за отсутствия регистрации результатов лабораторных исследований. Одобренное в конце 2018 г. многоцентровое исследование в Московском регионе имело целью выявление и анализ клинических изолятов L. monocytogenes, выделенных от госпитализированных пациентов. Начиная проект, мы опирались на данные по разнообразию генотипов клинических изолятов листерий в европейской части России, полученные как в ретроспективном исследовании коллекционных изолятов 1971–1999 гг. [1], так и в анализе изолятов 2015–2017 гг. [2]. Поскольку с развитием индустрии полуфабрикатов и продуктов быстрого приготовления L. monocytogenes стала актуальным патогеном пищевого происхождения, в исследование были включены также изоляты, выделенные из продуктов питания на объектах, контролируемых департаментом Роспотребнадзора по Москве. Несмотря на малую долю положительных по листерии образцов пищевой продукции (0,6%) среди 6883, прошедших лабораторный контроль в 2020 г. в Москве3, разнообразие источников L. monocytogenes достаточно широко: мясо, птица, рыба, молочные продукты [3–6]. Не следует забывать и об окружающей среде как источнике листерий, что показали исследования природных парков и водоёмов Центрального федерального округа [7], а также водных объектов вблизи животноводческих предприятий в Вологодской области [8]. Пандемия COVID-19 повлекла за собой появление новой группы риска — пациентов, перенёсших COVID-19, для которых вероятны как развитие листериозного сепсиса и/или менингита/менингоэнцефалита, так и высокий риск неблагоприятного исхода [9]. Микробиологические исследования в ходе мониторинга призваны были прежде всего оценить изменение самого возбудителя инфекции с привлечением молекулярно-генетических методов, используемых центрами по контролю и профилактике заболеваний США и Европы, а также вошедших в арсенал научно-исследовательских центров нашей страны.
Материалы и методы
За время мониторинга (с ноября 2018 г. по февраль 2023 г.) проанализировали 73 клинических изолята L. monocytogenes, выделенные в 9 стационарах Москвы, и 44 пищевых изолята, предоставленные филиалом ФБУЗ «Центр гигиены и эпидемиологии в городе Москве» в Центральном административном округе г. Москвы. В группу наблюдения и изучения эпикриза вошли госпитализированные пациенты, из образцов которых были выделены изоляты листерий, предоставленные для молекулярно-генетического исследования. Исследование проводили при добровольном информированном согласии пациентов. Протокол исследования одобрен Комитетом по биомедицинской этике НИЦЭМ им. Н.Ф. Гамалеи (протокол № 14 от 04.07.2018).
Культивирование листерий, анализ с помощью молекулярно-генетических методов, включающих MLST (MultiLocus Sequence Typing), MvLST (Multi-virulent-locus sequence typing) и полногеномное секвенирование, сборку и аннотацию геномов выполняли, как описано ранее [10].
Анализ аллелей MLST и аллельных профилей (ST, Sequence Type) проводили с помощью ресурсов Bacterial Isolate Genome Sequence Database for L. monocytogenes (BIGSdb-Lm4). Проанализированные изоляты и новые аллельные профили депонировали в базе данных сайта, ID: 49370–49375; 75929–75933; 76308–76312; 76385–76389; 77384–78379; 78656–78660; 78713–78717; 82478–82494; 98277–98278; 98280–98297; 100872–100876; 102088–102092.
Аллели MvLST определяли, используя в качестве референсов ранее опубликованные последовательности. Новые варианты аллелей inlA, inlB, inlE зарегистрировали в GenBank (Accession Numbers: MW538938, MW965279; MW538939, MZ486423; OM240824, OQ606809). Для новых профилей интерналинов, IP (Internalin genes (inlA, inlB, inlC, inlE) Profile) продолжали ранее установленную нумерацию [11].
Секвенированные геномы депонировали в GenBank в биопроект PRJNA605697. Выявленные плазмиды зарегистрировали под номерами MZ869810, MZ869811, OM867528–OM867530, OP752358–OP752360, OP921773, OP921774.
Анализ корового генома проводили согласно схеме MLST для 1748 локусов [12] с помощью открытой биоинформационной платформы4. Факторы вирулентности определяли, используя Virulence Factors Database5 и VF analyzer6 [13], а также BIGSdb-Lm database. Для анализа резистома использовали схему «Antibiotic Resistance» BIGSdb-Lm database, а также данные Comprehensive Antibiotic Resistance Database7 [14]. Поиск мобильных элементов осуществляли с помощью программы MobileElementFinder8. Для определения принадлежности плазмид к RepA филогенетическим группам проводили сравнение последовательностей генов RepA, кодирующих белок инициации репликации [15]. Для выравнивания плазмид и построения деревьев использовали «CLC Genomics Workbench v. 21.0.1» с плагином «Whole Genome Alignment v. 21.0», привлекая в качестве референсов гомологичные плазмиды, представленные в GenBank (CP015985, MZ089999, MZ147615, MZ065170, KU513859).
Результаты
Общая характеристика случаев инвазивного листериоза в выборке
Мониторинг возбудителя ИЛ в Московском регионе продолжался более 4 лет. Он охватывал как период до пандемии COVID-19, так и время пандемии. В соответствии с основными группами риска выявленные случаи ИЛ распределили по двум основным когортам: ПЛ и МС. Доля ПЛ в выборке за все время наблюдения составила 37%, несколько снизившись в период пандемии: с 42 до 35%. Доля когорты МС подросла в пандемию с 58 до 65%, в том числе за счёт случаев ИЛ у лиц более молодого возраста, чем ранее выявленный порог в 59 лет [3].
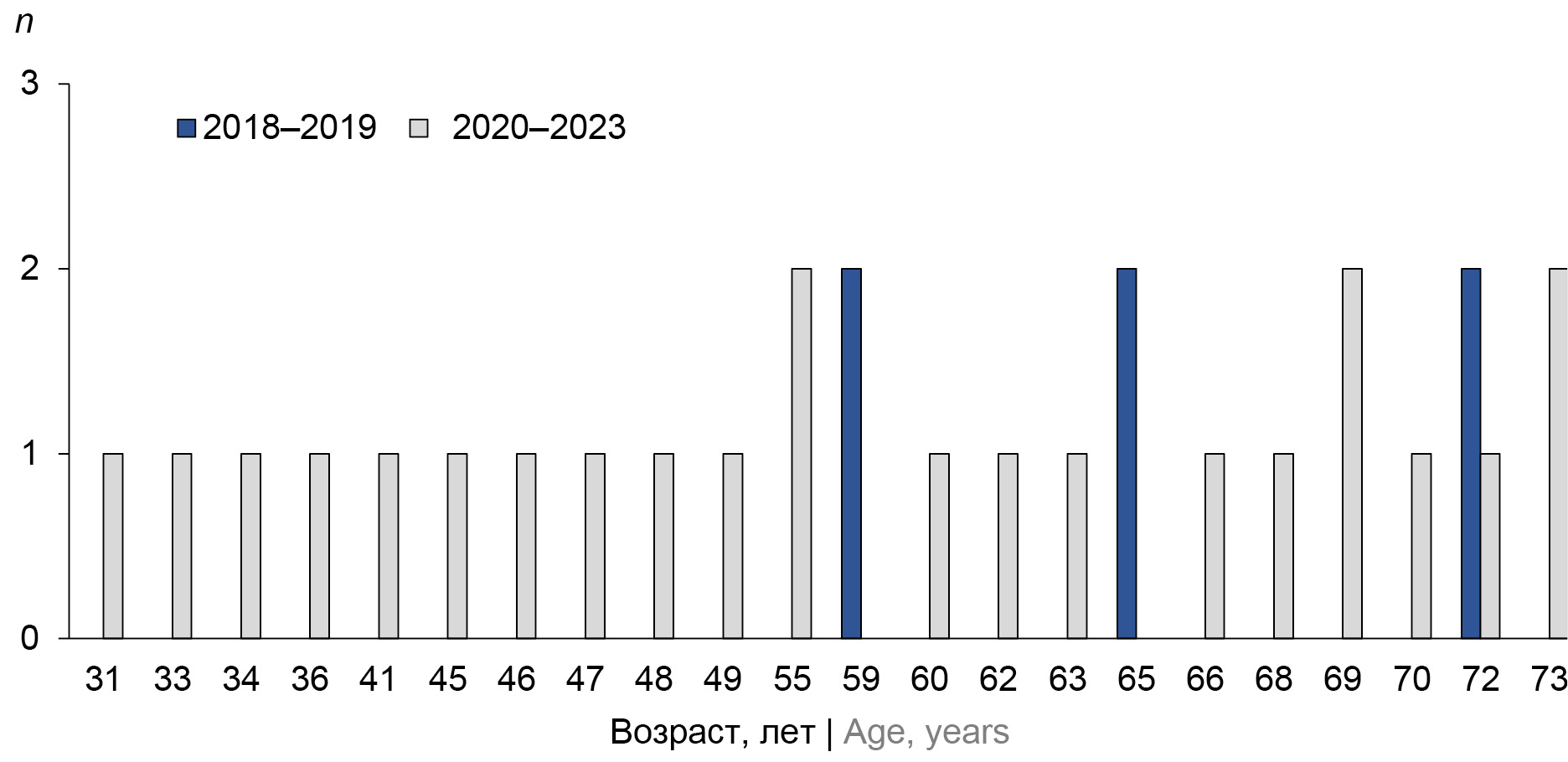
Минимальный возраст заболевших в этой когорте снизился до 31 года (рис. 1). Доля пациентов в возрасте до 59 лет составила 34%. При этом доля умерших в группе наблюдения возросла в 1,57 раза (с 36% до пандемии до 57% в период COVID-19).
Рис. 1. Возраст пациентов с ИЛ до пандемии и в период пандемии COVID-19 в когорте МС. / Fig. 1. Age of patients with IL before and during the COVID-19 pandemic in the MS cohort.
Количество накопленных за всё время наблюдения случаев ИЛ позволило оценить распределение заболевших по месяцам года в соответствии с датой постановки диагноза (рис. 2). ИЛ в обеих когортах встречается во все месяцы года с небольшими подъёмами в марте–апреле и июле–ноябре.
Рис. 2. Распределение выявленных случаев ИЛ по месяцам года. / Fig. 2. Distribution of detected IL cases by months. MS — meningitis-septicemia; PL — perinatal listeriosis.
Молекулярно-генетический анализ выделенных изолятов L. monocytogenes
Разнообразие генотипов в периоды наблюдения. 73 клинических изолята L. monocytogenes, выделенных за время мониторинга, относились к 24 генотипам. К более вирулентной PLI принадлежали 7 генотипов (ST1, ST4, ST5, ST6, ST194, ST204, ST219); к PLII — 14 генотипов (ST8, ST7, ST14, ST17, ST20, ST21, ST29, ST37, ST101, ST121, ST155, ST241, ST391, ST425, ST451, ST1365, ST2096).
Пандемия COVID-19 разделила время мониторинга возбудителя ИЛ на два периода: 2018–2019 и 2020–2023 гг. Последний, в свою очередь, можно подразделить на три этапа в соответствии с генетическими характеристиками циркулировавших вариантов SARS-CoV-2:
- I этап (март 2020 г. – май 2021 г.) — циркуляция SARS-CoV-2 разных панголиний [16];
- II этап (июнь 2021 г. – январь 2022 г.) — преобладание варианта Delta [17];
- III этап (февраль 2022 г. – настоящее время) — преобладание варианта Omicron [18].
Со сменой этапов пандемии мы наблюдали изменения в спектре генотипов клинических изолятов L. monocytogenes (рис. 3). Доминировавшие до пандемии аутохтонный ST7 (PLII) и привнесённый ST6 (PLI) [3, 10] на I этапе пандемии отмечены в единичных случаях. Выявленные ST4, ST219 (PLI) и ST391 (PLII) были новыми для России генотипами. L. monocytogenes ST21, ST37, ST425, выделенные на I этапе пандемии, ранее не регистрировали при ИЛ у людей. На II этапе детектировали ещё 2 новых для клинических изолятов генотипа: ST8 (PLII) и ST194 (PLI). На III этапе выявили 4 генотипа L. monocytogenes, ранее не зарегистрированные в России: ST17, ST29, ST204, ST1365 — все PLII, а также ST121 (PLII), ранее не отмеченный у клинических изолятов.
Рис. 3. Разнообразие генотипов клинических изолятов L. monocytogenes до пандемии и на этапах пандемии COVID-19. I–III — периоды пандемии COVID-19; красными точками отмечены изоляты филогенетической линии I, новые генотипы для клинических изолятов L. monocytogenes обведены фиолетовой рамкой. Наличие плазмиды в геноме обозначено pl в овале. /Fig. 3. Diversity of genotypes of L. monocytogenes clinical isolates before and during the COVID-19 pandemic. I–III — periods of the COVID-19 pandemic; isolates of phylogenetic lineage I are marked with red dots; new genotypes for L. monocytogenes clinical isolates are framed in purple. The presence of a plasmid in the genome is marked by pl shown in an oval.
Разнообразие генотипов в группах наблюдения. Мы сравнили разнообразие генотипов изолятов L. monocytogenes в анализируемых когортах в течение всего времени мониторинга, подразделив когорту МС на 2 группы в соответствии с полом пациентов (МС_M — мужчины; МС_F — женщины). Как видно из рис. 4, во всех группах были выявлены изоляты ST4, ST6, ST7 и ST8. У изолятов группы МС_F было по 1 общему генотипу с изолятами групп ПЛ (ST21) и МС_M (ST425), тогда как между изолятами групп ПЛ и МС_M было 3 общих генотипа (ST1, ST194 и ST451). У изолятов группы МС_M выявлено наибольшее количество генотипов (17), коэффициент Шеннона составил 3,7. Изоляты ST37 встречались в группе МС_M чаще всего (21%) и не отмечены в других группах. Изоляты именно этого генотипа занимали 2-е место по частоте встречаемости (20,5%) в группе «пищевых изолятов» (рис. 5, а) и были выделены изо всех разновидностей продуктов (рис. 5, б).
Рис. 4. Разнообразие генотипов клинических изолятов L. monocytogenes в группах наблюдения. Розовая рамка — генотипы, общие для всех групп; синяя — общие для MC_М и ПЛ; фиолетовая — общие для MC_F и ПЛ; зелёная — общие для MC_М и MC_F. / Fig. 4. Diversity of genotypes of L. monocytogenes clinical isolates in study groups. MS_F — a group of women in the meningitis-septicemia cohort; MS_M — group of men in the meningitis-septicemia cohort; PL — perinatal listeriosis. The pink frame — genotypes common for all groups; the blue frame – genotypes common for MS_M and PL; the purple frame — genotypes common for MS_F and PL; the green frame — genotypes common for MS_M and MS_F.
Рис. 5. Изоляты L. monocytogenes, выделенные из продуктов питания. а — частота встречаемости L. monocytogenes разных генотипов в продуктах; б — распределение по категориям продуктов L. monocytogenes наиболее представленных генотипов. В красной рамке — наиболее представленные генотипы L. monocytogenes. / Fig. 5. L. monocytogenes isolates from food product. а — the frequency of occurrence of different L. monocytogenes genotypes in products; b — distribution of the most represented L. monocytogenes genotypes by product categories. The red frame shows the most represented L. monocytogenes genotypes.
В целом совпадение по генотипам с группой пищевых изолятов было выше в группе МС_M: совпадающих генотипов насчитывалось 6 (ST7, ST8, ST37, ST121, ST155, ST451), тогда как в группах ПЛ и МС_F только 2–3 генотипа совпадали с генотипами пищевых изолятов.
Совпавшие по генотипам пищевые и клинические изоляты требовали более пристального внимания. Прежде всего у них проверяли профиль генов интерналинов (IP). Отметим, что у пищевых изолятов наиболее часто встречающихся генотипов (ST121, ST37, ST7) IP были идентичными с профилями клинических изолятов. Однако для изолятов других генотипов были выявлены новые профили генов IP.
Новые профили IP. 12 новых профилей генов IP (56, 58–62, 66–71) были зарегистрированы в настоящем исследовании в 2020–2023 гг. (таблица). Только один новый IP был выявлен у изолята PLI нового для России ST219, остальные — у изолятов PLII, относящихся как к новым генотипам (26, 391, 1365, 204, 17, 29), так и к генотипам, которые ранее выявляли у изолятов из других источников. Например, изолят ST21, выделенный в 1975 г. от козы с листериозом в Северо-Кавказском федеральном округе (BIGSdb-Lm ID 41525) [1], имел иной IP, чем клинические изоляты ST21, выделенные в 2020 г. IP отличаются аллелем локуса inlC. Клинические изоляты ST425 также отличались по IP от изолята, выделенного в 2009 г. из окружающей среды (из фекалий оленя Cervus elaphus, BIGSdb-Lm ID 5909 [19]): различия в аллелях inlB. Изоляты ST155 показали самые разнообразные IP. В многоцентровом исследовании до пандемии COVID-19 изоляты ST155 выделяли дважды: из продуктов (рыба) и от пациента. IP изолятов отличались аллелями локуса inlE. Изолят, выделенный в 2021 г. из рыбы, совпал по IP с клиническим изолятом, однако изолят 2022 г. также из рыбы вновь продемонстрировал новый IP, отличающийся по локусу inlE. Сравнив изоляты ST155 нашей выборки с изолятами, выделенными в Дальневосточном федеральном округе в 2006 г. от грызунов [20], мы обнаружили отличие в другом локусе — inlB. Изоляты ST451 и ST403 ранее выделяли в России [2], но не типировали IP, поэтому IP изолятов этих генотипов зарегистрировали впервые.
Новые профили интерналинов / New Internalin Profile
Изолят Isolate | BIGSdb-Lm ID | CC | ST | inlA | inlB | inlC | inlE | IP | PL | Источник Source |
GIMC2056:LmcEH-1 | 49374 | CC21 | 21 | 12 | 14 | 7 | 8 | 56 | II | Клиника | Clinical |
GIMC2060:LmcH24-1 | 75931 | CC11 | 451 | 22 | 23 | 6 | 6 | 58 | II | Клиника | Clinical |
GIMC2062:Lmc5157 | 75933 | CC26 | 26 | 7 | 14 | 6 | 6 | 59 | II | Пища | Food |
GIMC2073:LmcUH25 | 77384 | CC4 | 219 | 23 | 8 | 4 | 3 | 60 | I | Клиника | Clinical |
GIMC2077:LmcBH-1 | 78714 | CC89 | 391 | 15 | 14 | 17 | 6 | 61 | II | Клиника | Clinical |
GIMC2079:LmcUH26 | 78377 | CC90 | 425 | 13 | 24 | 13 | 8 | 62 | II | Клиника | Clinical |
GIMC2097:Lmc929 | 82487 | CC403 | 403 | 9 | 23 | 21 | 17 | 66 | II | Пища | Food |
GIMC2117:Lmc22984 | 98289 | CC155 | 155 | 10 | 14 | 19 | 18 | 67 | II | Пища | Food |
GIMC2120:LmcH51-1 | 98292 | ST1365 | 1365 | 22 | 20 | 6 | 9 | 68 | II | Клиника | Clinical |
GIMC2124:LmcUH35 | 98296 | CC204 | 204 | 7 | 13 | 18 | 6 | 69 | II | Клиника | Clinical |
GIMC2126:LmcH51-3 | 100872 | CC8 | 17 | 12 | 14 | 6 | 17 | 70 | II | Клиника, пища Clinical, Food |
GIMC2133:LmcZhH_1 | 102090 | CC29 | 29 | 9 | 14 | 19 | 6 | 71 | II | Клиника | Clinical |
Примечание. IP 57 (7; 13, 16, 6) идентифицирован С.А. Ермолаевой и Е.К. Псаревой (данные не опубликованы); IP 63–65 опубликованы в [4]; CC — Clonal Complex; ST — sequence type; IP — Internalin genes (inlA, inlB, inlC, inlE) profile; PL — phylogenetic lineage.
Note. IP 57 (7; 13, 16, 6) was identified by S. Ermolaeva and E. Psareva (the data have not been published); IP 63–65 were published in [4]; CC — clonal complex; ST — sequence type; IP — internalin gene (inlA, inlB, inlC, inlE) profile; PL — phylogenetic lineage.
Анализ геномов изолятов L. monocytogenes
Сопоставление коровых геномов (cgMLST). Наиболее полную картину об эпидемической взаимосвязи изолятов одного генотипа даёт сравнение коровых геномов (1748 локусов). Как видно из рис. 6, изоляты ST4, ST425, ST21, выделенные от разных пациентов, имели 0–5 отличий, что доказывает эпидемическую связь изолятов одного генотипа. Коровые геномы изолятов ST8, ST37 и ST451 различались 22–74 локусами, таким образом, для листерий этих генотипов характерны множественные источники инфекции.
Рис. 6. Геномные характеристики изолятов L. monocytogenes. а — количество локусов, различающих коровые геномы одного генотипа; б — плазмидом выборки изолятов. Оранжевая линия показывает допустимое количество отличий (7–10), позволяющих отнести изоляты к одной эпидемической вспышке. Виды листерий, обозначенные на древе гомологии: L.m — L. monocytogenes, L.w — L. welshimeri. Страны происхождения изолятов: IT — Италия; PL — Польша; RU — Россия. Источники изолятов: F — пища; E — окружающая среда; FPE — среда производства продуктов; CL — клинические изоляты. Обозначение цветными кругами: красный — клинические изоляты; голубой — пищевые изоляты; зеленый — изоляты из окружающей среды. / Fig. 6. Genomic characteristics of L. monocytogenes isolates. a — the number of loci differentiating core genomes of the same genotype; b — plasmidome of the isolate subset. The orange line indicates the permissible number of differences (7–10), based on which isolates can be assigned to the same epidemic outbreak. The Listeria species indicated on the phylogenetic tree: L.m — L. monocytogenes, L.w — L. welshimeri. Countries of origin of isolates: IT — Italy; PL — Poland; RU — Russia. Sources of isolates: F — food; E — environment; FPE — food production environment; CL — clinical isolates. Colored circles: red — clinical isolates; blue — food isolates; green — isolates from the environment.
Плазмиды в геномах L. monocytogenes. Плазмиды в геномах листерий, особенно в геномах клинических изолятов, встречаются достаточно редко [21]. Из 72 геномов Listeria spp., секвенированных в наших проектах (PRJNA658237, PRJNA326717, PRJNA326713, PRJNA605697), только в 11 (15%) были плазмиды: 1 — в геноме изолята L. welshimeri ST2331, выделенного на мясоперерабатывающем производстве [11]; 9 — в геномах клинических изолятов L. monocytogenes PLII (ST8, ST20, ST121, ST425); 1 — в геноме пищевого изолята L. monocytogenes ST121. Плазмиды различались по размеру и по группе репликации. К группе G1 RepA относились плазмиды изолятов L. welshimeri ST2331 (57530 bp) и L. monocytogenes ST20, ST425 (72763 bp); к группе G2 RepA — плазмиды изолятов ST121 (62207 bp) и ST8 (86632 bp). Гены устойчивости к кадмию присутствовали во всех плазмидах. Плазмида изолятов ST20, ST425 содержала гены устойчивости к другим тяжёлым металлам, оксидативному стрессу, изменениям pH и осмоса, плазмида ST121 — гены IV системы секреции, обеспечивающие конъюгативный трансфер. Самая большая плазмида изолятов ST8 включала все перечисленные гены.
Анализ факторов вирулентности в геномах изолятов выявленных генотипов. Поиск факторов вирулентности (VF) в геномах L. monocytogenes можно проводить с помощью Virulence Factors Database, включающей 45 факторов, или BIGSdb-Lm database с расширенным списком из 76 факторов, в том числе генов, входящих в острова патогенности LIPI-3 и LIPI-4. Большинство анонсированных в базах данных VF обнаружены в секвенированных геномах 21 генотипа обеих филогенетических линий: PLI — 7 ST; PLII — 14 ST. Из полного списка VF у всех представителей PLII отсутствовали ген aut_IVb (LMOF2365_RS00075), кодирующий фактор инвазии, а также гены LIPI-3 и LIPI-4. В геномах L. monocytogenes PLI не обнаружены гены aut (lmo1076, инвазия), tagB (lmo1088, биосинтез тейхоевой кислоты) и inlL (LMON_RS10535, интерналин). Среди представителей генотипов PLII ген inlL был только в геномах 6 ST из 14, vip (lmo0320, инвазия) — также в 6 из 14ST, inlG (lmo0262, интерналин) — в 11 из 14 ST. В геномах PLI ген inlG был обнаружен только у L. monocytogenes ST6. LIPI-3 и LIPI-4 были представлены не во всех геномах PLI. LIPI-3 (синтез листериолизина S) содержался в геномах изолятов ST1, ST6, ST4, ST219, тогда как у изолятов ST194, ST2, ST145 он отсутствовал. LIPI-4 (система транспорта сахаров, PTS) присутствовал только в геномах изолятов ST4, ST219 (CC4) и изолятов ST194 (СС315).
Гены антибиотикорезистентности выделены в отельную схему «Antibiotic Resistance» разработчиками BIGSdb-Lm database. Схема включает 25 локусов [12], представленных как генами хромосомы, так и генами, которые могут быть привнесены с помощью плазмид и транспозонов. Во всех проанализированных геномах обнаружили 5 генов: fosX (lmo1702), lin (lmo0919), mprF (lmo1695), sul (lmo0224) и norB (lmo2818), обеспечивающих устойчивость к фосфомицину, линкозамиду, катионным пептидам, сульфонамиду и хинолонам, по данным разработчиков. Выявленные гены имели генотип-специфичные аллели. Анализ последовательностей гена sul (lmo0224) показал, что они кодируют дигидроптероат синтазу, участвующую в биосинтезе фолатов и являющуюся мишенью для сульфонамида. Только наличие замен на участке 58–71 аминокислотных остатков (нумерация по E. coli), формирующих соединительную петлю между β-нитью 5 и α-спиралью E, позволяет снизить сродство к сульфонамиду и обеспечить резистентность [22]. В секвенированных геномах таких замен не обнаружено. Таким образом, lmo0224 точнее именовать folP и при отсутствии замен относить к генам корового генома.
Помимо активности MFS транспортера (norB), осуществляющего эффлюкс антибиотика, устойчивость к хинолонам могут обеспечить модификации ферментов — мишеней этого класса антибактериальных препаратов. К ним относятся гираза (GyrA, lmo0007) и топоизомераза (ParC, lmo1287). Чаще всего с устойчивостью ассоциированы мутации в позициях 83 и 87 (в нумерации GyrA E. coli) в одном из двух ферментов [23]. Во всех проанализированных геномах мы обнаружили Ser83 и Glu87 в последовательности ParC, что отвечает характеристикам резистентных к хинолонам топоизомераз.
Дополнительные гены резистентности мы выявили при исследовании только одной культуры, выделенной из отделяемого цервикального канала родильницы. Как показало полногеномное секвенирование, культура содержала небольшую примесь Enterococcus faecalis ST41. Гены ermB и cat, обеспечивающие устойчивость к макролидам, линкозамиду, стрептограмину B и фениколам, обнаружили в составе плазмиды, а гены tetM, dfrD, определяющие устойчивость к тетрациклину и триметаприму, в мобильном элементе хромосомы E. faecalis.
Обсуждение
Продолжительный мониторинг случаев ИЛ в Московском регионе позволил пополнить количество охарактеризованных клинических изолятов L. monocytogenes, выделенных в европейской части России. В базе данных BIGSdb-Lm к 26 изолятам 1971–2017 гг. прибавили 73 изолята 2018–2023 гг. Мониторинг показал выросшее разнообразие генотипов клинических изолятов L. monocytogenes как в первой, так и во второй филогенетических линиях. Увеличилась доля генотипов PLI у клинических изолятов с 15% в 1971–2017 гг. до 37% в 2018–2023 гг. В группе ПЛ произошёл самый существенный рост доли генотипов PLI (52% за весь период), увеличившись с 38% в 2018–2019 гг. до 58% в 2020–2023 гг. Сравнение с данными наиболее крупной европейской коллекции клинических изолятов — референсного центра Франции по листериям (2584 изолята PLI и PLII, собранных в 2005–2013 гг.) — показывает, что в группе ПЛ доля PLI уже приближается к показателям Франции (PLI — 66%) [24], для всей нашей выборки 2018–2023 гг. это показатель почти в 2 раза ниже. Настораживает разнообразие гипервирулентных генотипов среди изолятов, выделенных в группе ПЛ: ST1, ST4, ST6, ST219, ST194. M.M. Maury и соавт., авторы термина «гипервирулентный», доказали в экспериментах на модельных животных более сильную ассоциацию СС4 (ST4, ST219) с перинатальной инфекцией, а также роль LIPI-4 (система транспорта сахаров, PTS) в преодолении бактерией плацентарного барьера [24]. LIPI-4 мы обнаружили также в геномах изолятов ST194 (CC315). Если изоляты СС4 не выделяли из продуктов питания в России, то изоляты ST194 (CC315) были обнаружены E.K. Psareva и соавт. в коллекции изолятов, выделенных из молочных продуктов в 2005 г. [4]. Из генотипов изолятов PLII, строго ассоциированных с продуктами питания во Франции (ST9, ST121) [24], в период мониторинга среди клинических отмечен изолят ST121. Из промежуточных между изолятами, достоверно ассоциированными с клиническими проявлениями и изолятами, ассоциированными с продуктами, в нашем списке генотипов PLII 14 из 17 ST совпало со списком генотипов Франции. Из масштабных исследований последних лет заслуживает внимание изучение изолятов, выделенных при абортах крупного рогатого скота (191 изолят 2013–2018 гг.) в граничащей с Россией Латвии [25]. Доля изолятов PLI в этом исследовании составила 2%. Более половины изолятов относились к ST29, ST37, ST451 и ST7 [25], обнаруженным и нами при мониторинге. В целом из списка генотипов PLII мониторинга со списком генотипов Латвии совпало 11 из 17 ST.
Вызывает опасение снижение минимального возраста пациентов с ИЛ, а также существенный рост доли умерших в когорте МС в период пандемии COVID-19. Большинство пациентов более молодого возраста имели в анамнезе лабораторно подтверждённый COVID-19, предшествовавший ИЛ. Можно предположить, что именно госпитализация в связи с COVID-19 послужила причиной эпидемической связи изолятов L. monocytogenes одного генотипа из перечня ST4, ST425, ST21. Например, в Италии в период COVID-19 (в сентябре–октябре 2020 г.) описана вспышка внутрибольничного листериоза, вызванного L. monocytogenes ST451. Контаминацию обнаружили в мясе на больничной кухне. Инфицированы 4 пациента (1 иммунокомпрометированный, 3 с онкозаболеваниями) [26]. В Германии 13 из 39 случаев листериоза, вызванного L. monocytogenes ST8, было связано с пребыванием пациентов в лечебно-профилактических учреждениях и питанием готовой к употреблению мясной продукцией одного и того же производства. Различия в коровых геномах изолятов составили 0–10 локусов, что доказало эпидемическую связь [27].
Анализ генов антибиотикорезистентности показал наличие во всех секвенированных геномах генотип-специфичных аллелей fosX (lmo1702), lin (lmo0919), mprF (lmo1695), norB (lmo2818), рarC (lmo1287), обеспечивающих устойчивость к фосфомицину, линкозамиду, катионным пептидам и хинолонам. Выявление дополнительных генов резистентности (ermB, cat, tetM, dfrD) в культуре с примесью E. faecalis ещё раз подчёркивает необходимость проверки чистоты культуры перед анализом, особенно при выделении из такого биотопа, как цервикальный канал, в котором у 18% беременных женщин показано наличие E. faecalis в норме и в 2 раза чаще при воспалительных процессах [28].
Дополнительные гены устойчивости к дезинфектантам, а также стрессовым факторам окружающей среды обнаружили в геномах изолятов, содержащих плазмиды: ST8, ST20, ST121, ST425. Обработка как внутрибольничных, так и производственных помещений при выявлении таких изолятов требует разработки новых комбинаций дезинфицирующих средств.
Заключение
Многоцентровой мониторинг ИЛ в Московском регионе показал, что не только L. monocytogenes гипервирулентных генотипов первой филогенетической линии (PLI), но прежде всего гиповирулентных генотипов PLII является возбудителем заболевания пациентов групп риска. Новая группа риска — пациенты, перенёсшие COVID-19, вероятность внутрибольничного инфицирования, расширившееся разнообразие генотипов L. monocytogenes, появление штаммов с плазмидами, несущими гены устойчивости к дезинфектантам, стрессовым факторам окружающей среды, — вот новые реалии, которые необходимо учитывать при диагностике и лечении ИЛ.
Этическое утверждение. Исследование проводилось при добровольном информированном согласии пациентов. Протокол исследования одобрен Комитетом по биомедицинской этике НИЦЭМ им. Н.Ф. Гамалеи (протокол № 14 от 04.07.2018).
Благодарность. Мы благодарим Институт Пастера за курирование и обслуживание баз данных BIGSdb-Pasteur на https://bigsdb.pasteur.fr/.
Источник финансирования. Работа выполнена при финансовой поддержке Государственного задания НИЦЭМ имени почетного академика Гамалеи Н.Ф.
Конфликт интересов. Авторы декларируют отсутствие явных и потенциальных конфликтов интересов, связанных с публикацией настоящей статьи.
1 WHO. Listeriosis. URL: https://www.who.int/news-room/fact-sheets/detail/listeriosis (дата обращения 10.06.2023).
2 Управление Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека по городу Москве. Динамика инфекционной и паразитарной заболеваемости населения МОСКВЫ за январь–декабрь 2021 года в сравнении с аналогичным периодом прошлого года.
URL: https://77.rospotrebnadzor.ru (дата обращения: 10.05.2023)
3 Управление Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека по городу Москве.Сведения о деятельности лабораторий санитарно-гигиенического, микробиологического и паразитологического профиля федеральных бюджетных учреждений здравоохранения — центров гигиены и эпидемиологии за 2020 год.
URL: https://77.rospotrebnadzor.ru (дата обращения: 10.05.2023)
4 Bacterial Isolate Genome Sequence Database for L. monocytogenes. URL: https://bigsdb.pasteur.fr/listeria/
5 Virulence Factors Database. URL: http://www.mgc.ac.cn/VFs/
6 URL: http://www.mgc.ac.cn/cgi-bin/VFs/v5/main.cgi?func=VFanalyzer/
7 Comprehensive Antibiotic Resistance Database.
URL: https://card.mcmaster.ca/
8 URL: https://cge.food.dtu.dk/services/
Об авторах
Ольга Львовна Воронина
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Гамалеи Н.Ф.
Email: olv550@gmail.com
ORCID iD: 0000-0001-7206-3594
к.б.н., доцент, зав. лаб. анализа геномов
Россия, МоскваНаталья Николаевна Рыжова
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Гамалеи Н.Ф.
Email: rynatalia@yandex.ru
ORCID iD: 0000-0001-5361-870X
к.б.н., с.н.с. лаб. анализа геномов
Россия, МоскваМарина Сергеевна Кунда
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Гамалеи Н.Ф.
Email: markunda99@gmail.com
ORCID iD: 0000-0003-1945-0397
к.б.н., с.н.с. лаб. анализа геномов
Россия, МоскваЕкатерина Ивановна Аксенова
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Гамалеи Н.Ф.
Автор, ответственный за переписку.
Email: aksenova16@yandex.ru
ORCID iD: 0000-0003-2704-6730
к.б.н., с.н.с. лаб. анализа геномов
Россия, МоскваТатьяна Игоревна Карпова
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Гамалеи Н.Ф.
Email: dragovtceva@yandex.ru
ORCID iD: 0000-0002-9633-7876
д.б.н., в.н.с. лаб. легионеллеза
Россия, МоскваАлина Рантиковна Мелкумян
Городская клиническая больница имени Ф.И. Иноземцева
Email: alinamelkumyan@yandex.ru
ORCID iD: 0000-0002-5494-415X
к.м.н., зав. центром лабораторной диагностики
Россия, МоскваЕлена Анатольевна Климова
Московский государственный медико-стоматологический университет имени А.И. Евдокимова
Email: elena_klimova_@mail.ru
ORCID iD: 0000-0003-4319-8144
д.б.н., профессор каф. инфекционных болезней и эпидемиологии
Россия, МоскваГалина Николаевна Кареткина
Московский государственный медико-стоматологический университет имени А.И. Евдокимова
Email: karetkinagn@mail.ru
ORCID iD: 0000-0001-7850-2826
к.м.н., доцент каф. инфекционных болезней и эпидемиологии
Россия, МоскваЕвгений Андреевич Посуховский
Московский государственный медико-стоматологический университет имени А.И. Евдокимова
Email: kaempfer@inbox.ru
ORCID iD: 0000-0002-7763-0313
врач-ординатор кафедры инфекционных болезней и эпидемиологии
Россия, МоскваОльга Александровна Груздева
Российская медицинская академия непрерывного профессионального образования
Email: gruzdeva_oa@mail.ru
ORCID iD: 0000-0002-1244-1925
д.м.н., профессор каф. эпидемиологии
Россия, МоскваИгорь Семенович Тартаковский
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Гамалеи Н.Ф.
Email: itartak@list.ru
ORCID iD: 0000-0003-4825-8951
д.б.н., профессор, зав. лаб. легионеллеза
Россия, МоскваСписок литературы
- Psareva E.K., Egorova I.Y., Liskova E.A., et al. Retrospective study of Listeria monocytogenes isolated in the territory of inner Eurasia from 1947 to 1999. Pathogens. 2019;8(4):184. DOI: https://doi.org/10.3390/pathogens8040184
- Асташкин Е.И., Алексеева Е.А., Борзенков В.Н. и др. Молекулярно-генетическая характеристика полирезистентных штаммов Listeria monocytogenes и идентификация новых сиквенс-типов. Молекулярная генетика, микробиология и вирусология. 2021;39(4):3–13. DOI: https://doi.org/10.17116/molgen2021390413 EDN: https://www.elibrary.ru/eixxrf. Astashkin E.I., Alekseeva E.A., Borzenkov V.N., et al. Molecular-genetic characteristics of polyresistant Listeria monocytogenes strains and identification of new sequence types. Molecular Genetics, Microbiology and Virology. 2021; 36(4): 159–69. DOI: https://doi.org/10.3103/S0891416821040029 EDN: https://www.elibrary.ru/xverak
- Воронина О.Л., Кунда М.С., Рыжова Н.Н. и др. Листериоз: генотипирование как ключ к выявлению возможного источника заражения. Клиническая микробиология и антимикробная химиотерапия. 2019;21(4):261–73. Voronina O.L., Kunda M.S., Ryzhova N.N., et al. Listeriosis: genotyping as a key for identification a possible source of infection. Clinical Microbiology and Antimicrobial Chemotherapy. 2019;21(4):261–73. DOI: https://doi.org/10.36488/cmac.2019.4.261-273 EDN: https://www.elibrary.ru/chgmuc
- Psareva E.K., Liskova E.A., Razheva I.V., et al. Diversity of Listeria monocytogenes strains isolated from food products in the Central European part of Russia in 2000-2005 and 2019-2020. Foods. 2021;10(11):2790. DOI: https://doi.org/10.3390/foods10112790
- Yushina Yu.K., Kuznetsova O.A., Tutelyan A.V., et al. Prevalence of Listeria monocytogenes in meat products during 2017–2019 depending on technological factors and seasons. Theory and Practice of Meat Processing. 2022; 7(4): 238–46. DOI: https://doi.org/10.21323/2414-438X-2022-7-4-238-246
- Белова Л.В., Репникова Н.Е., Сухова Е.А. Результаты микробиологических исследований рыбы и рыбопродуктов и требования по профилактике заболеваний листериозом. Проблемы медицинской микологии. 2022;24(2):35–7. Belova L.V., Repnikova N.E., Sukhova E.A. Results of microbiological studies of fish and fish products and requirements for the prevention of listeriosis diseases. Problems in Medical Mycology. 2022;24(2):35–7. DOI: https://doi.org/10.24412/1999-6780-2022-2-34-37 EDN: https://www.elibrary.ru/ctfvvk
- Voronina O.L., Ryzhova N.N., Kunda M.S., et al. Diversity and pathogenic potential of Listeria monocytogenes isolated from environmental sources in the Russian Federation. IJMER. 2015;5(3):5–15.
- Алексеева E.A., Полосенко О.В., Фурсова Н.К. и др. Первый случай выявления Listeria monocytogenes сиквенс-типов ST7, ST20, ST425 в сточных водах при обследовании водных объектов Вологодской области. Журнал микробиологии, эпидемиологии и иммунобиологии. 2022;99(4):453–64. Alekseeva E.A., Polosenko O.V., Fursova N.K., et al. The first case of detection of Listeria monocytogenes sequence types ST7, ST20, ST425 in wastewater during an investigation of water bodies in the Vologda region. Journal of Microbiology Epidemiology and Immunobiology. 2022;99(4):453–64. DOI: https://doi.org/10.36233/0372-9311-266 EDN: https://www.elibrary.ru/gashhr
- Климова Е.А., Воронина О.Л., Кареткина Г.Н. и др. Листериоз и пандемия COVID-19. Инфекционные болезни: новости, мнения, обучение. 2022;11(1):102–12. Klimova E.A., Voronina O.L., Karetkina G.N., et al. Listeriosis and the COVID-19 pandemic. Infectious Diseases: News, Opinions, Training. 2022;11(1):102–12. DOI: https://doi.org/10.33029/2305-3496-2022-11-1-102-112 EDN: https://www.elibrary.ru/cgttxv
- Воронина О.Л., Тартаковский И.С., Ющук Н.Д. и др. Анализ спорадических случаев инвазивного листериоза в мегаполисе. Журнал микробиологии, эпидемиологии и иммунобиологии. 2020;97(6):547–55. Voronina O.L., Tartakovskii I.S., Yushchuk N.D., et al. Analysis of sporadic cases of invasive listeriosis in a metropolis. Journal of Microbiology Epidemiology and Immunobiology. 2020;97(6):547–55. DOI: https://doi.org/10.36233/0372-9311-2020-97-6-3 EDN: https://www.elibrary.ru/dziqzj
- Воронина О.Л., Рыжова Н.Н., Аксёнова Е.И. и др. Генетическое разнообразие листерий, выявленных в производственной среде переработки мяса. Молекулярная генетика, микробиология и вирусология. 2023;41(1):24–31. Voronina O.L., Ryzhova N.N., Aksenova E.I., et al. Genetic diversity of listeria found in the meat processing environment. Molecular Genetics, Microbiology and Virology. 2023;41(1):24–31. DOI: https://doi.org/10.17116/molgen20234101124 EDN: https://www.elibrary.ru/wtlkqz
- Moura A., Criscuolo A., Pouseele H., et al. Whole genome-based population biology and epidemiological surveillance of Listeria monocytogenes. Nat. Microbiol. 2016;2:16185. DOI: https://doi.org/10.1038/nmicrobiol.2016.185.
- Chen L., Yang J., Yu J., et al. VFDB: a reference database for bacterial virulence factors. Nucleic Acids Res. 2005;33(Issue suppl. 1):D325–8. DOI: https://doi.org/10.1093/nar/gki008
- Alcock B.P., Huynh W., Chalil R., et al. CARD 2023: expanded curation, support for machine learning, and resistome prediction at the Comprehensive Antibiotic Resistance Database. Nucleic Acids Res. 2023;51(D1):D690–9. DOI: https://doi.org/10.1093/nar/gkac920
- Chmielowska C., Korsak D., Chapkauskaitse E., et al. Plasmidome of Listeria spp. — the repA-family business. Int. J. Mol. Sci. 2021;22(19):10320. DOI: https://doi.org/10.3390/ijms221910320
- Gushchin V.A., Dolzhikova I.V., Shchetinin A.M., et al. Neutralizing activity of sera from Sputnik V-vaccinated people against variants of concern (VOC: B.1.1.7, B.1.351, P.1, B.1.617.2, B.1.617.3) and Moscow endemic SARS-CoV-2 variants. Vaccines. 2021;9(7):779. DOI: https://doi.org/10.3390/vaccines9070779
- Klink G.V., Safina K.R., Nabieva E., et al. The rise and spread of the SARS-CoV-2 AY.122 lineage in Russia. Virus Evol. 2022;8(1):veac017. DOI: https://doi.org/10.1093/ve/veac017
- Gangavarapu K., Latif A.A., Mullen J.L., et al. Outbreak.info genomic reports: scalable and dynamic surveillance of SARS-CoV-2 variants and mutations. Nat. Methods. 2023; 20(4): 512–22. DOI: https://doi.org/10.1038/s41592-023-01769-3
- Воронина О.Л., Кунда М.С., Рыжова Н.Н. и др. Закономерности селекции полигостальных убиквитарных микроорганизмов на примере представителей трех таксонов. Молекулярная биология. 2015;49(3):430–41. DOI: https://doi.org/10.7868/S0026898415030179 EDN: https://www.elibrary.ru/tqqvfh. Voronina O.L., Kunda M.S., Ryzhova N.N., et al. Regularities of the ubiquitous polyhostal microorganisms selection by the example of three taxa. Molecular Biology. 2015;49(3):380–90. DOI: https://doi.org/10.1134/S0026893315030176 EDN: https://www.elibrary.ru/ueytrz
- Adgamov R., Zaytseva E., Thiberge J.M., et al. Genetically related Listeria monocytogenes strains isolated from lethal human cases and wild animals. In: Caliskan M., ed. Genetic Diversity in Microorganisms. Chapter 9. IntechOpen; 2012. DOI: https://doi.org/10.5772/32913
- Lebrun M., Loulergue J., Chaslus-Dancla E., Audurier A. Plasmids in Listeria monocytogenes in relation to cadmium resistance. Appl. Environ. Microbiol. 1992;58(9):3183–6. DOI: https://doi.org/10.1128/aem.58.9.3183-3186.1992
- Vedantam G., Guay G.G., Austria N.E., et al. Characterization of mutations contributing to sulfathiazole resistance in Escherichia coli. Antimicrob. Agents Chemother. 1998;42(1):88–93. DOI: https://doi.org/10.1128/AAC.42.1.88
- Aldred K.J., Kerns R.J., Osheroff N. Mechanism of quinolone action and resistance. Biochemistry. 2014;53(10):1565–74. DOI: https://doi.org/10.1021/bi5000564
- Maury M.M., Tsai Y.H., Charlier C., et al. Uncovering Listeria monocytogenes hypervirulence by harnessing its biodiversity. Nat. Genet. 2016;48(3):308–13. DOI: https://doi.org/10.1038/ng.3501
- Šteingolde Ž., Meistere I., Avsejenko J., et al. Characterization and genetic diversity of Listeria monocytogenes isolated from cattle abortions in Latvia, 2013–2018. Vet. Sci. 2021;8(9):195. DOI: https://doi.org/10.3390/vetsci8090195
- Russini V., Spaziante M., Zottola T., et al. A nosocomial outbreak of invasive listeriosis in an Italian hospital: epidemiological and genomic features. Pathogens. 2021;10(5):591. DOI: https://doi.org/10.3390/pathogens10050591
- Lachmann R., Halbedel S., Adler M., et al. Nationwide outbreak of invasive listeriosis associated with consumption of meat products in health care facilities, Germany, 2014–2019. Clin. Microbiol. Infect. 2021;27(7):1035.e1–e5. DOI: https://doi.org/10.1016/j.cmi.2020.09.020
- Каганова М.А., Спиридонова Н.В., Казакова А.В. и др. Особенности микробиоты цервикального канала при дородовом излитии околоплодных вод и доношенной беременности. Акушерство и гинекология. 2019;(5):77–84. Kaganova M.A., Spiridonova N.V., Kazakova A.V., et al. Features of the cervical canal microbiota in prenatal amniorrhea and full-term pregnancy. Obstetrics and Gynecology. 2019;(5):77–84.DOI: https://doi.org/10.18565/aig.2019.5.77-84 EDN: https://www.elibrary.ru/hscrpw
Дополнительные файлы