Анализ последовательностей некодирующей контрольной области ДНК изолятов вируса Джона Каннингема у пациентов с рассеянным склерозом, получавших натализумаб
- Авторы: Тюменцева М.А.1, Тюменцев А.И.1, Захарова М.Н.2, Аскарова Л.Ш.2, Симанив Т.О.2, Пирадов М.А.2, Акимкин В.Г.1
-
Учреждения:
- Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
- Научный центр неврологии
- Выпуск: Том 100, № 1 (2023)
- Страницы: 7-25
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/4739
- DOI: https://doi.org/10.36233/0372-9311-341
- ID: 4739
Цитировать
Аннотация
Введение. Вирус Джона Каннингема (JCPyV) вызывает фатальное демиелинизирующее заболевание центральной нервной системы, известное как прогрессирующая мультифокальная лейкоэнцефалопатия (ПМЛ). У здоровых людей некодирующая контрольная область (NCCR) JCPyV не перестраивается, в то время как NCCR у пациентов с ослабленным иммунитетом часто перестраиваются и могут быть связаны с развитием ПМЛ. В связи с этим у пациентов, получающих терапию натализумабом, действующим на уровне гематоэнцефалического барьера и вызывающим уменьшение миграции лимфоцитов и моноцитов в воспалительные очаги, повышается риск развития ПМЛ.
Цель исследования — проанализировать последовательности NCCR изолятов JCPyV у пациентов с рассеянным склерозом (РC), получавших натализумаб.
Материалы и методы. Для оценки структуры NCCR JCPyV у российских пациентов с РС, получавших натализумаб, методом ПЦР с вложенной парой праймеров проанализировано 26 образцов плазмы крови и 8 образцов цереброспинальной жидкости. NCCR, присутствующие в образцах, были клонированы и секвенированы по методу Сэнгера. Все последовательности JCPyV NCCR сравнивали с последовательностью архетипа и картировали. Последовательности NCCR были также проанализированы на наличие предполагаемых сайтов связывания факторов транскрипции.
Результаты. Обнаружены 48 последовательностей NCCR. Анализ показал, что до 55% NCCR идентифицируются как перестроенные NCCR, остальные являются архетипоподобными. Все полученные последовательности можно разделить на 6 типов с преобладанием одного паттерна перестройки. Этот перестроенный NCCR был также обнаружен у пациента с подтверждённым диагнозом ПМЛ с плохим прогнозом. Все перестроенные NCCR характеризовались наличием дополнительных сайтов связывания факторов транскрипции.
Заключение. Проведённое исследование позволило впервые выявить паттерны NCCR, характерные для больных РС, принимавших натализумаб, в России, что подтверждает необходимость дальнейшего исследования перестроек NCCR у пациентов с РС, получавших натализумаб, для расширения представлений о возникновении нейровирулентных вариантов JCPyV.
Ключевые слова
Полный текст
Введение
Полиомавирус 2 человека, также известный как вирус JC или вирус Джона Каннингема (JCPyV), является представителем рода Betapolyomavirus, семейства Polyomaviridae. JCPyV — это вирус с двуцепочечным ДНК-геномом без оболочки, геном которого составляет приблизительно 5100 п.о. Геном JCPyV можно разделить на три части: некодирующую контрольную область (NCCR), область ранних вирусных генов и область поздних вирусных генов [1].
NCCR — это гипервариабельная промоторная/энхансерная область JCPyV длиной около 400 п.о., которая содержит большинство элементов, необходимых для инициации ранней и поздней транскрипции и экспрессии вирусных генов, включая TATA-боксы и сайты связывания факторов транскрипции [2]. Область ранних вирусных генов начинается слева от точки начала репликации и кодирует малый Т-антиген, большой Т-антиген и укороченный вариант большого Т-антигена. Эти белки отвечают за репликацию вируса и экспрессию поздних генов [3, 4]. Область поздних вирусных генов расположена справа от точки начала репликации и кодирует три структурных белка: главный капсидный белок VP1 и минорные капсидные белки VP2 и VP3. Каждый вирион JCPyV содержит в общей сложности 72 пентамерных капсомера VP1 с T = 7 икосаэдрической симметрией. Внутри капсида одна молекула — VP2 или VP3 — присоединена к каждому капсомеру VP1 [5].
JCPyV является этиологическим агентом, ведущим к развитию редкого прогрессирующего, в основном фатального, демиелинизирующего заболевания центральной нервной системы, известного как прогрессирующая мультифокальная лейкоэнцефалопатия (ПМЛ), развивающегося у пациентов с нарушенным гуморальным и клеточным иммунитетом [6]. JCPyV может проникать через гематоэнцефалический барьер в центральную нервную систему, где он заражает олигодендроциты и астроциты [7, 8]. По некоторым данным, 70–90% людей инфицированы JCPyV [9, 10]. У здоровых людей обнаруживается неперестроенная «архетипическая» форма JCPyV, которая считается безопасной, в то время как перестроенная форма, известная как «прототип», обнаруживается в центральной нервной системе пациентов с ПМЛ. «Прототип» почти всегда имеет различия в NCCR по сравнению с последовательностью JCPyV, обнаруженной у здоровых людей [11, 12]. Эти различия в NCCR могут изменять тропизм JCPyV, изменяя сайты связывания ДНК для клеточных факторов транскрипции в клетках, пермиссивных для инфекции [2, 13]. Предполагается также, что эти различия активизируют репликацию вируса и транскрипцию генов в глиальных клетках, в конечном итоге запуская литическую фазу [14, 15].
Иммунодефицит или иммуносупрессия приводят к реактивации JCPyV и переходу от «архетипа» к «прототипу». Известны случаи перехода JCPyV в агрессивную форму, индуцирующую ПМЛ, при использовании таких препаратов, как натализумаб, ритуксимаб, диметилфумарат, финголимод, эфализумаб, брентуксимаб ведотин и другие иммуносупрессоры [16–21].
Натализумаб — гуманизированное моноклональное антитело, специфически связывающее α4-интегрин, — препарат для лечения рецидивирующей формы РС. Натализумаб назначается пациентам с высокоактивным течением заболевания. В среднем ПМЛ диагностируется в 4,19 случая на 1000 пациентов, получающих натализумаб (95% ДИ 3,89–4,49) [22, 23].
Существуют три основных фактора риска, связанных с развитием ПМЛ во время лечения натализумабом:
- наличие антител к JCPyV (серопозитивные пациенты с анти-JCPyV);
- продолжительность терапии, особенно после 2 лет применения;
- предшествующая иммуносупрессивная терапия до начала терапии натализумабом [24].
Для предупреждения развития ПМЛ были разработаны специальные клинические рекомендации по ведению пациентов с рассеянным склерозом (РС), получающих терапию натализумабом. В клиническую практику внедрены планы управления рисками, включающими регулярный контроль индекса антител против JCPyV и потенциальной сероконверсии, а также МРТ головного мозга (для выявления характерных для ПМЛ изменений) [25–29]. При подозрении на ПМЛ проводится идентификация ДНК JCPyV в цереброспинальной жидкости (ЦСЖ) методом ПЦР [24].
Диагноз вероятного ПМЛ устанавливается при наличии характерных клинических и нейровизуализационных проявлений при отсутствии выявления JCPyV в ЦСЖ и мозговой ткани методом ПЦР, диагноз лабораторно подтверждённого ПМЛ — при выявлении ДНК JCPyV в ЦСЖ пациента. Гистологически подтверждённый ПМЛ устанавливается при выделении JCPyV методом ПЦР из биопсийного материала мозга больного. Для точного выявления инфицированных JCPyV пациентов с РС, получавших натализумаб, настоятельно рекомендуется обнаружение ДНК JCPyV и анализ последовательности NCCR для выявления механизмов перестройки NCCR, которые могут коррелировать с возникновением нейровирулентных вариантов [30].
Цель настоящей работы — оценить разнообразие вариантов NCCR в плазме и ЦСЖ пациентов с РС из России, получавших натализумаб.
Материалы и методы
Образцы
Образцы плазмы и ЦСЖ были получены от пациентов с РС, принимавших натализумаб, с индексом антител против JCPyV ≥ 1,5. Каждый пациент, должным образом проинформированный, подписал информированное согласие на участие в исследовании. Протокол исследования одобрен Этическим комитетом Научного центра неврологии (протокол № 1-2/22 от 19.01.2022).
Все образцы хранили в замороженном состоянии и оттаивали на льду перед использованием для выделения вирусной ДНК.
Экстракция вирусной ДНК
ДНК JCPyV экстрагировали в общей сложности из 26 образцов плазмы и 8 образцов ЦСЖ. Образцы крови собирали в пробирки «VACUETTE® 4 мл LH Lithium Heparin» («Greiner Bio-One») и центрифугировали при 3500 об/мин в течение 10 мин. ДНК выделяли из 200 мкл плазмы и/или ЦСЖ с помощью набора «DNeasy® Blood & Tissue Kit» («Qiagen Inc.»), элюцию ДНК проводили в 75 мкл элюирующего буфера. Концентрацию ДНК JCPyV определяли с помощью спектрофотометра «NanoDrop 2000c» («Thermo Fisher Scientific»). Все выделенные образцы ДНК хранили в замороженном виде до использования.
Количественное определение ДНК JCPyV
Количественное определение ДНК JCPyV проводили с использованием набора «AmpliSens® JCV-BKV screen/monitor-FRT PCR» (ЦНИИ Эпидемиологии Роспотребнадзора) с пределом обнаружения 5 × 102 гЭкв/мл в соответствии с рекомендациями производителя.
Амплификация последовательностей JCPyV NCCR
Для амплификации последовательностей NCCR из вирусной ДНК методом ПЦР с вложенной парой праймеров использовали две пары праймеров [31]. Для первого раунда ПЦР пара внешних праймеров:
- 1 — прямой праймер 5'-GATTCCTCCCTATTCAGCACTTTG-3';
- 1 — обратный праймер 5'-CACCTGTGCAAAAGTCCAGC-3'.
Эта пара праймеров позволяет амплифицировать фрагменты генома архетипа JCPyV (номер в GenBank: AB038249 [32]) размером 540 п.о. Для второго раунда амплификации пара внутренних праймеров:
- 2 — прямой праймер 5'-GGCCTCCTAAAAAGCC-3';
- 2 — обратный праймер 5'-TCCACTCCAGGTTTTACTAA-3'.
Эти праймеры использовались для амплификации фрагмента размером 386 п.о. архетипа JCPyV (номер в GenBank: AB038249).
Высокоточная ДНК-полимераза «Phusion™ High-Fidelity» («Thermo Fisher Scientific») была использована для первого раунда амплификации. Температурный профиль реакции:
- начальная денатурация: 98°C в течение 30 с;
- 35 циклов: 98°C в течение 10 с, 55°C в течение 45 с, 72°C в течение 30 с;
- окончательная элонгация: 72°C в течение 5 мин.
Из первого раунда ПЦР забирали 3 мкл ампликона для проведения второго раунда ПЦР с использованием Taq-полимеразы (смесь для ПЦР 2-Blue, ЦНИИ Эпидемиологии Роспотребнадзора).
Температурный профиль амплификации:
- начальная денатурация: 95ºC в течение 3 мин;
- 40 циклов: 95°C в течение 15 с, 55°C в течение 45 с, 72°C в течение 30 с;
- окончательная элонгация: 72°C в течение 5 мин.
Продукты ПЦР, полученные после второго раунда амплификации, анализировались на 1% агарозном геле путём окрашивания бромидом этидия. Продукты ПЦР использовали для прямого секвенирования ДНК по Сэнгеру и клонирования.
Клонирование последовательностей JCPyV NCCR
NCCR JCPyV, присутствующие в образцах плазмы крови и/или ЦСЖ, амплифицированные методом ПЦР с вложенной парой праймеров [31], были клонированы в вектор pGEM-T (Promega®). Полученные в результате клонирования плазмиды трансформировали в химически компетентные клетки E. coli XL10-Gold. Трансформированные E. coli выращивали при 37°C на чашках Luria-Bertani с добавлением ампициллина (100 мкг/мл), X-gal (80 мкг/мл) и IPTG (0,5 мМ). Скрининг полученных клонов проводили с использованием праймеров M13forward и M13reverse. После проведения скрининга секвенировали 10–20 клонов, полученных из каждого образца плазмы крови и/или ЦСЖ.
Секвенирование последовательностей JCPyV NCCR
Продукты ПЦР, соответствующие областям JCPyV NCCR, перед секвенированием очищали с помощью набора для очистки «QIAquick PCR» («Qiagen Inc.») согласно протоколу производителя. Секвенирование ДНК выполняли с использованием генетического анализатора «Applied Biosystems 3500xL».
Амплификация последовательностей JCPyV VP1
Для определения генотипа JCPyV изолятов из образцов плазмы и/или ЦСЖ фрагмент гена вирусного белка 1 (VP1) JCPyV длиной 215 п.о. амплифицировали с использованием одной пары праймеров: JLP-15 (5'-ACAGTGTGGCCAGAATTCCACTACC-3') и JLP-16 (5'-TAAAGCCTCCCCCCCAACAGAAA-3') [33]. ПЦР проводили с использованием Taq-полимеразы (ПЦР-смесь 2-Blue, ЦНИИ Эпидемиологии Роспотребнадзора) при следующих условиях проведения реакции:
- начальная денатурация: 95°С, 3 мин;
- 40 циклов: 95°С в течение 15 с, 55°С в течение 45 с, 72°С в течение 30 с;
- окончательная элонгация: 72°C в течение 5 мин.
Продукты ПЦР анализировали с использованием 1% агарозного геля методом электрофореза путём окрашивания бромидом этидия, а затем проводили прямое секвенирование ДНК по Сэнгеру.
Секвенирование последовательностей ДНК JCPyV NCCR и VP1
Продукты ПЦР, соответствующие областям JCPyV NCCR и VP1, очищали с помощью набора для очистки ПЦР «QIAquick» («Qiagen Inc.») в соответствии с протоколом производителя. Секвенирование ДНК проводили с использованием генетического анализатора «Applied Biosystems 3500xL».
Все последовательности NCCR сравнивали с последовательностью CY архетипа (номер в GenBank: AB038249) [32]. Все последовательности NCCR были картированы с помощью программного обеспечения «SnapGene Viewer» и разделены на сегменты A, B, C, D, E и F, состоящие из 25, 23, 55, 66, 18 и 69 пар оснований соответственно [11].
Последовательности, полученные после амплификации области VP1, сравнивали с соответствующими последовательностями изолятов JCPyV разных генотипов/подтипов с регистрационными номерами GenBank AF015526 (генотип 1A), AF281599 (генотип 1B), AF030085 (генотип 2A), AF015532 (генотип 2B), AF015534 (генотип 2C), AF015536 (генотип 2D), AF295731 (генотип 3A), U73501 (генотип 3B), AF015528 (генотип 4), AF015537 (генотип 6), AF295737 (генотип 7) и AF281623 (генотип 8). Выравнивание последовательностей и филогенетический анализ проводили с использованием программного обеспечения «MEGA 7» [34].
Анализ нуклеотидных последовательностей NCCR
Последовательности NCCR анализировали на присутствие предполагаемых сайтов связывания факторов транскрипции с использованием виртуального лабораторного инструмента PROMO1 [35, 36].
Регистрационные номера последовательностей
Все последовательности NCCR были депонированы в базу данных GenBank NCBI, им были присвоены номера OM479515–OM479562.
Статистический анализ данных
Статистический анализ (t-критерий Стьюдента) был выполнен с использованием встроенного модуля «unpaired t test» программного обеспечения «GraphPad Prism™ 9».
Результаты
Количественное определение ДНК и генотипы JCPyV
Для 26 образцов плазмы крови и 8 образцов ЦСЖ, полученных от пациентов с РС, получавших натализумаб, с индексом антител к JCPyV ≥ 1,5, было проведено количественное определение ДНК JCPyV (табл. 1). Только у 1 пациента (NAT-1) была выявлена ДНК JCPyV в плазме и ЦСЖ. У этого пациента был диагностирован ПМЛ, и образцы были собраны, когда он уже находился в вегетативном состоянии. Большинство образцов из текущего исследования содержали ДНК JCPyV в количестве, меньшем чем предел обнаружения набора для ПЦР, используемого для количественного определения ДНК JCPyV (ниже 5×102 гЭкв/мл). Это согласуется с тем фактом, что во многих подтверждённых случаях ПМЛ продемонстрировано низкое количество копий JCPyV [24].
Таблица 1. Характеристика образцов, полученных от пациентов с РС, принимавших натализумаб
Table 1. Characteristics of samples obtained from MS patients treated with natalizumab
№ No. | Идентификатор пациента Patient ID | Количество инфузий натализумаба, шт. Number of natalizumab infusions, pcs | Индекс антител к JCPyV Anti-JCPyV antibody index | Количественное определение ДНК JCPyV, гЭкв/мл Quantification of JCPyV DNA, gEq/ml | Генотип JCPyV JCPyV genotype |
Плазма крови | Blood plasma | |||||
1 | NAT-1/1 | Н. д. | N. d. | Н. д. | N. d. | 6,88 × 102 | 1A |
2 | NAT-1/2 | Н. д. | N. d. | Н. д. | N. d. | ˂ 1 × 103 | 1A |
3 | NAT-2 | 36 | 2,14 | Н. о. | N. def. | Н. о. | N. def. |
4 | NAT-3 | 22 | 2,127 | Н. о. | N. def. | 1B |
5 | NAT-4/1 | 16 | 2,43 | Н. о. | N. def. | 1B |
6 | NAT-4/2 | 16 | 2,43 | Н. о. | N. def. | 1B |
7 | NAT-5 | 9 | 2,64 | Н. о. | N. def. | 1B |
8 | NAT-6 | 11 | Н. д. | N. d. | Н. о. | N. def. | 1B |
9 | NAT-7 | 32 | 2,63 | Н. о. | N. def. | Н. о. | N. def. |
10 | NAT-8/1 | 24 | 2,11 | Н. о. | N. def. | 1B |
11 | NAT-8/2 | 24 | 2,64 | Н. о. | N. def. | 1B |
12 | NAT-9 | 18 | 2,194 | Н. о. | N. def. | 1A |
13 | NAT-10/1 | 25 | 1,529 | Н. о. | N. def. | 1B |
14 | NAT-10/2 | 25 | 1,529 | Н. о. | N. def. | 1A |
15 | NAT-11 | 20 | 0,259 | Н. о. | N. def. | Н. о. | N. def. |
16 | NAT-12 | 26 | 2,582 | Н. о. | N. def. | 1B |
17 | NAT-13 | 28 | 2,38 | Н. о. | N. def. | 1B |
18 | NAT-14 | 35 | 2,179 | Н. о. | N. def. | 1A |
19 | NAT-15/1 | 27 | 2,353 | Н. о. | N. def. | 1A |
20 | NAT-16 | 12 | 2,773 | Н. о. | N. def. | Type 1 |
21 | NAT-17 | 40 | 3,4 | Н. о. | N. def. | 1B |
22 | NAT-18 | 24 | Н. д. | N. d. | Н. о. | N. def. | Н. о. | N. def. |
23 | NAT-19 | Н. д. | N. d. | Н. д. | N. d. | Н. о. | N. def. | 1B |
24 | NAT-20 | Н. д. | N. d. | Н. д. | N. d. | Н. о. | N. def. | 1B |
25 | NAT-21 | Н. д. | N. d. | Н. д. | N. d. | Н. о. | N. def. | 1B |
26 | NAT-22 | Н. д. | N. d. | 2,7 | Н. о. | N. def. | 1B |
ЦСЖ | Cerebrospinal fluid | |||||
1 | NAT-1/1 | Н. д. | N. d. | – | 1,45 × 103 | 1A |
2 | NAT-2 | 36 | – | Н. о. | N. def. | Н. о. | N. def. |
3 | NAT-3 | 22 | – | Н. о. | N. def. | 1B |
4 | NAT-4 | 16 | – | Н. о. | N. def. | 1B |
5 | NAT-5 | 9 | – | Н. о. | N. def. | 1B |
6 | NAT-10/3 | 25 | – | Н. о. | N. def. | 1B |
7 | NAT-15/2 | 27 | – | Н. о. | N. def. | 1B |
8 | NAT-18 | 24 | Н. д. | N. d. | Н. о. | N. def. | Н. о. | N. def. |
Примечание. Н. д. — нет данных; н. о. — не определено.
Note. N. d. — no data; N. def. — not defined.
Все обнаруженные изоляты JCPyV принадлежали к типу 1 (генотипы 1A и 1B, табл. 1), широко распространённому в Европе [37]. Наиболее часто наблюдаемым генотипом был тип 1В — у 75% пациентов. Интересно, что у пациента NAT-10 в плазме крови, отобранной в разные временные промежутки, был обнаружен JCPyV разных генотипов (09.07.2019 — 1B, 03.10.2019 — 1А), в то время как в ЦСЖ пациента, отобранной 21.10.2019, был обнаружен JCPyV генотипа 1B. У пациента NAT-15 в плазме крови, отобранной 01.11.2019, был выявлен JCPyV генотипа 1A, а в ЦСЖ, полученной 06.02.2020, — JCPyV генотипа 1B.
Типы последовательностей JCPyV NCCR у пациентов с РС, получавших натализумаб
NCCR JCPyV были обнаружены у 21 пациента с РС, получавшего натализумаб: 37 независимых последовательностей JCPyV NCCR были получены из 26 образцов плазмы, 10 независимых последовательностей JCPyV NCCR — из 8 образцов ЦСЖ. Дополнительно был проанализирован 1 случай первичного иммунодефицита ID-1, где NCCR JCPyV был обнаружен в плазме крови.
В соответствии с классификацией NCCR [31] более 50% NCCR, полученных из плазмы пациентов с РС, принимавших натализумаб, относятся к перестроенным NCCR подтипа II-R (52,63%; 20 из 38 последовательностей JCPyV NCCR). Почти половина (48,65%; 18 из 38) NCCR JCPyV, полученных из плазмы пациентов с РС, принимавших натализумаб, относятся к архетипоподобным NCCR подтипа II-S. Точно так же 60% (6 из 10) NCCR, полученные из ЦСЖ пациентов с РС, принимавших натализумаб, относятся к перестроенным NCCR подтипа II-R, а остальные NCCR — к архетипоподобному подтипу II-S (40%, 4 из 10).
Следует отметить, что JCPyV NCCR, полученные от 1 больного с подтверждённым диагнозом ПМЛ с неблагоприятным прогнозом (образцы были собраны, когда он уже находился в вегетативном состоянии) и выявляемой ДНК JCPyV, относились к перестроенным JCPyV NCCR подтипа II-R. С другой стороны, NCCR от другого пациента с РС, получавшего натализумаб, с подтверждённым диагнозом ПМЛ с благоприятным исходом и неопределяемой ДНК JCPyV принадлежали к архетипоподобному подтипу II-S.
Кроме того, последовательность NCCR, полученная из плазмы пациента с первичным иммунодефицитом (ID-1; номер в GenBank: OM479515), который не принимал натализумаб, на 99% гомологична опубликованному архетипу NCCR, за исключением 1 точечной мутации в сегменте F, и принадлежит к архетипу подтипа II-S NCCR.
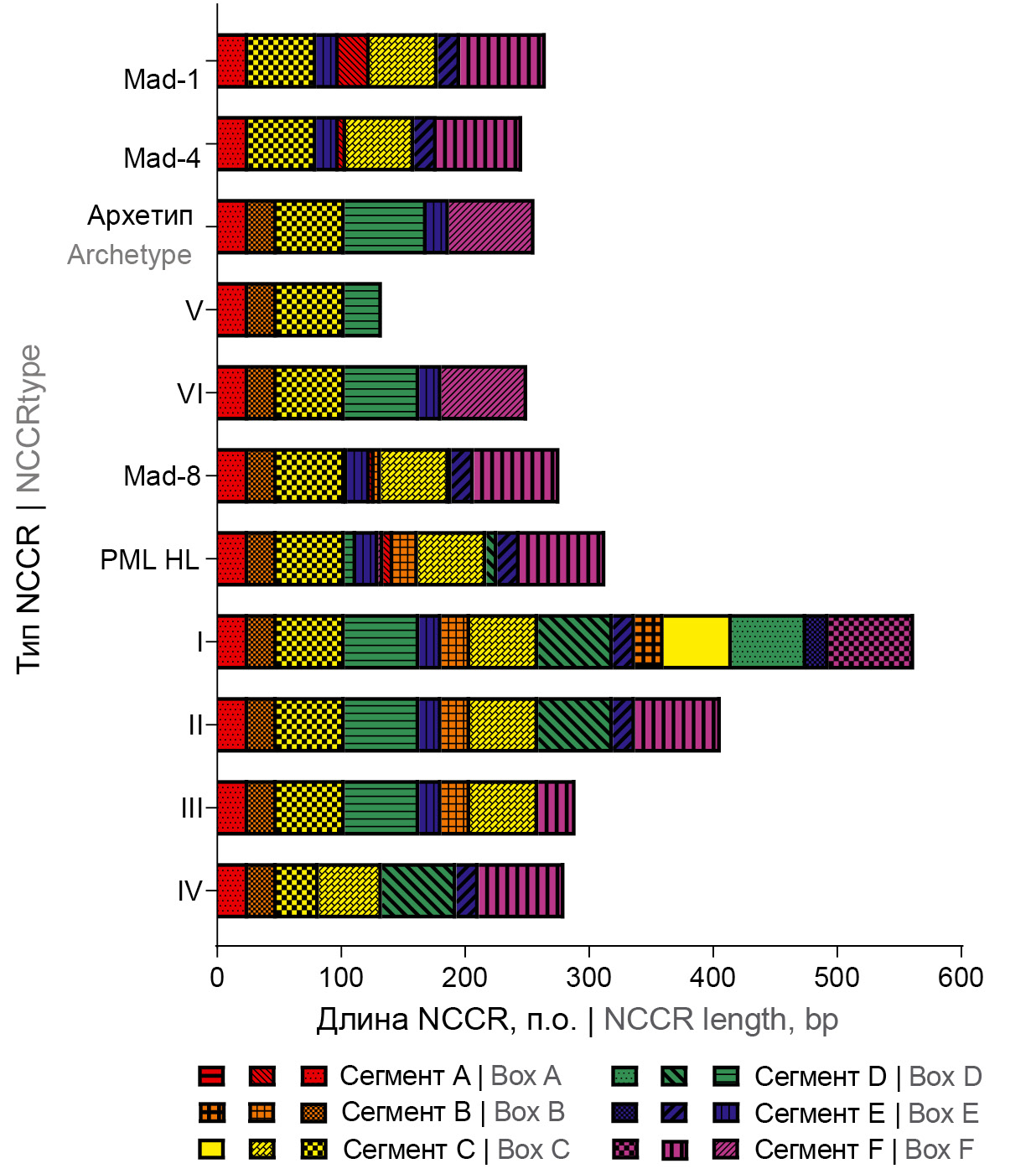
Все полученные последовательности JCPyV NCCR были разделены на 6 различных типов (без учёта точечных мутаций): типы I–IV относятся к перестроенным NCCR подтипа II-R (всего 26 NCCR, 20 из плазмы крови и 6, полученных из ЦСЖ, соответственно); тип V и VI — к архетипоподобному подтипу II-S (всего 22 NCCR, 18 из плазмы крови и 4 из ЦСЖ соответственно), формулы последовательностей приведены в табл. 2. Схематично обнаруженные типы последовательностей JCPyV NCCR представлены на рис. 1.
Таблица 2. Характеристика последовательностей NCCR, выявленных у пациентов с РС, получавших натализумаб
Table 2. Characterization of NCCR sequences identified in MS patients treated with natalizumab
№ No. | Номер в GenBank GenBank accession number | Формула последовательности Sequence formula |
Плазма крови | Blood plasma | ||
Тип I | Type I (II-R, (BCDE)3) | ||
1 | OM479519 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
Тип II | Type II (II-R, (BCDE)2) | ||
2 | OM479520 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
3 | OM479516 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
4 | OM479517 | A1-25B1-23C1-45-CCTAACCTCCTA-D3-39D46-66E1-18-F1-2-A25-B1-23C1-45 CCTAACCTCCTA-D3-39D46-66E1-18-F1-69 |
5 | OM479526 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
6 | OM479528 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
7 | OM479534 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
8 | OM479538 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
9 | OM479531 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
10 | OM479523 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
11 | OM479542 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
12 | OM479545 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
13 | OM479552 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
14 | OM479557 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
Тип III | Type III (II-R, BCDEBC) | ||
15 | OM479550 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55F40-69 |
16 | OM479558 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55F40-69 |
17 | OM479561 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55F40-69 |
18 | OM479555 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-3-B1-23C1-55F40-69 |
Тип IV | Type IV (II-R, C1-34C5-55) | ||
19 | OM479529 | A1-25B1-23C1-34C5-55D1-39D46-66E1-18F1-69 |
20 | OM479535 | A1-25B1-23C1-34C5-55D1-39D46-66E1-18F1-69 |
Тип V (II-S, ABCD30 и другие укороченные NCCR) | Type V (II-S, ABCD30 and other truncated NCCR) | ||
21 | OM479522 | A1-25B1-23C1-55D1-30 |
22 | OM479530 | A1-25B1-23C1-55D1-30 |
23 | OM479541 | A1-25B1-23C1-55D1-30 |
24 | OM479544 | A1-25B1-23C1-55D1-30 |
25 | OM479559 | A1-25B1-23C1-55D1-30 |
26 | OM479562 | A1-25B1-23C1-55D1-30 |
27 | OM479533 | A1-25B1-23C1-55D1-39D46-66 |
Тип VI (II-S, архетипоподобные с делецией 6 нуклеотидов в сегменте D) Type VI (II-S, archetype-like with Box D typical deletion) | ||
28 | OM479518 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
29 | OM479532 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
30 | OM479539 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
31 | OM479536 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
32 | OM479540 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
33 | OM479543 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
34 | OM479560 | A1-24B1-23C1-55D1-39D46-66E1-18F1-69 |
35 | OM479549 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
36 | OM479551 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
37 | OM479556 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
Архетип | Archetype | ||
38 | OM479515 | A1-25B1-23C1-55D1-66E1-18F1-69 |
ЦСЖ | Cerebrospinal fluid | ||
Тип I | Type I (II-R, (BCDE)3) | ||
1 | OM479554 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
Тип II | Type II (II-R, (BCDE)2) | ||
2 | OM479525 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
3 | OM479527 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
4 | OM479553 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55D1-39D46-66E1-18F1-69 |
Тип III | Type III (II-R, BCDEBC) | ||
5 | OM479521 | A1-25B1-23C1-55D1-39D46-66E1-18-F1-2-A25-B1-23C1-55F40-69 |
6 | OM479546 | A1-24B1-23C1-55D1-39D46-66E1-18-GGGAAT-B4-20-ATG-C1-52-CCCCA-F42-67 |
Тип V (II-S, ABCD30 и другие укороченные NCCR) | Type V (II-S, ABCD30 and other truncated NCCR) | ||
7 | OM479547 | A1-25B1-23C1-55D1-30 |
Тип VI (II-S, архетипоподобные с делецией 6 нуклеотидов в сегменте D) Type VI (II-S, archetype-like with Box D typical deletion) | ||
8 | OM479524 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
9 | OM479537 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
10 | OM479548 | A1-25B1-23C1-55D1-39D46-66E1-18F1-69 |
Рис. 1. Схематичное изображение структуры последовательностей JCPyV NCCR, выявленных у пациентов с РС, получавших натализумаб.
Fig. 1. Schematic representation of the structure of JCPyV NCCR sequences identified in MS patients treated with natalizumab.
Тип I представлен NCCR типа II-R, в котором фрагмент, состоящий из сегментов B, C, D и E (BCDE), тандемно повторяется трижды. NCCR I типа был обнаружен в образце плазмы пациента NAT-3 (5%; 1 из 20 перестроенных NCCR) и в образце ЦСЖ пациента NAT-18 (16,7%, 1 из 6 перестроенных NCCR). Подобно NCCR типа I, тип II представлен NCCR типа II-R, но BCDE-фрагмент тандемно повторяется дважды. NCCR типа II — самый распространённый среди образцов NCCR, он был обнаружен в 13 образцах плазмы (65%; 13 из 20 перестроенных NCCR) и 3 СМЖ (50%; 3 из 6 перестроенных NCCR).
NCCR типа I представляет собой новый перестроенный уникальный вариант NCCR, не имеющий гомологии c последовательностями NCCR, опубликованными ранее в NCBI GenBank (подтверждено нуклеотидным Blast-анализом). NCCR типа II также является перестроенным редким вариантом NCCR, имеющим гомологию с последовательностью NCCR NIID11-68 (номер GenBank: LC164353), опубликованной ранее в NCBI GenBank. Вариант NCCR типа II впервые идентифицирован в плазме крови и ЦСЖ пациента с подтверждённым диагнозом ПМЛ с неблагоприятным прогнозом и детектируемой ДНК JCPyV.
Тип III NCCR относится к типу II-R, для которого характерна дополнительная копия BC-фрагмента, интегрированная между сегментами E и F. NCCR III типа был обнаружен в 4 образцах плазмы (20%; 4 из 20 перестроенных NCCR) и в 2 образцах ЦСЖ (33,3%; 2 из 6 перестроенных NCCR).
Тип IV представлен II-R NCCR, где участок С повторяется тандемно. Этот вариант NCCR был обнаружен только в плазме крови 2 пациентов с РС, получавших натализумаб, NAT-8 и NAT-10 (10%; 2 из 20 перестроенных NCCR).
NCCR типов V и VI являются архетипоподобными NCCR подтипа II-S. Тип V — укороченный вариант NCCR, в котором отсутствуют либо сегменты E и F (8 из 9 случаев неперестроенных NCCR), либо сегменты A, B и C (1 из 9 неперестроенных NCCR). NCCR типа V наиболее часто обнаруживали в плазме крови (8 из 9 неперестроенных NCCR), а 1 раз — в образце ЦСЖ от больного NAT-15.
Тип VI представлен типом II-S NCCR с более чем 98% гомологией опубликованному архетипу NCCR CY (номер GenBank: AB038249), за исключением нуклеотидной делеции длиной 6 п.о. в сегменте D. Более половины (55,5%) NCCR типа VI обнаружены в образцах плазмы крови (10 из 18 II-S NCCR) и 75% (3 из 4 II-S NCCR) — в образцах ЦСЖ. Следует отметить, что NCCR типа VI был обнаружен в плазме крови и ЦСЖ пациента с диагнозом ПМЛ с благоприятным исходом и неопределяемой ДНК JCPyV.
Сравнительный анализ встречаемости предполагаемых сайтов связывания факторов транскрипции JCPyV NCCR у пациентов с РС, получавших натализумаб
Для проведения сравнительного анализа встречаемости предполагаемых сайтов связывания факторов транскрипции в анализ были включены последовательности «архетипа» NCCR (в качестве референсной последовательности), а также последовательности «прототипов» NCCR: Mad-1 [12, 38], Mad-4 [39, 40], Mad-8 [40] и PML HL [41]. Проведённый анализ показал, что NCCR всех типов содержат сопоставимое с архетипическим NCCR количество сайтов связывания факторов транскрипции Pentamut, L3 (Spi-B), L6 (Spi-B), L18 (Spi-B), L8 (Spi-B), Pseudo-NF-1 site, p53, AP-1-like site, AP-4, PEA3, GABP, GABP-alpha, TATA box, TFIID и TBP. В то же время количество сайтов связывания факторов транскрипции GA box (SP-1 site), c-Jun, NF-1 motif, NF-1, c-Fos, HMG I(Y), MEF-2A, C/EBPalpha, TEF-1 и GF1 в перестроенных NCCR типов I-IV было выше по сравнению с архетипоподобными NCCR (архетип, тип V и VI) (табл. 3).
Таблица 3. Предполагаемые сайты связывания факторов транскрипции в JCPyV NCCR у пациентов с РС, получавших натализумаб
Table 3. Putative binding sites of transcription factors in JCPyV NCCR in MS patients treated with natalizumab
Примечание. Белым обозначены предполагаемые сайты связывания факторов транскрипции, количество которых не изменилось по сравнению с последовательностью архетипа; светло-серым — предполагаемые сайты связывания факторов транскрипции, количество которых уменьшилось по сравнению с последовательностью архетипа; тёмно-серым — предполагаемые сайты связывания факторов транскрипции, количество которых увеличилось по сравнению с последовательностью архетипа.
Note. White indicates putative transcription factor binding sites that have not changed in number compared to the archetype sequence; light grey indicates putative transcription factor binding sites that have decreased in number compared to the archetype sequence; dark grey indicates putative transcription factor binding sites that have increased in number compared to the archetype sequence.
Кроме того, было показано, что в последовательностях NCCR, принадлежащих к типу II-R (тип I, II, III, IV, Mad-8 и PML HL), предполагаемые сайты связывания факторов транскрипции NF-1, GF1 и AP-1 встречаются достоверно чаще (p ˂ 0,0001), чем в последовательностях NCCR, принадлежащих к типу II-S (архетип, типы V и VI) (рис. 2).
Рис. 2. Сравнительный анализ встречаемости предполагаемых сайтов связывания факторов транскрипции NF-1, GF1 и AP-1 в JCPyV NCCR у пациентов с РС, получавших натализумаб.
Проанализированы 22 образца, относящихся к архетипоподобным NCCR подтипа II-S, и 26 образцов, принадлежащих к перестроенным NCCR подтипа II-R. ****p ˂ 0,0001.
Fig. 2. Comparative analysis of the occurrence of putative binding sites for transcription factors NF-1, GF1, and AP-1 in JCPyV NCCR in MS patients treated with natalizumab.
22 II-S archetype-like NCCR samples and 26 II-R rearranged NCCR samples were analyzed. ****p ˂ 0.0001.
Гетерогенность популяций JCPyV NCCR у пациентов с РС, получавших натализумаб
В ходе проведённого анализа выявлено, что у 12 пациентов с РС, принимавших натализумаб, в организме циркулируют несколько вариантов вируса (табл. 4). У 6 пациентов были обнаружены 2 варианта NCCR, у 4 — 3 варианта, у 2 — 4 варианта (табл. 4). Реже всего в комбинациях вариантов вируса встречались NCCR типов I и IV (2 из 12); NCCR типа III встречался 5 раз в обнаруженных комбинациях, NCCR типов II и V — 7 раз. Самым часто встречающимся NCCR в комбинациях вариантов вируса был архетипоподобный NCCR типа VI (9 из 12).
Таблица 4. Множественные варианты NCCR, обнаруженные у пациентов с РС, получавших натализумаб
Table 4. Multiple NCCR variants found in MS patients treated with natalizumab
№ No. | Идентификатор пациента Patient ID | Плазма крови, тип NCCR Blood plasma, NCCR type | ЦСЖ, тип NCCR Cerebrospinal fluid, NCCR type |
1 | NAT-3 | I, II | III |
2 | NAT-4 | II, V | II, VI |
3 | NAT-8 | II, IV, V | Н. д. | N. d. |
4 | NAT-9 | V, VI | Н. д. | N. d. |
5 | NAT-10 | II, IV, VI | VI |
6 | NAT-13 | V, VI | Н. д. | N. d. |
7 | NAT-14 | II, VI | Н. д. | N. d. |
8 | NAT-15 | II, V | III, V, VI |
9 | NAT-18 | II, III, VI | I, II |
10 | NAT-19 | III, VI | Н. д. | N. d. |
11 | NAT-21 | V, VI | Н. д. | N. d. |
12 | NAT-22 | III, V | Н. д. | N. d. |
Примечание. Н. д. — нет данных.
Note. N. d. — no data.
Отметим, что различные варианты вируса встречались в разных компартментах организма (плазма крови и ЦСЖ). Однако достоверных отличий в частоте встречаемости перестроенных и архетипоподобных вариантов NCCR в разных компартментах организма не обнаружено.
Кроме того, анализ вариантов NCCR, обнаруженных в парных образцах плазмы крови и ЦСЖ (8 пациентов), показал, что в 3 случаях в плазме крови и ЦСЖ был один вариант NCCR (тип II в 2 случаях, тип VI — в 1), тогда как в остальных случаях — различные (множественные) варианты вируса (табл. 5).
Таблица 5. Варианты NCCR, обнаруженные в парных образцах плазмы крови и ЦСЖ у пациентов с РС, получавших натализумаб
Table 5. NCCR variants detected in paired plasma and cerebrospinal fluid samples from MS patients treated with natalizumab
№ No. | Идентификатор пациента Patient ID | Плазма крови, тип NCCR Blood plasma, NCCR type | ЦCЖ, тип NCCR Cerebrospinal fluid, NCCR type |
1 | NAT-1 | II | II |
2 | NAT-2 | VI | VI |
3 | NAT-3 | I, II | III |
4 | NAT-4 | II, V | II, VI |
5 | NAT-5 | II | II |
6 | NAT-10 | II, IV, VI | VI |
7 | NAT-15 | II, V | III, V, VI |
8 | NAT-18 | II, III, VI | I, II |
Обсуждение
Развитие ПМЛ у пациентов с РС, принимающих иммуносупрессивные препараты, такие как натализумаб, требует поиска биологических факторов (предикторов), способствующих риску развития этой серьёзной инфекции головного мозга [42]. Изучение структуры последовательности NCCR JCPyV является одним из таких подходов. В настоящей работе впервые на территории России были проанализированы последовательности NCCR JCPyV и выявлен уникальный паттерн дупликаций сегментов NCCR, не имеющий гомологии последовательностей с NCCR, данные о которых опубликованы ранее.
NCCR представляет собой гипервариабельную регуляторную область JCPyV длиной около 400 п.о., которая содержит большинство элементов, необходимых для инициации ранней и поздней транскрипции и экспрессии вирусных генов, включая TATA-боксы и сайты связывания факторов транскрипции [2]. В составе NCCR «архетипа» JCPyV выделяют 6 сегментов — A, B, C, D, E и F, состоящих из 25, 23, 55, 66, 18 и 69 п.о. соответственно [11]. Нейровирулентные JCPyV NCCR перестроены. Например, «прототип» последовательности NCCR JCPyV, известный как Mad-1, который впервые был выделен у пациента с ПМЛ [12, 38], имеет делеции в сегменте D и состоит только из сегментов A, C и E, расположенных в виде тандемных повторов длиной 98 п.о., за которыми следует сегмент F [43–45]. Существует множество перестроенных изолятов JCPyV [46], в том числе другие Mad-изоляты, которые были получены из тканей пациентов с ПМЛ [47], например Mad-8, по сегментному составу схожий с Mad-1, но имеющий в составе часть сегмента B, а также одиночные инсерции. При этом вариант Mad-8 более распространён среди NCCR, обнаруживаемых у пациентов с ПМЛ, по сравнению с Mad-1 [43, 47, 48].
Известно, что JCPyV NCCR разделены на четыре группы [49]: I-S, I-R (как Mad-1 и Mad-4), II-S и II-R (как Mad-7 и Mad-8). NCCR группы I не содержат инсерций, тогда как NCCR группы II имеют вставку по крайней мере части последовательности из одного из участков сегментов B или D. Подтипы S (от англ. singular) не содержат повторов, тогда как для подтипов R (от англ. repeat) характерны повторы. NCCR типа II-S называют архетипами или архетипоподобными, если они содержат делеции. NCCR типа II-R обычно называют перестроенными NCCR [49].
В ходе нашего исследования показано, что в совокупности до 55% JCPyV NCCR у пациентов с РС, получавших натализумаб, относятся к перестроенным NCCR подтипа II-R, а остальные представляют собой архетипоподобные подтипы II-S. Полученные данные несколько отличаются от опубликованных ранее, где до 80–100% NCCR, идентифицированных в образцах ЦСЖ и/или плазмы/сыворотки крови пациентов, страдающих ПМЛ или получавших натализумаб, относятся к перестроенным NCCR подтипа II-R [31, 50, 51]. Кроме того, анализ последовательностей NCCR у пациентов с РС, получавших натализумаб, выявил только 4 паттерна перестройки NCCR (типы I–IV) и 2 архетипоподобных паттерна (типы V и IV; табл. 2). Эти результаты отличаются от данных, описанных ранее С.Е. Reid и соавт., где каждый пациент с ПМЛ имел уникальный набор делеций и дупликаций в пределах NCCR [50]. Типы I и III представляют собой уникальные перестроенные NCCR, обнаруженные впервые. Варианты NCCR типа IV были описаны ранее у пациентов с синдромом первичного иммунодефицита, ПМЛ и пациентов с РС, получавших натализумаб [50–53]. Укороченные формы NCCR также были описаны ранее у пациентов с РС, получавших натализумаб [50].
NCCR типа II — самый распространённый среди образцов перестроенных NCCR, обнаруженный более чем в 30% случаев (16 из 48), считается редким и встречается лишь в 1,7% (17 из 989) изученных случаев в мире [46]. Интересно, что такого рода NCCR ранее обнаруживали исключительно в ЦСЖ [46], тогда как в нашем исследовании более 80% таких NCCR были представлены в плазме крови больных РС, получавших натализумаб.
Мутации в перестроенных NCCR сложны и, скорее всего, являются результатом гомологичной рекомбинации, которая приводит к большим делециям и тандемным дупликациям сегментов в NCCR [43], как в случае NCCR, обнаруженных в ходе нашей работы.
Сравнительный анализ показал, что фланкирующие сегменты A и F никогда не затрагивались во время перестройки, за исключением NCCR типа III, где часть сегмента F была удалена. Эти наблюдения согласуются с перестройками NCCR, наблюдаемыми у пациентов с ПМЛ, получавших натализумаб [50].
Дублированный сегмент B часто обнаруживался как часть относительно длинных тандемных или одиночных повторов (BCDE или BC в нашем исследовании) в перестроенных JCPyV NCCR у пациентов с РС, получавших натализумаб. Аналогичные дупликации сегмента B (частичные или полноразмерные) были обнаружены в последовательностях NCCR Mad-8 JCPyV, обнаруженных в тканях мозга больных ПМЛ [47], в последовательностях NCCR Mad-8 JCPyV из тканей миндалин детей [40] и в некоторых NCCR у пациентов с ПМЛ, получавших натализумаб [50, 51, 55]. Только NCCR типа IV имел единственную копию сегмента B в своём составе.
Отметим, что сегмент C всегда был дуплицирован в перестроенных JCPyV NCCR исследованной когорты, получавшей натализумаб, либо как часть относительно длинных повторов (BCDE или BC) в NCCR типа I–типа III, либо как одиночная дупликация в NCCR типа IV. Дупликации сегмента C характерны для многих NCCR, обнаруживаемых у пациентов с синдромом первичного иммунодефицита, ПМЛ, и пациентов с РС, получавших натализумаб [50–53].
Интересно, что сегмент D присутствовал во всех NCCR пациентов с РС, получавших натализумаб. Этот факт не согласуется с предыдущими сообщениями, описывающими его полное или частичное отсутствие в NCCR из плазмы крови и/или ЦСЖ пациентов с ПМЛ, получавших натализумаб [46, 50]. Подавляющее большинство NCCR, обнаруженных в ходе исследования, имели идентичную уникальную делецию длиной 6 п.о. (AAACCA) в сегменте D. Эта делеция наблюдалась в 41 из 48 последовательностей NCCR, полученных из образцов плазмы крови и ЦСЖ во всех типах NCCR (как перестроенных, так и архетипоподобных), за исключением укороченных NCCR (7 из 48), принадлежащих к типу V. Важно отметить, что когда эта делеция присутствовала в повторяющемся фрагменте BCDE, она была общей для всех копий, что позволяет предположить, что делеция произошла до дупликации, как предполагалось ранее [50, 55]. Эта уникальная делеция, по-видимому, является вызванным натализумабом изменением в исследуемой когорте (результат избирательного давления, вызванного лечением натализумабом), поскольку она присутствовала в подавляющем большинстве проанализированных последовательностей NCCR, но отсутствовала в NCCR из плазмы и ЦСЖ пациента с первичным иммунодефицитом.
Сегмент E присутствовал во всех NCCR у пациентов с РС, получавших натализумаб. В NCCR типов I и II он был дуплицирован как часть относительно длинного повтора BCDE. Это наблюдение согласуется с ранее описанными структурами NCCR у пациентов с ПМЛ [47, 50, 51, 54]. NCCR типа III имели единственную копию сегмента E.
Преобладание повторов в последовательностях NCCR ведёт к увеличению количества энхансерных элементов, предположительно имеющих решающее значение для вирусного патогенеза [46]. Так, каждый BCDE-фрагмент содержит несколько энхансерных элементов, включая сайты связывания факторов транскрипции NF1, CRE-TAR, GA-домен (сайт связывания фактора транскрипции SP-1), GF-1, AP-1 и др. (табл. 3). Известно, что эти энхансерные элементы модулируют транскрипцию, экспрессию и репликацию JCPyV, а также его тропизм [13, 44, 56–61].
Наш анализ показал, что сегмент D содержит несколько энхансерных элементов, в том числе 4 сайта связывания, TFIID, TBP, TEF-1 и c-Fos, предсказанные онлайн-инструментом PROMO [35, 36]. Уникальная делеция 6 п.о. (AAACCA), обнаруженная в сегменте D, приводила к потере предсказанного сайта связывания c-Fos (прогноз был сделан для человеческого фактора и вирусных сайтов) по сравнению с последовательностью архетипа. c-Fos представляет собой ядерный фосфопротеин, образующий прочный, но нековалентно связанный комплекс с фактором транскрипции JUN/AP-1 [62, 63]. c-Fos экспрессируется с низкой тканевой специфичностью и обнаруживается практически во всех отделах организма2. Известно, что c-Fos активирует ранние и поздние промоторы JCPyV [64], поэтому потеря этого предполагаемого сайта связывания в сегменте D может не привести к переходу к нейровирулентному варианту, но этот факт требует дальнейшего изучения.
Интересным показался тот факт, что в организме 12 пациентов с РС, принимавших натализумаб, были обнаружены множественные варианты NCCR, причём варианты, обнаруженные в плазме крови и ЦСЖ одного пациента, различались. Ранее было показано, что JCPyV в организме человека существует как квазивид (т.е. популяция высокородственных особей), который является общим для ЦСЖ и плазмы крови у одного пациента [53]. Наряду с этим в большей части парных образцов варианты NCCR, обнаруженные в плазме крови и ЦСЖ, отличались. Известно, что клетки костного мозга и происходящие из него В-лимфоциты считаются потенциальными местами для нейротропной трансформации JCPyV из-за присущей способности к перестройке ДНК, которая может способствовать появлению перестроенных вариантов JCPyV [65–67]. Ранее было высказано предположение, что как инфицированные вирусом лимфоциты, так и бесклеточный вирус участвуют в распространении вируса путём преодоления гематоэнцефалического барьера [67–70]. Вероятнее всего, перестройки в JCPyV NCCR в инфицированных лимфоцитах, преодолевших гематоэнцефалический барьер, продолжаются, в связи с чем в ЦСЖ и возникают варианты NCCR, отличающиеся (полностью или частично) от тех, что выявляются в плазме крови.
Смена генотипов JCPyV, которую мы наблюдали в парных образцах двух пациентов (NAT-10 и NAT-15), указывает на то, что в условиях иммуносупрессии организма (на фоне приёма натализумаба) JCPyV высокоизменчив не только в области NCCR, но и в области, кодирующей VP1. Наличие множественных вариантов JCPyV, их различающаяся компартментализация внутри организма пациента, а также смена генотипа JCPyV во времени и в зависимости от проанализированного компартмента организма (плазма крови или ЦСЖ) отражают процессы, в ходе которых вирус непрерывно адаптируется к своей клеточной среде, о чём упоминалось ранее другими исследователями [53].
Заключение
Проведённое исследование позволило впервые выявить паттерны NCCR, характерные для больных РС, принимавших натализумаб, в России, а также впервые выявить новый перестроенный вариант NCCR и продемонстрировало острую необходимость проведения дальнейших исследований перестроек JCPyV NCCR у пациентов с РС, получавших натализумаб, поскольку это может дать новое представление о возникновении нейровирулентных вариантов JCPyV. В перспективе определение перестроек JCPyV NCCR может улучшить стратификацию риска ПМЛ, ускорить своевременную диагностику и оптимизировать ресурсы здравоохранения, способствуя снижению прямых и косвенных затрат на заболевание РС.
Этическое утверждение. Исследование проводилось при добровольном информированном согласии пациентов. Протокол исследования одобрен Этическим комитетом Научного центра неврологии (протокол № 1-2/22 от 19.01.2022).
1 URL: http://alggen.lsi.upc.es/cgi-bin/promo_v3/promo/promoinit.cgi?dirDB = TF_8.3
2 URL: https://www.proteinatlas.org/ENSG00000170345-FOS/tissue
Об авторах
Марина Алексеевна Тюменцева
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: tyumentseva@cmd.su
ORCID iD: 0000-0002-3145-3702
к.б.н., зав. лаб. геномного редактирования отдела молекулярной диагностики и эпидемиологии
Россия, МоскваАлександр Игоревич Тюменцев
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: tyumentseva@cmd.su
ORCID iD: 0000-0003-0537-2586
к.б.н., зав. лаб. экспериментальной фармакологии отдела молекулярной диагностики и эпидемиологии
Россия, МоскваМария Николаевна Захарова
Научный центр неврологии
Email: tyumentseva@cmd.su
ORCID iD: 0000-0002-1072-9968
д.м.н., профессор, г.н.с., рук. 6-го неврологического отделения Института клинической и профилактической неврологии
Россия, МоскваЛола Шавкатовна Аскарова
Научный центр неврологии
Email: tyumentseva@cmd.su
ORCID iD: 0000-0003-1606-7245
к.м.н., с.н.с. 6-го неврологического отделения Института клинической и профилактической неврологии
Россия, МоскваТарас Олегович Симанив
Научный центр неврологии
Email: tyumentseva@cmd.su
ORCID iD: 0000-0001-7256-2668
к.м.н., с.н.с. 6-го неврологического отделения Института клинической и профилактической неврологии
Россия, МоскваМихаил Александрович Пирадов
Научный центр неврологии
Email: tyumentseva@cmd.su
ORCID iD: 0000-0002-6338-0392
д.м.н., профессор, академик РАН, директор
Россия, МоскваВасилий Геннадиевич Акимкин
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Автор, ответственный за переписку.
Email: tyumentseva@cmd.su
ORCID iD: 0000-0003-4228-9044
д.м.н., профессор, академик РАН, директор
Россия, МоскваСписок литературы
- Wharton K.A. Jr., Quigley C., Themeles M., Dunstan R.W., Doyle K., Cahir-McFarland E., et al. JC Polyomavirus abundance and distribution in progressive multifocal leukoencephalopathy (PML) brain tissue implicates myelin sheath in intracerebral dissemination of infection. PLoS One. 2016; 11(5): e0155897. https://doi.org/10.1371/journal.pone.0155897
- Marshall L.J., Major E.O. Molecular regulation of JC virus tropism: insights into potential therapeutic targets for progressive multifocal leukoencephalopathy. J. Neuroimmune. Pharmacol. 2010; 5(3): 404–17. https://doi.org/10.1007/s11481-010-9203-1
- Frisque R.J. Structure and function of JC virus T’ proteins. J. Neurovirol. 2001; 7(4): 293–7. https://doi.org/10.1080/13550280152537120
- Prins C., Frisque R.J. JC virus T’ proteins encoded by alternatively spliced early mRNAs enhance T antigen-mediated viral DNA replication in human cells. J. Neurovirol. 2001; 7(3): 250–64. https://doi.org/10.1080/13550280152403290
- Gasparovic M.L., Gee G.V., Atwood W.J. JC virus minor capsid proteins Vp2 and Vp3 are essential for virus propagation. J. Virol. 2006; 80(21): 10858–61. https://doi.org/10.1128/JVI.01298-06
- Del Valle L., Piña-Oviedo S. HIV disorders of the brain: pathology and pathogenesis. Front. Biosci. 2006; 11(1): 718–32. https://doi.org/10.2741/1830
- Major E.O. Progressive multifocal leukoencephalopathy in patients on immunomodulatory therapies. Annu. Rev. Med. 2010; 61(1): 35–47. https://doi.org/10.1146/annurev.med.080708.082655
- White M.K., Khalili K. Pathogenesis of progressive multifocal leukoencephalopathy – revisited. J. Infect. Dis. 2011; 203(5): 578–86. https://doi.org/10.1093/infdis/jiq097
- Delbue S., Ferraresso M., Ghio L., Carloni C., Carluccio S., Belingheri M., et al. A review on JC virus infection in kidney transplant recipients. Clin. Dev. Immunol. 2013; 2013: 926391. https://doi.org/10.1155/2013/926391
- Kmieciak D., Debicki S., Trzeciak W.H. Occurrence rate and genotype distribution of the JC virus (JCV) in a sample from the Polish population. J. Med. Virol. 2008; 80(6): 1079–83. https://doi.org/10.1002/jmv.21153
- Yogo Y., Kitamura T., Sugimoto C., Ueki T., Aso Y., Hara K., et al. Isolation of a possible archetypal JC virus DNA sequence from nonimmunocompromised individuals. J. Virol. 1990; 64(6): 3139–43. https://doi.org/10.1128/JVI.64.6.3139-3143.1990
- Frisque R.J., Bream G.L., Cannella M.T. Human polyomavirus JC virus genome. J. Virol. 1984; 51(2): 458–69. https://doi.org/10.1128/JVI.51.2.458-469.1984
- Marshall L.J., Dunham L., Major E.O. Transcription factor Spi-B binds unique sequences present in the tandem repeat promoter/enhancer of JC virus and supports viral activity. J. Gen. Virol. 2010; 91(Pt. 12): 3042–52. https://doi.org/10.1099/vir.0.023184-0
- Gosert R., Kardas P., Major E.O., Hirsch H.H. Rearranged JC virus noncoding control regions found in progressive multifocal leukoencephalopathy patient samples increase virus early gene expression and replication rate. J. Virol. 2010; 84(20): 10448–56. https://doi.org/10.1128/JVI.00614-10
- Daniel A.M., Swenson J.J., Mayreddy R.P., Khalili K., Frisque R.J. Sequences within the early and late promoters of archetype JC virus restrict viral DNA replication and infectivity. Virology. 1996; 216(1): 90–101. https://doi.org/10.1006/viro.1996.0037
- McGuigan C., Craner M., Guadagno J., Kapoor R., Mazibrada G., Molyneux P., et al. Stratification and monitoring of natalizumab-associated progressive multifocal leukoencephalopathy risk: recommendations from an expert group. J. Neurol. Neurosurg. Psychiatry. 2016; 87(2): 117–25. https://doi.org/10.1136/jnnp-2015-311100
- Carson K.R., Evens A.M., Richey E.A., Habermann T.M., Focosi D., Seymour J.F., et al. Progressive multifocal leukoencephalopathy after rituximab therapy in HIV-negative patients: a report of 57 cases from the Research on Adverse Drug Events and Reports project. Blood. 2009; 113(20): 4834–40. https://doi.org/10.1182/blood-2008-10-186999
- Diebold M., Altersberger V., Décard B.F., Kappos L., Derfuss T., Lorscheider J. A case of progressive multifocal leukoencephalopathy under dimethyl fumarate treatment without severe lymphopenia or immunosenescence. Mult. Scler. 2019; 25(12): 1682–5. https://doi.org/10.1177/1352458519852100
- Berger J.R., Cree B.A., Greenberg B., Hemmer B., Ward B.J., Dong V.M., et al. Progressive multifocal leukoencephalopathy after fingolimod treatment. Neurology. 2018; 90(20): e1815–21. https://doi.org/10.1212/wnl.0000000000005529
- Schwab N., Ulzheimer J.C., Fox R.J., Schneider-Hohendorf T., Kieseier B.C., Monoranu C.M., et al. Fatal PML associated with efalizumab therapy: insights into integrin αLβ2 in JC virus control. Neurology. 2012; 78(7): 458–67; discussion 465. https://doi.org/10.1212/WNL.0b013e3182478d4b
- Carson K.R., Newsome S.D., Kim E.J., Wagner-Johnston N.D., von Geldern G., Moskowitz C.H., et al. Progressive multifocal leukoencephalopathy associated with brentuximab vedotin therapy: A report of 5 cases from the Southern Network on Adverse Reactions (SONAR) project: Brentuximab-Associated PML. Cancer. 2014; 120(16): 2464–71. https://doi.org/10.1002/cncr.28712
- Vukusic S., Rollot F., Casey R., Pique J., Marignier R., Mathey G., et al. Progressive multifocal leukoencephalopathy incidence and risk stratification among natalizumab users in France. JAMA Neurol. 2020; 77(1): 94–102. https://doi.org/10.1001/jamaneurol.2019.2670
- Campagnolo D., Dong Q., Lee L., Ho P.R., Amarante D., Koendgen H. Statistical analysis of PML incidences of natalizumab-treated patients from 2009 to 2016: outcomes after introduction of the Stratify JCV® DxSelectTM antibody assay. J. Neurovirol. 2016; 22(6): 880–1. https://doi.org/10.1007/s13365-016-0482-z
- Datapharm. Physician information and management guidelines for patients with multiple sclerosis receiving TYSABRI therapy. Available at: https://www.medicines.org.uk/emc/rmm/2196/Document
- Gorelik L., Lerner M., Bixler S., Crossman M., Schlain B., Simon K., et al. Anti-JC virus antibodies: implications for PML risk stratification: Anti-JCV Antibodies. Ann. Neurol. 2010; 68(3): 295–303. https://doi.org/10.1002/ana.22128
- Viscidi R.P., Khanna N., Tan C.S., Li X., Jacobson L., Clifford D.B., et al. JC virus antibody and viremia as predictors of progressive multifocal leukoencephalopathy in human immunodeficiency virus-1-infected individuals. Clin. Infect. Dis. 2011; 53(7): 711–5. https://doi.org/10.1093/cid/cir507
- Bloomgren G., Richman S., Hotermans C., Subramanyan M., Goelz S., Natarajam A., et al. Risk of natalizumab-associated progressive multifocal leukoencephalopathy. Surv. Anesthesiol. 2012; 56(6): 329–30. https://doi.org/10.1097/01.sa.0000422027.26626.8d
- Sindic C.J., Trebst C., Van Antwerpen M.P., Frye S., Enzensberger W., Hunsmann G., et al. Detection of CSF-specific oligoclonal antibodies to recombinant JC virus VP1 in patients with progressive multifocal leukoencephalopathy. J. Neuroimmunol. 1997; 76(1-2): 100–4. https://doi.org/10.1016/s0165-5728(97)00037-4
- Khanna N., Wolbers M., Mueller N.J., Garzoni C., Du Pasquier R.A., Fux C.A., et al. JC virus-specific immune responses in human immunodeficiency virus type 1 patients with progressive multifocal leukoencephalopathy. J. Virol. 2009; 83(9): 4404–11. https://doi.org/10.1128/JVI.02657-08
- Prezioso C., Zingaropoli M.A., Iannetta M., Rodio D.M., Altieri M., Conte A., et al. Which is the best PML risk stratification strategy in natalizumab-treated patients affected by multiple sclerosis? Mult. Scler. Relat. Disord. 2020; 41(102008): 102008. https://doi.org/10.1016/j.msard.2020.102008
- L’Honneur A.S., Leh H., Laurent-Tchenio F., Hazan U., Rozenberg F., Bury-Moné S. Exploring the role of NCCR variation on JC polyomavirus expression from dual reporter minicircles. PLoS One. 2018; 13(6): e0199171. https://doi.org/10.1371/journal.pone.0199171
- Kato A., Sugimoto C., Zheng H.Y., Kitamura T., Yogo Y. Lack of disease-specific amino acid changes in the viral proteins of JC virus isolates from the brain with progressive multifocal leukoencephalopathy. Arch. Virol. 2000; 145(10): 2173–82. https://doi.org/10.1007/s007050070047
- Pagani E., Delbue S., Mancuso R., Borghi E., Tarantini L., Ferrante P. Molecular analysis of JC virus genotypes circulating among the Italian healthy population. J. Neurovirol. 2003; 9(5): 559–66. https://doi.org/10.1080/13550280390241269
- Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Mol. Biol. Evol. 2016; 33(7): 1870–4. https://doi.org/10.1093/molbev/msw054
- Messeguer X., Escudero R., Farré D., Núñez O., Martínez J., Albà M.M. PROMO: detection of known transcription regulatory elements using species-tailored searches. Bioinformatics. 2002; 18(2): 333–4. https://doi.org/10.1093/bioinformatics/18.2.333
- Farré D., Roset R., Huerta M., Adsuara J.E., Roselló L., Albà M.M., et al. Identification of patterns in biological sequences at the ALGGEN server: PROMO and MALGEN. Nucleic. Acids. Res. 2003; 31(13): 3651–3. https://doi.org/10.1093/nar/gkg605
- Hirsch H.H., Kardas P., Kranz D., Leboeuf C. The human JC polyomavirus (JCPyV): virological background and clinical implications. APMIS. 2013; 121(8): 685–727. https://doi.org/10.1111/apm.12128
- Padgett B., Zurhein G., Walker D., Eckroade R., Dessel B. Cultivation of papova-like virus from human brain with progressive multifocal leucoencephalopathy. Lancet. 1971; 297(7712): 1257–60. https://doi.org/10.1016/s0140-6736(71)91777-6
- Marshall L.J., Moore L.D., Mirsky M.M., Major E.O. JC virus promoter/enhancers contain TATA box-associated Spi-B-binding sites that support early viral gene expression in primary astrocytes. J. Gen. Virol. 2012; 93(Pt. 3): 651–61. https://doi.org/10.1099/vir.0.035832-0
- Monaco M.C., Jensen P.N., Hou J., Durham L.C., Major E.O. Detection of JC virus DNA in human tonsil tissue: evidence for site of initial viral infection. J. Virol. 1998; 72(12): 9918–23. https://doi.org/10.1128/JVI.72.12.9918-9923.1998
- Naess H., Glad S., Storstein A., Rinaldo C.H., Mørk S.J., Myhr K.M., et al. Progressive multifocal leucoencephalopathy in an immunocompetent patient with favourable outcome. A case report. BMC Neurol. 2010; 10: 32. https://doi.org/10.1186/1471-2377-10-32
- Pavlovic D., Patera A.C., Nyberg F., Gerber M., Liu M. Progressive Multifocal Leukeoncephalopathy Consortium. Progressive multifocal leukoencephalopathy: current treatment options and future perspectives. Ther. Adv. Neurol. Disord. 2015; 8(6): 255–73. https://doi.org/10.1177/1756285615602832
- Cortese I., Reich D.S., Nath A. Progressive multifocal leukoencephalopathy and the spectrum of JC virus-related disease. Nat. Rev. Neurol. 2021; 17(1): 37–51. https://doi.org/10.1038/s41582-020-00427-y
- Ferenczy M.W., Marshall L.J., Nelson C.D.S., Atwood W.J., Nath A., Khalili K., et al. Molecular biology, epidemiology, and pathogenesis of progressive multifocal leukoencephalopathy, the JC virus-induced demyelinating disease of the human brain. Clin. Microbiol. Rev. 2012; 25(3): 471–506. https://doi.org/10.1128/CMR.05031-11
- Ciardi M.R., Zingaropoli M.A., Iannetta M., Prezioso C., Perri V., Pasculli P., et al. JCPyV NCCR analysis in PML patients with different risk factors: exploring common rearrangements as essential changes for neuropathogenesis. Virol. J. 2020; 17(1): 23. https://doi.org/10.1186/s12985-020-1295-5
- Wilczek M.P., Pike A.M.C., Craig S.E., Maginnis M.S., King B.L. Rearrangement in the hypervariable region of JC Polyomavirus genomes isolated from patient samples and impact on transcription factor-binding sites and disease outcomes. Int. J. Mol. Sci. 2022; 23(10): 5699. https://doi.org/10.3390/ijms23105699
- Martin J.D., King D.M., Slauch J.M., Frisque R.J. Differences in regulatory sequences of naturally occurring JC virus variants. J. Virol. 1985; 53(1): 306–11. https://doi.org/10.1128/JVI.53.1.306-311.1985
- Martin J.D., Foster G.C. Multiple JC virus genomes from one patient. J. Gen. Virol. 1984; 65(Pt. 8): 1405–11. https://doi.org/10.1099/0022-1317-65-8-1405
- Jensen P.N., Major E.O. A classification scheme for human polyomavirus JCV variants based on the nucleotide sequence of the noncoding regulatory region. J. Neurovirol. 2001; 7(4): 280–7. https://doi.org/10.1080/13550280152537102
- Reid C.E., Li H., Sur G., Carmillo P., Bushnell S., Tizard R., et al. Sequencing and analysis of JC virus DNA from natalizumab-treated PML patients. J. Infect. Dis. 2011; 204(2): 237–44. https://doi.org/10.1093/infdis/jir256
- Marshall L.J., Ferenczy M.W., Daley E.L., Jensen P.N., Ryschkewitsch C.F., Major E.O. Lymphocyte gene expression and JC virus noncoding control region sequences are linked with the risk of progressive multifocal leukoencephalopathy. J. Virol. 2014; 88(9): 5177–83. https://doi.org/10.1128/JVI.03221-13
- Nakamichi K., Tajima S., Lim C.K., Saijo M. High-resolution melting analysis for mutation scanning in the non-coding control region of JC polyomavirus from patients with progressive multifocal leukoencephalopathy. Arch. Virol. 2014; 159(7): 1687–96. https://doi.org/10.1007/s00705-014-1988-4
- Van Loy T., Thys K., Ryschkewitsch C., Lagatie O., Monaco M.C., Major E.O., et al. JC virus quasispecies analysis reveals a complex viral population underlying progressive multifocal leukoencephalopathy and supports viral dissemination via the hematogenous route. J. Virol. 2015; 89(2): 1340–7. https://doi.org/10.1128/jvi.02565-14
- Muiño E., Rubio M.A., Navalpotro I., Munteis E. Progressive multifocal leukoencephalopathy in an immunocompetent patient. Neurologia. 2017; 32(5): 337–9. https://doi.org/10.1016/j.nrleng.2015.08.011
- Johnson E.M., Wortman M.J., Dagdanova A.V., Lundberg P.S., Daniel D.C. Polyomavirus JC in the context of immunosuppression: a series of adaptive, DNA replication-driven recombination events in the development of progressive multifocal leukoencephalopathy. Clin. Dev. Immunol. 2013; 2013: 197807. https://doi.org/10.1155/2013/197807
- Kumar K.U., Devireddy L.R., Tang S.C., Pater A., Pater M.M. Human JC virus nuclear factor 1 binding motifs and large tumor antigen region required for transactivation of late promoter. J. Neurochem. 2002; 67(2): 473–81. https://doi.org/10.1046/j.1471-4159.1996.67020473.x
- Mason S., Piper M., Gronostajski R.M., Richards L.J. Nuclear factor one transcription factors in CNS development. Mol. Neurobiol. 2009; 39(1): 10–23. https://doi.org/10.1007/s12035-008-8048-6
- Rodio D.M., Anzivino E., Mischitelli M., Bellizzi A., Scrivo R., Scribano D., et al. Increased prevalence of human Polyomavirus JC viruria in chronic inflammatory rheumatic diseases patients in treatment with anti-TNF α: A 18 month follow-up study. Front. Microbiol. 2016; 7: 672. https://doi.org/10.3389/fmicb.2016.00672
- Sumner C., Shinohara T., Durham L., Traub R., Major E.O., Amemiya K. Expression of multiple classes of the nuclear factor-1 family in the developing human brain: differential expression of two classes of NF-1 genes. J. Neurovirol. 1996; 2(2): 87–100. https://doi.org/10.3109/13550289609146542
- Ravichandran V., Sabath B.F., Jensen P.N., Houff S.A., Major E.O. Interactions between c-Jun, nuclear factor 1, and JC virus promoter sequences: Implications for viral tropism. J. Virol. 2006; 80(21): 10506–13. https://doi.org/10.1128/jvi.01355-06
- Chen N.N., Kerr D., Chang C.F., Honjo T., Khalili K. Evidence for regulation of transcription and replication of the human neurotropic virus JCV genome by the human S(mu)bp-2 protein in glial cells. Gene. 1997; 185(1): 55–62. https://doi.org/10.1016/s0378-1119(96)00630-0
- Angel P., Karin M. The role of Jun, Fos and the AP-1 complex in cell-proliferation and transformation. Biochim. Biophys. Acta. Rev. Cancer. 1991; 1072(2-3): 129–57. https://doi.org/10.1016/0304-419x(91)90011-9
- Wisdom R. AP-1: one switch for many signals. Exp. Cell. Res. 1999; 253(1): 180–5. https://doi.org/10.1006/excr.1999.4685
- Sadowska B., Barrucco R., Khalili K., Safak M. Regulation of human Polyomavirus JC virus gene transcription by AP-1 in glial cells. J. Virol. 2003; 77(1): 665–72. https://doi.org/10.1128/jvi.77.1.665-672.2003
- Houff S.A., Major E.O., Katz D.A., Kufta C.V., Sever J.L., Pittaluga S., et al. Involvement of JC virus-infected mononuclear cells from the bone marrow and spleen in the pathogenesis of progressive multifocal leukoencephalopathy. N. Engl. J. Med. 1988; 318(5): 301–5. https://doi.org/10.1056/NEJM198802043180507
- Marzocchetti A., Wuthrich C., Tan C.S., Tompkins T., Bernal-Cano F., Bhargava P., et al. Rearrangement of the JC virus regulatory region sequence in the bone marrow of a patient with rheumatoid arthritis and progressive multifocal leukoencephalopathy. J. Neurovirol. 2008; 14(5): 455–8. https://doi.org/10.1080/13550280802356837
- Monaco M.C., Atwood W.J., Gravell M., Tornatore C.S., Major E.O. JC virus infection of hematopoietic progenitor cells, primary B lymphocytes, and tonsillar stromal cells: implications for viral latency. J. Virol. 1996; 70(10): 7004–12. https://doi.org/10.1128/JVI.70.10.7004-7012.1996
- Chapagain M.L., Verma S., Mercier F., Yanagihara R., Nerurkar V.R. Polyomavirus JC infects human brain microvascular endothelial cells independent of serotonin receptor 2A. Virology. 2007; 364(1): 55–63. https://doi.org/10.1016/j.virol.2007.02.018
- Chapagain M.L., Nerurkar V.R. Human polyomavirus JC (JCV) infection of human B lymphocytes: a possible mechanism for JCV transmigration across the blood-brain barrier. J. Infect. Dis. 2010; 202(2): 184–91. https://doi.org/10.1086/653823
- Tornatore C., Berger J.R., Houff S.A., Curfman B., Meyers K., Winfield D., et al. Detection of JC virus DNA in peripheral lymphocytes from patients with and without progressive multifocal leukoencephalopathy. Ann. Neurol. 1992; 31(4): 454–62. https://doi.org/10.1002/ana.410310426
Дополнительные файлы