Протективный потенциал структурных белков вируса SARS-CoV-2 в защите от COVID-19
- Авторы: Должикова И.В.1, Гроусова Д.М.1, Зорков И.Д.1, Илюхина А.А.1, Ковыршина А.В.1, Зубкова О.В.1, Попова О.Д.1, Ожаровская Т.А.1, Зрелкин Д.И.1, Савина Д.М.1, Самохвалова Е.Г.1, Тухватулин А.И.1, Щебляков Д.В.1, Логунов Д.Ю.1, Гинцбург А.Л.1,2
-
Учреждения:
- Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
- Первый Московский государственный медицинский университет имени И.М. Сеченова (Сеченовский Университет)
- Выпуск: Том 101, № 6 (2024)
- Страницы: 769-778
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/18637
- DOI: https://doi.org/10.36233/0372-9311-577
- EDN: https://elibrary.ru/wrxlsr
- ID: 18637
Цитировать
Аннотация
Введение. Множество различных вакцин для профилактики COVID-19 в кратчайшие сроки получили разрешение на экстренное применение. В связи с высоким уровнем изменчивости возбудителя, важно учитывать вариабельность структурных белков вируса SARS-CoV-2 и их протективный потенциал в защите животных от COVID-19.
Цель исследования — сравнить протективный потенциал структурных белов вируса SARS-CoV-2 в защите животных от COVID-19.
Материалы и методы. В качестве модельных животных при исследовании вируса SARS-CoV-2 служили трансгенные мыши B6.Cg-Tg(K18-ACE2)2Prlmn/J (F1). Для иммунизации животных использовали препараты рекомбинантных аденовирусных векторов: rAd5-S, rAd5-N, rAd5-M. В работе применяли различные генетические, вирусологические и иммунологические методы, а также методы работы с животными.
Результаты. Наибольшее количество аминокислотных замен в структурных белках разных вариантов SARS-CoV-2 было обнаружено в гликопротеине S, наименьшее — в нуклеопротеине N. На модели COVID-19 у животных показано, что только использование гликопротеина S в качестве антигена в составе вакцинного препарата позволяет сформировать протективный иммунитет, который защищает 100% животных от летальной инфекции, вызванной вирусом SARS-CoV-2, при этом использование белка N позволяет защитить 50% животных от летальной инфекции, а белок М не обладает протективным потенциалом.
Заключение. Полученные данные, а также анализ данных эпидемиологической эффективности разных мРНК- и векторных вакцин демонстрируют, что использование гликопротеина S вируса SARS-CoV-2 в качестве антигена позволяет сформировать наиболее высокий уровень защиты. Учитывая постоянную смену циркулирующих вариантов вируса SARS-CoV-2, снижение эффективности используемых вакцин с исходным антигенным составом в отношении новых вариантов вируса и сохраняющийся высокий уровень заболеваемости COVID-19, необходимо проводить непрерывный мониторинг эффективности вакцинных препаратов в отношении новых вариантов вируса и своевременно проводить актуализацию антигенного состава вакцин при выявлении снижения эффективности.
Ключевые слова
Полный текст
Введение
С момента идентификации возбудителя COVID-19 — вируса SARS-CoV-2 — разработка вакцин идёт беспрецедентными и необычайными темпами благодаря совместным усилиям исследователей, промышленности и правительств разных стран. Это позволило в кратчайшие сроки получить разрешение на экстренное применение вакцин и внедрение их в гражданский оборот для вакцинации населения по всему миру.
Вирус SARS-CoV-2 относится к одноцепочечным (+)РНК-содержащим вирусам рода Betacoronavirus семейства Coronaviridae, размер генома варьирует от 29,8 до 29,9 тыс. нуклеотидов, вирионы имеют сферическую или эллипсоидальную форму со средним диаметром 108 ± 8 нм и содержат 4 основных структурных белка. Внешняя поверхность вириона покрыта поверхностным белком — гликопротеином S, наружная мембрана вируса содержит мембранный белок М и оболочечный белок Е. Внутри вириона находится рибонуклеопротеиновый комплекс, состоящий из нуклеокапсидного белка (N) и РНК вируса [1]. Поверхностный гликопротеин S вируса SARS-CoV-2 представляет собой тример с молекулярной массой около 600 кДа. Расположенный на внешней оболочке вириона, он играет критическую роль в вирусной инфекции благодаря распознаванию рецепторов и последующему слиянию мембран вируса и клетки-хозяина. Было показано, что S вызывает выраженный иммунный ответ, что делает его доминантной мишенью для разработки вакцин для профилактики COVID-19 [2–5]. Основным белковым компонентом внутри вириона является нуклеокапсидный белок N, который необходим для связывания и упаковки геномной РНК в рибонуклеопротеидный комплекс (RNP-комплекс) внутри вириона [6]. Мембранный белок коронавируса (M) является наиболее распространённым вирусным структурным белком и играет центральную роль в сборке и морфогенезе вируса [7]. Белки M и N потенциально могут служить мишенями для включения в состав кандидатных вакцинных препаратов для профилактики COVID-19 [8]. В дополнение к этим структурным белкам SARS-CoV-2 кодирует 16 неструктурных белков (nsp1–16) и 9 вспомогательных белков.
На сегодняшний день зарегистрировано около 50 вакцинных препаратов для профилактики COVID-191. Наиболее широкое распространение получили вакцины, созданные на 4 технологических платформах: векторные вакцины, мРНК-вакцины, инактивированные вакцины и субъединичные вакцины. Наибольшей эффективностью в защите от COVID-19 обладают векторные и мРНК-вакцины [9]. В состав большинства препаратов входит ключевой протективный антиген — поверхностный гликопротеин S вируса SARS-CoV-2. Иммунизация такими препаратами позволяет сформировать полноценный иммунный ответ к белку [10]: формируется как гуморальный иммунный ответ, препятствующий проникновению вируса в клетку, так и клеточный цитотоксический иммунный ответ, необходимый для элиминации заражённых клеток [11–15]. При иммунизации инактивированными вакцинами развивается более слабый гуморальный иммунный ответ и почти отсутствует Т-клеточный иммунный ответ. При этом антитела классов IgG, IgM и IgA формируются к различным белкам SARS-CoV-2, а не только к S [16, 17]. Вопрос иммунных коррелятов защиты от COVID-19 ещё изучается, при этом показана прямая корреляция между уровнем вируснейтрализующих антител и защитой от заболевания [18]. При этом большинство эпитопов для нейтрализующих антител находятся в области рецептор-связывающего домена и N-концевого домена гликопротеина S [19].
Вирус SARS-CoV-2 с момента проникновения в популяцию людей активно эволюционирует [20]. На протяжении 4 лет пандемии COVID-19 происходила постоянная смена циркулирующих вариантов возбудителя, что сопровождалось подъёмами заболеваемости. Самый значительный подъём был зарегистрирован в начале 2022 г. и был связан с распространением первых сублиний (ВА.1 и ВА.2) варианта Omicron вируса SARS-CoV-22. Появление и распространение новых сублиний варианта Omicron (ВА.1, ВА.5, ХВВ, ВА.2.86 и др.) сопровождались подъёмами заболеваемости по всему миру, в том числе среди вакцинированного населения, в связи с чем критически важным является проведение постоянного мониторинга эффективности вакцинных препаратов в отношении циркулирующих вариантов для своевременной смены антигенного состава вакцин3.
В русскоязычной литературе нет данных по сравнительному анализу протективной эффективности различных антигенов вируса SARS-CoV-2. Эти данные позволят адекватно оценить необходимость включения в состав вакцинных препаратов для профилактики COVID-19 различных антигенов для наибольшей эффективности в отношении разных вариантов вируса SARS-CoV-2.
С учетом высокого уровня изменчивости вируса, целью данного исследования являлось сравнение протективного потенциала структурных белов вируса SARS-CoV-2 в защите животных от COVID-19. Для достижения данной цели были поставлены следующие задачи: проанализировать вариабельность структурных белков вируса SARS-CoV-2, провести исследование протективной эффективности различных структурных белков вируса SARS-CoV-2 на модели COVID-19 у лабораторных животных.
Материалы и методы
Вирус
Вирус SARS-CoV-2 был получен из Государственной коллекции вирусов НИЦЭМ им. Н.Ф. Гамалеи: Ухань B.1.1.1 hCoV-19/Russia/Moscow_PMVL-1/2020. Все исследования с жизнеспособным вирусом SARS-CoV-2 проводили в помещениях класса BSL-3 согласно СанПиН 3.3686-21 «Санитарно-эпидемиологические требования по профилактике инфекционных болезней». Наработку вируса SARS-CoV-2 проводили в культуре клеток Vero E6. Титр инфекционного вируса определяли на культуре клеток Vero E6 по 50% инфекционной дозе для тканевой культуры (tissue culture infectious dose 50, TCID50). Титр TCID50 рассчитывали по методу Спирмена–Кербера.
Линии клеток млекопитающих
Культура клеток Vero E6 (клетки эпителия почки африканской зелёной мартышки) была получена из лаборатории клеточной микробиологии НИЦЭМ им. Н.Ф. Гамалеи.
Животные
В исследовании использовали трансгенных мышей F1, полученных от скрещивания трансгенных самцов B6.Cg-Tg(K18-ACE2)2Prlmn/J (Jackson Laboratory, https://www.jax.org/strain/034860; статус здоровья SOPF) и нетрансгенных самок C57BL/6 Gamrc (НИЦЭМ им. Н.Ф. Гамалеи; статус здоровья SPF), далее — hАСЕ2-трансгенные мыши. В исследовании использовали самок hАСЕ2-трансгенных мышей массой 18–20 г. Авторы подтверждают соблюдение институциональных и национальных стандартов по использованию лабораторных животных в соответствии с «Consensus Author Guidelines for Animal Use» (IAVES, 23.07.2010). Протокол исследования одобрен Комитетом по биомедицинской этике НИЦЭМ им. Н.Ф. Гамалеи (протокол № 24 от 21.04.2022). Наличие гена hACE2 у мышей линии С57BL/6 Tg(K18-ACE2)2Prlmn поколения F1 определяли с помощью полимеразной цепной реакции в реальном времени по протоколу Jackson Laboratory для данной линии мышей [21].
Работа с животными
Содержание и работу с животными осуществляли в соответствии с требованиями по содержанию лабораторных животных4. Лабораторных животных (n = 40) размещали в клетках для конвенционального содержания на время иммунизации; для проведения экспериментов, связанных с использованием вируса SARS-CoV-2, животных помещали в систему «IsoCage N» («Tecniplast»). Животные имели свободный доступ к воде и корму.
Иммунизация и заражение животных
Для иммунизации животных использовали препараты рекомбинантных вирусных векторов на основе аденовируса человека 5-го серотипа (rAd5), несущие гены структурных белков вируса SARS-CoV-2 варианта Ухань B.1: rAd5-S (несёт ген гликопротеина S), rAd5-N (несёт ген нуклеопротеина N), rAd5-M (несёт ген мембранного белка М). Животным групп (по 10 особей в группе), получавших вакцину, вводили препараты rAd5 в дозе 109 в.ч./животное внутримышечно двукратно с интервалом 21 сут. Животным контрольной группы (n = 10) вводили эквивалентный объём стерильного буферного раствора. Через неделю после 2-й иммунизации животных заражали интраназально вирусом SARS-CoV-2 в дозе 105 TCID50 и в течение 14 дней после заражения ежедневно оценивали динамику массы тела и выживаемость.
Отбор органов и определение вирусной нагрузки
По 4 животных каждой группы подвергали эвтаназии на 4-й день после заражения с помощью повышенной дозы ингаляционного анестетика с последующей цервикальной дислокацией. Проводили вскрытие животных, отбирали лёгкие для макроскопического анализа и анализа вирусной нагрузки. Отобранные органы промывали физиологическим раствором и готовили 10% гомогенат с использованием прибора «MPbio FastPrep-24». Гомогенаты центрифугировали при 12 000g в течение 10 мин, надосадочную жидкость использовали для дальнейшего анализа. Инфекционный титр вируса определяли на клетках Vero E6 по методике, описанной выше.
Статистические и биоинформатические методы
Статистическую обработку результатов исследований проводили с помощью компьютерной программы «GraphPad Prism 10.2.3». При анализе данных использовали t-критерий Стьюдента [22]. При анализе аминокислотных последовательностей структурных белков вируса SARS-CoV-2 была использована онлайн-база данных covSPECTRUM5. На портале cov-spectrum.org в режиме Compare variants проводили попарное сравнение новых вариантов вируса SARS-CoV-26 с исходным вариантом вируса7, в разделе Amino acid changes выбирали последовательность нужного белка. Для расчёта вариабельности аминокислотного состава количество детектируемых аминокислотных замен делили на общее количество аминокислотных остатков и выражали в процентах.
Результаты
Вариабельность структурных антигенов
Нами проведён биоинформатический анализ аминокислотных последовательностей структурных белков вируса SARS-CoV-2 различных вариантов, которые циркулировали на территории России с марта 2020 г. по июль 2024 г. (таблица). Среди 4 анализируемых белков наибольшее количество аминокислотных замен было обнаружено в гликопротеине S, наименьшее — в нуклеопротеине N. Так, в варианте KS.1, который циркулирует в России с весны 2024 г., в гликопротеине S выявлены 64 аминокислотные замены, а в нуклеопротеине N — 8.
Вариабельность аминокислотного состава структурных белков вируса SARS-CoV-2, циркулировавшего на территории России в 2020–2024 гг.
Variability of amino acid composition of structural proteins of SARS-CoV-2 virus circulating in Russia in 2020–2024
Вариант SARS-CoV-2 SARS-CoV-2 variant | Вариабельность аминокислотного состава, % относительно исходного варианта вируса Variability of amino acid composition, % relative to the original virus variant | |||
S | N | M | E | |
Delta | 0,79 | 0,59 | 0,45 | 0,00 |
Omicron BA.1 | 2,83 | 0,88 | 1,35 | 1,33 |
Omicron BA.5 | 2,67 | 1,03 | 1,35 | 1,33 |
Omicron XBB | 3,22 | 1,03 | 0,90 | 2,67 |
Omicron BA.2.86 | 4,63 | 1,17 | 2,25 | 1,33 |
Omicron KS.1 | 5,03 | 1,17 | 2,25 | 1,33 |
Источник: онлайн-база данных covSPECTRUM.
Source: covSPECTRUM online database.
Защитная эффективность структурных белков вируса SARS-CoV-2 на модели летальной инфекции у АСЕ2-трансгенных мышей
С целью сравнения защитной эффективности структурных белков вируса SARS-CoV-2 нами были получены рекомбинантные вирусные векторы на основе аденовируса человека 5-го серотипа, несущие гены гликопротеина S, нуклеопротеина N или мембранного белка М. Исследование проводили на hАСЕ2-трансгенных мышах. Животных вакцинировали двукратно с интервалом 21 сут и через 7 дней после 2-го введения препаратов животных заражали интраназально вирусом SARS-CoV-2 (Ухань-подобный) в дозе 105 TCID50 на животное. Анализ эффективности антигенов в защите от инфекции проводили по нескольким параметрам: летальность, тяжесть течения инфекции (снижение веса) и снижение вирусной нагрузки в лёгких вакцинированных животных в сравнении с контролем.
Анализ выживаемости показал, что только использование гликопротеина S в качестве антигена позволяет защитить 100% животных от летальной инфекции, вызванной вирусом SARS-CoV-2 (рис. 1, а). Анализ тяжести течения инфекции также продемонстрировал, что только использование гликопротеина S позволяет защитить 100% животных от инфицирования (рис. 1, б).
Рис. 1. Выживаемость (а) и динамика массы (б) hАСЕ2-трансгенных мышей, вакцинированных и контрольной групп (n = 10 — 0–4-й дни; n = 6 — 5–14-й дни) после заражения вирусом SARS-CoV-2.
На фрагменте б отмечено среднее и стандартное отклонение для каждой временнóй точки.
Fig. 1. Survival (a) and weight dynamics (b) of hACE2-transgenic mice from immunized and control groups (n = 10, days 0–4; n = 6, days 5–14) after infection with the SARS-CoV-2 virus.
The mean and standard deviation for each time point are shown in the figure (b).
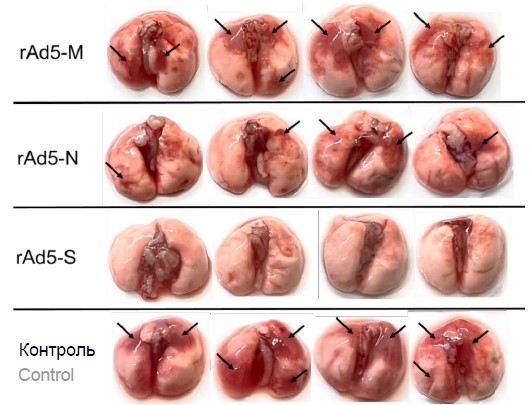
На 4-е сутки после заражения проводили эвтаназию части животных для макроскопического анализа состояния лёгких и анализа вирусной нагрузки. Анализ состояния лёгких на 4-е сутки после заражения показал, что наиболее выраженные повреждения обнаружены в группе контрольных животных и группе животных, получивших препарат на основе белка М (наблюдаются лейкоцитарная инфильтрация, ателектаз и изменение мембранных структур стенки альвеол). Менее выраженные повреждения выявлены у животных в группе, получившей препарат на основе нуклеопротеина N. У животных, получивших препарат на основе гликопротеина S, повреждения лёгких отсутствовали (рис. 2).
Рис. 2. Фотографии лёгких hАСЕ2-трансгенных мышей — вакцинированных и контрольной группы (n = 4), на 4-е сутки после заражения вирусом SARS-CoV-2. Стрелками отмечены зоны поражения лёгочной ткани.
Fig. 2. Photographs of the lungs of hACE2-transgenic mice from the immunized and control groups (n = 4) on day 4 after infection with the SARS-CoV-2 virus. Arrows indicate areas of lung tissue damage.
При анализе вирусной нагрузки в лёгких заражённых животных в контрольной группе и в группах, получивших препараты на основе нуклеопротеина N и мембранного белка М, обнаружен жизнеспособный вирус. У животных, получивших препарат на основе нуклеопротеина N, выявлено достоверное снижение вирусной нагрузки на 1,3 lg TCID50. В группе, получившей препарат на основе гликопротеина S, жизнеспособный вирус SARS-CoV-2 отсутствовал, а снижение вирусной нагрузки составило 5 lg TCID50 (рис. 3).
Рис. 3. Вирусная нагрузка в лёгких hАСЕ2-трансгенных мышей — вакцинированных и контрольной групп (n = 4), на 4-е сутки после заражения вирусом SARS-CoV-2.
Отмечены индивидуальные данные по каждому животному, среднее арифметическое и стандартное отклонение, уровень значимости р (t-критерий Стьюдента).
Fig. 3. Viral load in the lungs of hACE2-transgenic mice from the immunized and control groups (n = 4) on day 4 after infection with the SARS-CoV-2 virus.
The figure shows individual data for each animal, the arithmetic mean and standard deviation, as well as the significance level p (Student's t-test).
Обсуждение
При выборе антигена для включения в состав кандидатных вакцинных препаратов важно иметь понимание об иммунологических особенностях ответа на натуральную инфекцию COVID-19. Взаимодействие иммунных клеток с основными структурными белками вируса индуцирует формирование противовирусного иммунного ответа. Для вируса SARS-CoV-2 такими структурными белками являются S, М, N и E. Несмотря на иммуногенность гликопротеина S, нуклеопротеин N и мембранный белок M также вносят существенный вклад в развитие специфического иммунного ответа. У пациентов с COVID-19 на ранних сроках детектируют появление специфических антител к N-белку, в то время как антитела к гликопротеину S детектируют на 4–8-е сутки после появления симптомов заболевания, что, по всей видимости, связано с наибольшей представленностью нуклеопротеина N в вирионе [23, 24]. Клеточный иммунный ответ также играет важную роль в защите против COVID-19. В ряде исследований было показано, что активная пролиферация CD4+- и CD8+-Т-клеток коррелирует с менее тяжёлым течением болезни и высокой степенью элиминации вируса8. Клеточный иммунный ответ также в значительной степени специфичен к структурным белкам SARS-CoV-2. В результате определения репертуара Т-клеточных эпитопов SARS-CoV-2, включающего 21 исследование, показано, что из общего числа проанализированных CD4+-эпитопов к белку S относятся 33%, к белку N — 11%, к белку M — 10%; из общего числа проанализированных СD8+-эпитопов к белку S относятся 26%, к белку N — 7%, к белку M — 6% [25]. Полифункциональный N-специфичный CD8+-Т-клеточный ответ ассоциирован с более лёгкой тяжестью заболевания COVID-19 [26]. Поскольку N консервативен между различными вариантами SARS-CoV-2, N-специфичные CD4+-Т-клетки потенциально могут обеспечивать защиту против различных генетических вариантов SARS-CoV-2 [27].
Сегодня большинство вакцин для профилактики COVID-19, применяемых в клинической практике, основаны на использовании гликопротеина S вируса SARS-CoV-2. Однако до сих пор ведутся дебаты, какой антиген следует включать в состав препаратов для профилактики COVID-19. Это связано с высокой вариабельностью гликопротеина S и со снижением эффективности существующих вакцин на его основе в отношении новых вариантов вируса SARS-CoV-2.
При анализе аминокислотных последовательностей структурных белков вируса SARS-CoV-2 — вариантов от Delta до актуального KS.1 — нами было продемонстрировано, что наибольшее число замен выявлено в гликопротеине S, наименьшее — в нуклеопротеине N, что делает N белок наиболее консервативным среди структурных белков вируса SARS-CoV-2.
С целью прямого сравнения протективного потенциала структурных белков вируса SARS-CoV-2, наиболее представленных в вирионе, нами были получены кандидатные препараты на основе рекомбинантных аденовирусов человека 5-го серотипа, несущих структурные белки вируса SARS-CoV-2: rAd5-M, rAd5-N и rAd5-S. Данными препаратами иммунизировали hACE2-трансгенных мышей, после чего животных заражали вирусом SARS-CoV-2 B.1.1.1 для оценки протективной эффективности. Мембранный белок М не проявил защитной эффективности — все вакцинированные животные погибли после заражения, а в лёгких на 4-е сутки после заражения обнаружили жизнеспособный вирус в титре, схожем с контрольными невакцинированными животными. Аналогичные данные были получены J. Chen и соавт. при исследовании эффективности использования препаратов на основе плазмидных ДНК [28]. Использование в качестве антигена нуклеопротеина N позволяет снизить вирусную нагрузку в лёгких иммунизированных животных, однако снижение не достигает требуемых 2 lg, в то же время в лёгких вакцинированных животных выявили патологические повреждения, а защита от летальной инфекции, вызванной вирусом SARS-CoV-2, составила лишь 50%, что соотносится с исследованиями других авторов [29–32]. Использование в качестве антигена гликопротеина S позволило сформировать протективный иммунитет у животных (в лёгких отсутствуют патологические повреждения и жизнеспособный вирус) и защитить всех животных от летальной инфекции, вызванной вирусом SARS-CoV-2, что подтверждается исследованиями других авторов [33–36]. J. Chen и соавт. продемонстрировали, что совместная иммунизация двумя вакцинами на основе ДНК, несущими гены белков S и N, индуцирует более выраженный клеточный и гуморальный иммунный ответ, а также обладает большей протективной эффективностью в отношении вируса SARS-CoV-2 на модели инфекции у мышей [32]. R.L. Hajnik и соавт. на модели COVID-19 у хомяков показали, что вакцинация комбинированным препаратом мРНК-S+N индуцирует более выраженный протективный ответ в отношении вируса SARS-CoV-2 вариантов Delta и Omicron по сравнению с монокомпонентными препаратами [37]. Исходя из вышесказанного, следует рассмотреть возможность комбинации антигенов в составе вакцинных препаратов для профилактики COVID-19 в будущих исследованиях. Однако стоит учитывать, что включение в состав вакцинных препаратов нескольких антигенов значительно повышает стоимость производственного процесса и, как следствие, стоимость препарата.
Анализ данных эффективности разных вакцин для профилактики COVID-19 в рамках контролируемых клинических исследований по всему миру показал, что мРНК- и векторные вакцины, несущие ген гликопротеина S вируса SARS-CoV-2, позволяют сформировать наиболее высокий уровень защиты населения по показателям заболеваемости, госпитализации и COVID-19-ассоциированных смертей [9, 38]. Учитывая постоянную смену циркулирующих вариантов вируса SARS-CoV-2, снижение эффективности используемых вакцин в отношении новых вариантов вируса [39] и сохраняющийся высокий уровень заболеваемости COVID-19, необходимо проводить непрерывный мониторинг эффективности вакцинных препаратов в отношении новых вариантов вируса. В случае выявления снижения эффективности необходимо проводить актуализацию антигенного состава вакцин. Данные исследования гармонизуются с исследованиями ВОЗ, по результатам которых с 2022 г. ВОЗ выпускает рекомендации о смене антигенного состава вакцин9. В 2023 г. по результатам мониторинга эффективности была проведена актуализация антигенного состава российских вакцин линейки «Гам-КОВИД-Вак» (векторные вакцины, несущие ген гликопротеина S вируса SARS-CoV-2) под сублинию ХВВ, проведённые клинические исследования вакцин с обновлённым составом показали благоприятный профиль безопасности, формирование нейтрализующих антител к циркулировавшим в 2023 г. и циркулирующим в 1-й половине 2024 г. сублиниям варианта Omicron, и сегодня препараты внедрены в гражданский оборот для защиты населения от актуальных циркулирующих вариантов SARS-CoV-2.
Выводы
Вирус SARS-CoV-2 активно эволюционирует, что приводит к появлению новых вариантов вируса. Среди структурных белков наибольшей изменчивости подвержен поверхностный гликопротеин S, который играет важную роль в жизненном цикле вируса — интернализации, а также является ключевой мишенью для нейтрализующих антител.
Сравнительный анализ протективного потенциала разных структурных белков вируса SARS-CoV-2 на модели летальной инфекции у животных показал, что только использование гликопротеина S позволяет сформировать протективный иммунный ответ, который защищает 100% животных от летальной инфекции, вызванной вирусом SARS-CoV-2, при этом снижение вирусной нагрузки в лёгких животных на 4-е сутки после заражения составило 5,0 lg TCID50 (100 000 раз). В то же время использование нуклеопротеина N привело к снижению вирусной нагрузки на 1,3 lg TCID50 (20 раз), а защита от летальной инфекции составила 50%.
1 COVID-19 Vaccine tracker. Approved vaccines. URL: https://covid19.trackvaccines.org/vaccines/approved/
2 NextStrain. Genomic epidemiology of SARS-CoV-2 with subsampling focused globally since pandemic start.
URL: https://nextstrain.org/ncov/gisaid/global/all-time
WHO. WHO COVID-19 dashboard. URL: https://data.who.int/dashboards/covid19/cases?m49 = 001
3 OurWorldInData. United States: COVID-19 weekly death rate by vaccination status, All ages. URL: https://ourworldindata.org/grapher/united-states-rates-of-covid-19-deaths-by-vaccination-status
WHO. Statement on the antigen composition of COVID-19 vaccines. 18.05.2023. URL: https://www.who.int/news/item/18-05-2023-statement-on-the-antigen-composition-of-covid-19-vaccines
WHO. Statement on the antigen composition of COVID-19 vaccines. 26.04.2024. URL: https://www.who.int/news/item/26-04-2024-statement-on-the-antigen-composition-of-covid-19-vaccines
4 Руководство по лабораторным животным и альтернативным моделям в биомедицинских технологиях / под ред. Н.Н. Каркищенко, С.В. Грачева. М.; 2010. 354 с.
5 covSPECTRUM. Detect and analyze variants of SARS-CoV-2. URL: https://cov-spectrum.org/
6 covSPECTRUM. B.1.617.2 (Nextclade). URL: https://cov-spectrum.org/explore/World/AllSamples/from%3D2020-01-01%26to%3D2024-10-11/variants?nextcladePangoLineage=B.1.617.2&
covSPECTRUM. BA.1 (Nextclade). URL: https://cov-spectrum.org/explore/World/AllSamples/from%3D2020-01-01%26to%3D2024-10-11/variants?nextcladePangoLineage=ba.1&
covSPECTRUM. BA.5 (Nextclade). URL: https://cov-spectrum.org/explore/World/AllSamples/from%3D2020-01-01%26to%3D2024-10-11/variants?nextcladePangoLineage=ba.5&
covSPECTRUM. XBB (Nextclade). URL: https://cov-spectrum.org/explore/World/AllSamples/from%3D2020-01-01%26to%3D2024-10-11/variants?nextcladePangoLineage=xbb&
covSPECTRUM. BA.2.86 (Nextclade). URL: https://cov-spectrum.org/explore/World/AllSamples/from%3D2020-01-01%26to%3D2024-10-11/variants?nextcladePangoLineage=ba.2.86&
covSPECTRUM. KS.1 (Nextclade). URL: https://cov-spectrum.org/explore/World/AllSamples/from%3D2020-01-01%26to%3D2024-10-11/variants?nextcladePangoLineage=ks.1&
7 covSPECTRUM. B (Nextclade). URL: https://cov-spectrum.org/explore/World/AllSamples/from%3D2020-01-01%26to%3D2024-10-11/variants?nextcladePangoLineage = B&nextcladePangoLineage1=B.1&
8 CDC. CDC Museum COVID-19 timeline. Centers for Disease Control and Preventio.
URL: https://www.cdc.gov/museum/timeline/covid19.html
9 WHO. Technical Advisory Group on COVID-19 Vaccine Composition. URL: https://www.who.int/groups/technical-advisory-group-on-covid-19-vaccine-composition-(tag-co-vac)
Об авторах
Инна Вадимовна Должикова
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Автор, ответственный за переписку.
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0003-2548-6142
к. б. н., зав. лаб. Государственной коллекции вирусов
Россия, МоскваДарья Михайловна Гроусова
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0002-3299-4818
м. н. с. лаб. Государственной коллекции вирусов
Россия, МоскваИлья Дмитриевич Зорков
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0001-8311-2283
м. н. с. лаб. Государственной коллекции вирусов
Россия, МоскваАнна Алексеевна Илюхина
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0003-0728-5478
м. н. с. лаб. Государственной коллекции вирусов
Россия, МоскваАнна Витальевна Ковыршина
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0002-8757-7026
н. с. лаб. клеточной микробиологии
Россия, МоскваОльга Вадимовна Зубкова
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0001-7893-8419
к. б. н., в. н. с. лаб. иммунобиотехнологии
Россия, МоскваОльга Дмитриевна Попова
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0003-3248-1227
м. н. с. лаб. иммунобиотехнологии
Россия, МоскваТатьяна Андреевна Ожаровская
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0001-7147-1553
к. б. н., с. н. с. лаб. иммунобиотехнологии
Россия, МоскваДенис Игоревич Зрелкин
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0003-0899-8357
м. н. с. лаб. иммунобиотехнологии
Россия, МоскваДарья Михайловна Савина
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0002-2228-3406
н. с. подразделения по внедрению в производства научных разработок
Россия, МоскваЕкатерина Геннадьевна Самохвалова
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0002-0127-173X
н. с. лаб. Государственной коллекции вирусов
Россия, МоскваАмир Ильдарович Тухватулин
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0001-8506-2339
к. б. н., зав. лаб. микоплазм и Л-форм бактерий
Россия, МоскваДмитрий Викторович Щебляков
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0002-1289-3411
к. б. н., зав. лаб. иммунобиотехнологии
Россия, МоскваДенис Юрьевич Логунов
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0003-4035-6581
д. б. н., академик РАН, зав. лаб. клеточной микробиологии
Россия, МоскваАлександр Леонидович Гинцбург
Национальный исследовательский центр эпидемиологии и микробиологии имени почетного академика Н.Ф. Гамалеи; Первый Московский государственный медицинский университет имени И.М. Сеченова (Сеченовский Университет)
Email: dolzhikova@gamaleya.org
ORCID iD: 0000-0003-1769-5059
д. б. н., проф., академик РАН, директор, зав. кафедрой инфектологии и вирусологии
Россия, Москва; МоскваСписок литературы
- Ke Z., Oton J., Qu K., et al. Structures and distributions of SARS-CoV-2 spike proteins on intact virions. Nature. 2020;588(7838):498–502. DOI: https://doi.org/10.1038/s41586-020-2665-2
- Letko M., Marzi A., Munster V. Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses. Nat. Microbiol. 2020;5(4):562–9. DOI: https://doi.org/10.1038/s41564-020-0688-y
- Du S., Cao Y., Zhu Q., et al. Structurally resolved SARS-CoV-2 antibody shows high efficacy in severely infected hamsters and provides a potent cocktail pairing strategy. Cell. 2020;183(4):1013–23.e13. DOI: https://doi.org/10.1016/j.cell.2020.09.035
- Zost S.J., Gilchuk P., Case J.B., et al. Potently neutralizing and protective human antibodies against SARS-CoV-2. Nature. 2020;584(7821):443–9. DOI: https://doi.org/10.1038/s41586-020-2548-6
- Robbiani D.F., Gaebler C., Muecksch F., et al. Convergent antibody responses to SARS-CoV-2 in convalescent individuals. Nature. 2020;584(7821):437–42. DOI: https://doi.org/10.1038/s41586-020-2456-9
- Mu J., Xu J., Zhang L., et al. SARS-CoV-2-encoded nucleocapsid protein acts as a viral suppressor of RNA interference in cells. Sci. China Life Sci. 2020;63(9):1413–6. DOI: https://doi.org/10.1007/s11427-020-1692-1
- Zhang Z., Nomura N., Muramoto Y., et al. Structure of SARS-CoV-2 membrane protein essential for virus assembly. Nat. Commun. 2022;13(1):4399. DOI: https://doi.org/10.1038/s41467-022-32019-3
- Dai L., Gao G.F. Viral targets for vaccines against COVID-19. Nat. Rev. Immunol. 2021;21(2):73–82. DOI: https://doi.org/10.1038/s41577-020-00480-0
- Graña C., Ghosn L., Evrenoglou T., et al. Efficacy and safety of COVID-19 vaccines. Cochrane Database Syst. Rev. 2022;12(12):CD015477. DOI: https://doi.org/10.1002/14651858.CD015477
- Rijkers G.T., Weterings N., Obregon-Henao A., et al. Antigen presentation of mRNA-based and virus-vectored SARS-CoV-2 vaccines. Vaccines (Basel). 2021;9(8):848. DOI: https://doi.org/10.3390/vaccines9080848
- Sahin U., Muik A., Derhovanessian E., et al. COVID-19 vaccine BNT162b1 elicits human antibody and TH1 T cell responses. Nature. 2020;586(7830):594–9. DOI: https://doi.org/10.1038/s41586-020-2814-7
- Mulligan M.J., Lyke K.E., Kitchin N., et al. Phase I/II study of COVID-19 RNA vaccine BNT162b1 in adults. Nature. 2020;586(7830):589–93. DOI: https://doi.org/10.1038/s41586-020-2639-4
- Logunov D.Y., Dolzhikova I.V., Zubkova O.V., et al. Safety and immunogenicity of an rAd26 and rAd5 vector-based heterologous prime-boost COVID-19 vaccine in two formulations: two open, non-randomised phase 1/2 studies from Russia. Lancet. 2020;396(10255):887–97. DOI: https://doi.org/10.1016/S0140-6736(20)31866-3
- Tukhvatulin A.I., Dolzhikova I.V., Dzharullaeva A.S., et al. Safety and immunogenicity of rAd26 and rAd5 vector-based heterologous prime-boost COVID-19 vaccine against SARS-CoV-2 in healthy adolescents: an open-label, non-randomized, multicenter, phase 1/2, dose-escalation study. Front. Immunol. 2023;14:1228461. DOI: https://doi.org/10.3389/fimmu.2023.1228461
- Logunov D.Y., Dolzhikova I.V., Shcheblyakov D.V., et al. Safety and efficacy of an rAd26 and rAd5 vector-based heterologous prime-boost COVID-19 vaccine: an interim analysis of a randomised controlled phase 3 trial in Russia. Lancet. 2021;397(10275):671–81. DOI: https://doi.org/10.1016/S0140-6736(21)00234-8
- Al-Shudifat A.E., Al-Tamimi M., Dawoud R., et al. Anti-S and Anti-N antibody responses of COVID-19 vaccine recipients. Vaccines (Basel). 2023;11(9):1398. DOI: https://doi.org/10.3390/vaccines11091398
- Qaqish A., Abbas M.M., Al-Tamimi M., et al. SARS-CoV-2 antinucleocapsid antibody response of mRNA and inactivated virus vaccines compared to unvaccinated individuals. Vaccines (Basel). 2022;10(5):643. DOI: https://doi.org/10.3390/vaccines10050643
- Goldblatt D., Alter G., Crotty S., Plotkin S.A. Correlates of protection against SARS-CoV-2 infection and COVID-19 disease. Immunol. Rev. 2022;310(1):6–26. DOI: https://doi.org/10.1111/imr.13091
- Finkelstein M.T., Mermelstein A.G., Parker Miller E., et al. Structural analysis of neutralizing epitopes of the SARS-CoV-2 spike to guide therapy and vaccine design strategies. Viruses. 2021;13(1):134. DOI: https://doi.org/10.3390/v13010134
- Markov P.V., Ghafari M., Beer M., et al. The evolution of SARS-CoV-2. Nat. Rev. Microbiol. 2023;21(6):361–79. DOI: https://doi.org/10.1038/s41579-023-00878-2
- McCray PB Jr., Pewe L., Wohlford-Lenane C., et al. Lethal infection of K18-hACE2 mice infected with severe acute respiratory syndrome coronavirus. J. Virol. 2007;81(2):813–21. DOI: https://doi.org/10.1128/JVI.02012-06
- Унгуряну Т.Н., Гржибовский А.М. Краткие рекомендации по описанию, статистическому анализу и представлению данных в научных публикациях. Экология человека. 2011; (5): 55–60. Unguryanu T.N., Grjibovski A.M. Brief recommendations on description, analysis and presentation of data in scientific papers. Human Ecology. 2011; (5): 55–60.
- Tan Y.J., Goh P.Y., Fielding B.C., et al. Profiles of antibody responses against severe acute respiratory syndrome coronavirus recombinant proteins and their potential use as diagnostic markers. Clin. Diagn. Lab. Immunol. 2004;11(2):362–71. DOI: https://doi.org/10.1128/cdli.11.2.362-371.2004
- Wu H.S., Hsieh Y.C., Su I.J., et al. Early detection of antibodies against various structural proteins of the SARS-associated coronavirus in SARS patients. J. Biomed. Sci. 2004;11(1):117–26. DOI: https://doi.org/10.1007/BF02256554
- Andersen K.G., Rambaut A., Lipkin W.I., et al. The proximal origin of SARS-CoV-2. Nat. Med. 2020;26(4):450–2. DOI: https://doi.org/10.1038/s41591-020-0820-9
- Peng Y., Mentzer A.J., Liu G., et al. Broad and strong memory CD4+ and CD8+ T cells induced by SARS-CoV-2 in UK convalescent individuals following COVID-19. Nat. Immunol. 2020;21(11):1336–45. DOI: https://doi.org/10.1038/s41590-020-0782-6
- Yang G., Wang J., Sun P., et al. SARS-CoV-2 epitope-specific T cells: Immunity response feature, TCR repertoire characteristics and cross-reactivity. Front. Immunol. 2023;14:1146196. DOI: https://doi.org/10.3389/fimmu.2023.1146196
- Chen J., Deng Y., Huang B., et al. DNA vaccines expressing the envelope and membrane proteins provide partial protection against SARS-CoV-2 in mice. Front. Immunol. 2022;13:827605. DOI: https://doi.org/10.3389/fimmu.2022.827605
- Matchett W.E., Joag V., Stolley J.M., et al. Cutting Edge: nucleocapsid vaccine elicits spike-independent SARS-CoV-2 protective immunity. J. Immunol. 2021;207(2):376–9. DOI: https://doi.org/10.4049/jimmunol.2100421
- Rabdano S.O., Ruzanova E.A., Pletyukhina I.V., et al. Immunogenicity and in vivo protective effects of recombinant nucleocapsid-based SARS-CoV-2 vaccine Convacell®. Vaccines (Basel). 2023;11(4):874. DOI: https://doi.org/10.3390/vaccines11040874
- Primard C., Monchâtre-Leroy E., Del Campo J., et al. OVX033, a nucleocapsid-based vaccine candidate, provides broad-spectrum protection against SARS-CoV-2 variants in a hamster challenge model. Front. Immunol. 2023;14:1188605. DOI: https://doi.org/10.3389/fimmu.2023.1188605
- Chen J., Huang B., Deng Y., et al. synergistic immunity and protection in mice by co-immunization with DNA vaccines encoding the spike protein and other structural proteins of SARS-CoV-2. Vaccines (Basel). 2023;11(2):243. DOI: https://doi.org/10.3390/vaccines11020243
- Wu Y., Huang X., Yuan L., et al. A recombinant spike protein subunit vaccine confers protective immunity against SARS-CoV-2 infection and transmission in hamsters. Sci. Transl. Med. 2021;13(606):eabg1143. DOI: https://doi.org/10.1126/scitranslmed.abg1143
- Brocato R.L., Kwilas S.A., Kim R.K., et al. Protective efficacy of a SARS-CoV-2 DNA vaccine in wild-type and immunosuppressed Syrian hamsters. NPJ Vaccines. 2021;6(1):16. DOI: https://doi.org/10.1038/s41541-020-00279-z
- DiPiazza A.T., Leist S.R., Abiona O.M., et al. COVID-19 vaccine mRNA-1273 elicits a protective immune profile in mice that is not associated with vaccine-enhanced disease upon SARS-CoV-2 challenge. Immunity. 2021;54(8):1869–82.e6. DOI: https://doi.org/10.1016/j.immuni.2021.06.018
- Darling T.L., Ying B., Whitener B., et al. mRNA-1273 and Ad26.COV2.S vaccines protect against the B.1.621 variant of SARS-CoV-2. Med. 2022;3(5):309–24.e6. DOI: https://doi.org/10.1016/j.medj.2022.03.009
- Hajnik R.L., Plante J.A., Liang Y., et al. Dual spike and nucleocapsid mRNA vaccination confer protection against SARS-CoV-2 Omicron and Delta variants in preclinical models. Sci. Transl. Med. 2022;14(662):eabq1945. DOI: https://doi.org/10.1126/scitranslmed.abq1945
- Kyriakidis N.C., López-Cortés A., González E.V., et al. SARS-CoV-2 vaccines strategies: a comprehensive review of phase 3 candidates. NPJ Vaccines. 2021;6(1):28. DOI: https://doi.org/10.1038/s41541-021-00292-w
- Wu N., Joyal-Desmarais K., Ribeiro P.A.B., et al. Long-term effectiveness of COVID-19 vaccines against infections, hospitalisations, and mortality in adults: findings from a rapid living systematic evidence synthesis and meta-analysis up to December, 2022. Lancet Respir. Med. 2023;11(5):439–52. DOI: https://doi.org/10.1016/S2213-2600(23)00015-2
Дополнительные файлы