Поддесневой микробиом при заболеваниях пародонта и коморбидной патологии (метаанализ)
- Авторы: Царева Т.В.1, Балмасова И.П.1, Царев В.Н.1
-
Учреждения:
- Российский университет медицины
- Выпуск: Том 101, № 2 (2024)
- Страницы: 281-292
- Раздел: ОБЗОРЫ
- URL: https://microbiol.crie.ru/jour/article/view/18538
- DOI: https://doi.org/10.36233/0372-9311-500
- EDN: https://elibrary.ru/mwauhy
- ID: 18538
Цитировать
Аннотация
Проблема хронического пародонтита (ХП) активно обсуждается в связи с признанием того факта, что микробное поражение пародонта тесно связано с рядом системных заболеваний и, вероятно, играет важную роль в возникновении коморбидной патологии.
Цель метаанализа — характеристика состава поддесневого микробиома и определение особенностей формирования ассоциаций нового пародонтопатогена Filifactor alocis с другими пародонтопатогенными бактериями I и II порядка, а также комменсальными бактериями, колонизирующими данный биотоп.
В исследовании представлены данные обследования пациентов с обязательным использованием методов полимеразной цепной реакции и секвенированием генов 16S рРНК у 1529 здоровых лиц и 2394 пациентов с ХП, 136 человек с ассоциацией ХП и атеросклероза, 258 человек с ассоциацией ХП и сахарного диабета 2-го типа. Подтверждено, что основу орального микробиома в норме составляют представители микроаэрофильных стрептококков, коринебактерий, лактобацилл, а также представителей родов Veillonella и Sphingobacterium. Проведённое 16S-секвенирование и биоинформационный анализ позволили конкретизировать таксономическое место нового возбудителя F. alocis, а также представителей нормобиоты при ХП и коморбидной соматической патологии.
Ключевые слова
Полный текст
Введение
Хронический генерализованный пародонтит (ХП) — тяжёлое поражение дёсен и подлежащих тканей пародонта с рецидивирующим течением, которое является основной причиной утраты зубов в среднем и пожилом возрасте. По распространённости эта патология — ведущая стоматологическая проблема в мире среди взрослого работоспособного населения, хотя выявлены существенные вариации частоты в зависимости от возраста, региона, климатических и социальных условий (частота варьирует от 60 до 90%) [1–3]. Социально-медицинское значение проблемы ХП постоянно увеличивается, в том числе в связи с ростом заболевания агрессивным пародонтитом, развивающимся в молодом возрасте [4, 5]. Проблема ХП активно обсуждается на различных конгрессах, проводимых под эгидой ВОЗ, в связи с признанием того факта, что микробное поражение пародонта тесно связано с рядом системных заболеваний и, вполне вероятно, играет важную роль в возникновении коморбидной патологии [5–7].
Способность микроорганизмов колонизировать слизистую оболочку рта, ткани пародонта и поверхности зубных рядов с формированием многокомпонентной структурированной микробной биоплёнки чрезвычайно важна с биологической точки зрения. Бактерии осуществляют многочисленные метаболические, барьерные и протективные функции, способствуют целостности тканей пародонта, увеличивают пролиферацию клеток эпителия, готовят барьер слизистой оболочки к потенциальной агрессии патогенов за счёт выработки антимикробных пептидов и цитокинов [6].
Ключевым моментом развития инфекционно-воспалительного поражения тканей пародонта является колонизация микробной биоплёнки приоритетными патогенами, способными к внутриклеточному паразитизму и получившими название пародонтопатогенных. Локализация этой биоплёнки — область зубо-десневой борозды, а по мере прогрессирования деструктивного процесса — в формирующемся пародонтальном кармане [8, 9].
Таким образом, речь идёт о поддесневом микробиоме, формирующемся при колонизации биоплёнки как представителями нормобиоты, так и пародонтопатогенными видами бактерий.
В 1998 г. S.S. Socransky и соавт. предложили классификацию, основанную на изучении степени частоты определения представителей микрофлоры в очаге воспаления: выделены «красный», «оранжевый» и другие комплексы, а также в большей степени представляющие нормальную и транзиторную микрофлору — «жёлтый», «зелёный», «фиолетовый». К «красному» комплексу, в частности, отнесены Porphyromonas gingivalis, Tannerlla forsythia (по старой номенклатуре — Bacteroides forsytus), Treponema denticola [10]. Однако в настоящее время эта классификация больше имеет историческое значение, т. к. некоторые возбудители агрессивного пародонтита, например токсигенный серотип b актинобациллы Aggregatibacter actinomycetemcomitans, были описаны несколько позже [11].
В отечественной литературе после I Съезда пародонтологов России в 2005 г. впервые было введено понятие о пародонтопатогенных бактериях I порядка, которые обладают сочетанием трёх ведущих признаков: вертикальная и горизонтальная передача инфекционного агента от человека к человеку, способность к внутриклеточному паразитизму, а также токсикогенность (имеется в виду, прежде всего, продукция экзотоксинов), в отличие от пародонтопатогенов II порядка, которые не имеют полного набора этих признаков. К пародонтопатогенным видам I порядка были отнесены три возбудителя: P. gingivalis, T. forsythia и A. actinomycetemcomitans, токсигенный серотип b [8, 9, 12]. В дальнейших исследованиях были установлены диагностически значимые показатели микробного числа для разных видов пародонтопатогенных бактерий I и II порядков, которые с некоторыми коррективами представлены в табл. 1.
Таблица 1. Количественные параметры содержания пародонтопатогенных бактерий I и II порядков в биоплёнке десны в норме [6, 13]
Table 1. Quantitative parameters of the content of periodontal pathogenic bacteria of I and II orders in the gingival biofilm under normal circumstances [6, 13]
Группа пародонтопатогенов Periodontal pathogen group | Признаки группы Group traits | Виды пародонтопатогенов Periodontal pathogen type | Количество, КОЕ/мл Quantity, CFU/ml |
Пародонтопатогенные виды I порядка Periodontal pathogenic species of the I order | Представители данных видов являются возбудителями пародонтита; в норме у здорового человека отсутствуют (за исключением случаев «здорового носительства» в 6–12% случаев) Representatives of these species are pathogens of periodontitis; normally absent in a healthy person (except for healthy carriage in 6–12% of cases) | A. actinomycetemcomitans | > 102 |
B. (Tannerella) forsythia | > 102 | ||
P. gingivalis | > 102 | ||
Пародонтопатогенные виды II порядка Periodontal pathogenic species of the II order | Представители данных видов на дёснах в норме отсутствуют или определяются в малом количестве. Превышение указанного количества — признак развития пародонтита Representatives of these species on the gums are normally absent or determined in small numbers. Exceeding the specified number is a sign of periodontitis development | Prevotella intermedia | > 102 |
Treponema denticola | > 103 | ||
Fusobacterium nucleatum | > 103 | ||
Filifactor alocis | > 103 | ||
Actinomyces israelii | > 104 | ||
Parvimonas micros | > 104 | ||
Streptococcus intermedius | > 104 | ||
Eykenella corrodens | > 103 | ||
Selenomonas spp. | > 103 | ||
Wolinella recta | > 103 | ||
Candida albicans, C. krusei, C. glabrata | > 102 |
При развитии заболеваний пародонта довольно часто формируются синергические и/или мутуалистические взаимодействия между оральными патогенами (пародонтопатогенами), которые, как считается, способствуют возникновению и прогрессированию заболевания. Однако не следует забывать, что в основе таких взаимоотношений лежит потенцирование действия их факторов вирулентности как возбудителей инфекционного процесса [6, 13]. И если этиологическая роль P. gingivalis как ключевого пародонтопатогена сомнению не подвергается [14], то значение некоторых других труднокультивируемых бактерий, которые начали изучаться по данным современных молекулярных методов исследований, остаётся предметом дискуссий [15–17].
Одним из таких микробов, по-видимому, является Filifactor alocis, относительно недавно выделенный из класса Fusobacteria, типа Firmicutes в отдельный род, представленный единственным видом. Как подчёркивают Е. Aja и соавт., F. alocis является новым членом микробиома пародонта и в настоящее время предлагается использовать его в качестве диагностического индикатора заболеваний пародонта [18]. Однако из-за отсутствия генетических инструментов для изучения этого микроорганизма до последнего времени мало известно о его характеристиках и вирулентности, а значение при коморбидной патологии описано в единичных публикациях [19]. Это обстоятельство побудило нас в недавно проведённых исследованиях к созданию диагностического набора праймеров и зондов для мультиплексной полимеразной цепной реакции (ПЦР) для детекции данного вида пародонтопатогенных бактерий [20, 21]. С другой стороны, очевидна необходимость анализа имеющихся данных о составе и взаимоотношениях компонентов поддесневого микробиома.
Целью метаанализа являлись характеристика состава поддесневого микробиома и определение особенностей формирования ассоциаций нового пародонтопатогена F. alocis с другими пародонтопатогенными бактериями I и II порядков, а также комменсальными бактериями, колонизирующими данный биотоп.
Материалы и методы
Поиск проводили в базах данных MEDLINE (через PubMed), EMBASE и Cochrane Library с 2005 г. до октября 2023 г. по ключевым словам, обозначенным в цели исследования, с использованием стратегии поиска, соответствующей разработанным критериям включения в исследование и исключения из него.
Критерии включения:
1) исследования микробиома с использованием секвенирования генов 16S рРНК бактерий, включая метаанализы и систематические обзоры, в которых сравнивались результаты, полученные микробиологическими и молекулярно-биологическими методами (проценты встречаемости типов, родов, видов бактерий), в образцах поддесневого налета пациентов:
- системно здоровых, без явлений пародонтита (группа контроля);
- с ХП без системной патологии;
- с ХП при сопутствующем верифицированном атеросклерозе (АТ);
- с ХП при сопутствующем верифицированном сахарном диабете 2-го типа (СД2);
2) учитывали только исследования с указанием на выявление в составе микробиома относительно нового труднокультивируемого пародонтопатогенного вида — F. alocis.
Критерии исключения:
- исследования, опубликованные на других языках, кроме русского или английского;
- отсутствие первичных данных;
- отсутствие данных по образцам из зубодесневой борозды системно здоровых людей в группах пародонтально здоровых и/или из пародонтальных карманов глубиной 5 мм и более в группах больных ХП, в том числе с сопутствующей патологией;
- исследования, в которых оценивали только состояние пациентов с гингивитом без признаков пародонтита, с агрессивным или рефрактерным пародонтитом.
В ходе поиска было найдено в общей сложности 1437 исследований. После титульного скрининга 1211 работ были исключены, а 126 были отобраны. После прочтения рефератов 12 исследований были исключены, а 114 полнотекстовых публикаций подверглись всесторонней оценке. После ознакомления с этими исследованиями 92 были исключены за несоответствие критериям включения. Таким образом, в данное исследование были включены результаты из 22 источников литературы.
Результаты
После получения соответствующего массива проводили извлечение данных с обозначением следующей дополнительной информации:
- место проведения исследования;
- характеристики участников (по клиническому состоянию пародонта и наличию коморбидной патологии);
- тип микробиологической оценки (метод исследования);
- микробиологические результаты (в процентах) по отдельным таксонам бактерий в разных группах исследования.
Типы, роды, виды бактерий впоследствии были проиндексированы в соответствии с таксономическим указателем Национального центра биотехнологической информации США1.
Методические особенности, год и место выполнения исследований в использованных для метаанализа источниках литературы обобщены и представлены в табл. 2.
Таблица 2. Сведения об особенностях исследований, включённых в метаанализ
Table 2. Information about the characteristics of the studies included in the meta-analysis
Тип исследования Study type | Страна Country | Группа исследования Study group | n | Источник Source |
Оригинальное | Original | США | USA | Здоровые лица | Healthy patients | 15 | [22] |
Пациенты с ХП | Patients with CP | 15 | |||
Оригинальное | Original | США | USA | Здоровые лица | Healthy patients | 29 | [23] |
Пациенты с ХП | Patients with CP | 29 | |||
Оригинальное | Original | США | USA | Здоровые лица | Healthy patients | 5 | [24] |
Пациенты с ХП | Patients with CP | 2 | |||
Оригинальное | Original | США, Чили | USA, Chile | Здоровые лица | Healthy patients | 10 | [2] |
Пациенты с ХП | Patients with CP | 22 | |||
Систематический обзор Systemic review | США | USA | Здоровые лица | Healthy patients | 98 | [25] |
Пациенты с ХП | Patients with CP | 24 | |||
Оригинальное | Original | Бразилия, Канада | Brazil, Canada | Пациенты с ХП и АТ | Patients with CP and AT | 18 | [26] |
Оригинальное | Original | Бразилия | Brazil | Здоровые лица | Healthy patients | 27 | [27] |
Пациенты с ХП | Patients with CP | 59 | |||
Оригинальное | Original | Бразилия | Brazil | Здоровые лица | Healthy patients | 912 | [28] |
Пациенты с ХП | Patients with CP | 1918 | |||
Оригинальное | Original | Корея | Korea | Здоровые лица | Healthy patients | 12 | [29] |
Пациенты с ХП | Patients with CP | 10 | |||
Оригинальное | Original | Россия | Russia | Пациенты с ХП и АТ | Patients with CP and AT | 28 | [30] |
Оригинальное | Original | США | USA | Здоровые лица | Healthy patients | 76 | [31] |
Пациенты с ХП | Patients with CP | 76 | |||
Оригинальное | Original | США, Индия | USA, India | Здоровые лица | Healthy patients | 75 | [32] |
Пациенты с ХП | Patients with CP | 50 | |||
Пациенты с ХП и СД2 | Patients with CP and T2D | 50 | |||
Оригинальное | Original | США | USA | Здоровые лица | Healthy patients | 97 | [33] |
Пациенты с ХП и СД2 | Patients with CP and T2D | 98 | |||
Оригинальное | Original | Бразилия, США | Brazil, USA | Здоровые лица | Healthy patients | 98 | [34] |
Оригинальное | Original | Мексика | Mexico | Здоровые лица | Healthy patients | 59 | [35] |
Пациенты с ХП | Patients with CP | 67 | |||
Пациенты с ХП и СД2 | Patients with CP and T2D | 38 | |||
Оригинальное | Original | Саудовская Аравия Saudi Arabia | Здоровые лица | Healthy patients | 19 | [36] |
Пациенты с ХП и СД2 | Patients with CP and T2D | 15 | |||
Оригинальное | Original | Южная Африка South Africa | Здоровые лица | Healthy patients | 32 | [37] |
Пациенты с ХП | Patients with CP | 32 | |||
Пациенты с ХП и СД2 | Patients with CP and T2D | 32 | |||
Оригинальное | Original | Россия | Russia | Здоровые лица | Healthy patients | 16 | [21] |
Пациенты с ХП | Patients with CP | 15 | |||
Пациенты с ХП и СД2 | Patients with CP and T2D | 15 | |||
Оригинальное | Original | Тунис | Tunisia | Здоровые лица | Healthy patients | 10 | [38] |
Пациенты с ХП и АТ | Patients with CP and AT | 20 | |||
Оригинальное | Original | Италия | Italy | Здоровые лица | Healthy patients | 15 | [39] |
Пациенты с ХП и АТ | Patients with CP and AT | 15 | |||
Оригинальное | Original | Индия | India | Пациенты с ХП и АТ | Patients with CP and AT | 12 | [40] |
Оригинальное | Original | Япония | Japan | Пациенты с ХП и АТ | Patients with CP and AT | 43 | [41] |
Примечание. Во всех исследованиях проведено секвенирование генов 16S рРНК.
Note. 16S rRNA genes were sequenced in all studies.
Среди источников литературы, вошедших в исследование, было 2 систематических обзора с указанием объединённых из нескольких источников числовых данных, а остальные источники содержали результаты оригинальных исследований. Количественные данные, использованные при проведении метаанализа, содержались либо в составе графического материала, либо в виде дополнительной информации к публикации.
Обобщённая характеристика контингентов лиц, подвергнутых анализу по результатам оригинальных исследований, представлена в табл. 3. Число обследованных здоровых лиц и пациентов, страдающих ХП, содержалось в 16 и 12 источниках соответственно и было довольно значимым (1529 и 2394 человек). Что касается групп пациентов с ХП, ассоциированным с системными патологическими процессами, то условиям поиска удовлетворяли только 6 источников, содержащих данные по составу субгингивального микробиома у 136 человек с ассоциацией ХП и АТ, и 6 источников по результатам аналогичных исследований, в которых анализировались данные 258 человек с ассоциацией ХП и СД2.
Таблица 3. Суммарная характеристика обследованных лиц, включённых в метаанализ
Table 3. Cumulative characteristics of the study population included in the meta-analysis
Анализируемые признаки Analyzed attributes | Группа исследования | Study groups | ||||
здоровые лица healthy patients | пациенты с ХП patients with CP | пациенты с ХП и АТ patients with CP and AT | пациенты с ХП и СД2 patients with CP and T2D | ||
n | 1529 | 2394 | 136 | 258 | |
Возраст, лет | Age, years | 18–70 | 18–64 | 40–79 | 26–62 | |
Пол, % | Sex, % | мужчины | male | 20–80 | 19–73 | 5–80 | 19–51 |
женщины | female | 20–80 | 27–81 | 20–95 | 49–81 | |
Курение, % | Smokers, % | 0–50 | 17–21 | 24–56 | 17–52 | |
Глубина пародонтальных карманов, мм Depth of periodontal pockets, mm | < 3 | ≥ 5 | ≥ 5 | ≥ 5 | |
Индекс кровоточивости, % Bleeding index, % | < 10 | ≥ 30 | ≥ 30 | ≥ 30 | |
Уровень гликозилированного гемоглобина, % Glycosylated hemoglobin level, % | < 6% | < 6 | < 7% | > 6,5 | |
Особенностью сравнительных данных, содержащихся во всех 6 источниках по ассоциации ХП и АТ, является изучение не только субгингивального микробиома, но и таксономического состава бактерий, обнаруженных в АТ-бляшках стенок сосудов, удалённых хирургическим путём в процессе оказания как экстренной, так и плановой помощи пациентам.
Так, не только метагеномными методами, но и с помощью ПЦР было установлено, что пародонтопатогенные бактерии I порядка, относящиеся к видам P. gingivalis, A. actinomycetemcomitans и T. forsythia, были обнаружены более чем в 60% образцов, полученных после эндартериоэктомии у испанских пациентов [43]. Однако данные ПЦР тромбов японских пациентов с острым инфарктом миокарда показали, что среди пародонтопатогенов наиболее распространённым организмом был A. actinomycetemcomitans, обнаруженный примерно в 20% образцов [44].
Кроме этого, АТ-бляшки содержали Acinetobacter (39%), Chryseobacterium (9%), Rhizobium (5%) и Staphylococcus (4%). Сравнение этого микробиома с субгингивальной микробиотой показало, что 22 бактериальных рода были общими для двух разных локусов, при этом Acinetobacter оказался доминирующим [40].
Ещё в одном исследовании бактериальная ДНК была обнаружена в 12 образцах при эндартериоэктомии (34,3%). Идентифицировано 23 вида/филотипа бактерий. Протеобактерии и фирмикуты составляли 78,3 и 21,7% идентифицированных таксонов соответственно. При этом 15 (60,9%) таксонов принадлежали бактериям, не культивируемым на питательных средах. Пародонтопатоген A. actinomycetemcomitans был обнаружен в 7 (20%) образцах, за которыми следовали виды из рода Pseudomonas. Таким образом, результаты свидетельствуют о роли микробиоты полости рта, в частности, А. actinomycetemcomitans, в развитии воспаления в процессе атерогенеза [45].
Важно отметить, что ни в одном случае не указывалось на наличие в АТ-бляшках представителей вида F. alocis, в связи с чем эти данные по микробиому АТ-бляшек не были включены в проводимый метаанализ.
Сравнительный анализ таксономического состава субгингивального микробиома в группах исследования
Для сравнительной оценки общих тенденций изменений со стороны микробиома в группах людей со здоровым пародонтом, при ХП без системной патологии, ХП в ассоциации с АТ или с СД2 прежде всего определялось соотношение бактерий, принадлежащих к разным типам: Firmicutes, Proteobacteria, Bacteroidetes, Actinobacteria, Fusobacteria, Synergistes, Spirochaetes.
Сравнительный статистический анализ результатов осуществляли методом однофакторного дисперсионного анализа (One-way ANOVA), он был направлен на исследование значимости различия между средними величинами с учётом разброса (дисперсии) данных по каждому показателю. Количественным выражением такого разброса служил критерий Фишера (F) с указанием вероятности (р) его статистической значимости.
Как следует из представленных данных, статистически значимых различий по процентному содержанию отдельных типов бактерий в составе микробиома, кроме таксономической группы ТМ7, не установлено (табл. 4) в связи с высокой гетерогенностью данных. Этот вывод распространяется и на тип Firmicutes, к которому принадлежит основной объект нашего внимания — вид F. alocis.
Таблица 4. Представительство бактерий каждого типа в субгингивальном микробиоме по группам исследования
Table 4. Representation of bacteria of each type in the subgingival microbiome by study group
Тип бактерий Bacteria type | Представительство бактерий в микробиоме, % (медиана (минимум; максимум)) Bacterial representation in the microbiome, % (median (minimum; maximum)) | One-way ANOVA | ||||
здоровые лица healthy patients | пациенты с ХП patients with CP | пациенты с ХП и АТ patients with CP and AT | пациенты с ХП и СД2 patients with CP and T2D | F | p | |
Firmicutes | 29,0 (0,6; 43,3) | 30,2 (1,1; 41,0) | 45,6 (38,3; 52,8) | 31,4 (24,5; 68,8) | 2,162 | 0,126 |
Proteobacteria | 30,2 (15,4; 57,9) | 27,9 (15,5; 29,2) | 18,6 (15,1; 22,2) | 17,2 (6,5; 26,6) | 0,958 | 0,438 |
Bacteroides | 10,4 (0; 31,0) | 11,7 (0,1; 35,0) | 17,5 (5,7; 11,0) | 18,8 (11,8; 28,5) | 0,964 | 0,426 |
Actinobacteria | 15,6 (3,6; 16,2) | 7,3 (5,7; 11,0) | 2,7 (1,5; 3,9) | 15,4 (7,2; 16,9) | 1,211 | 0,340 |
Fusobacteria | 12,6 (2,0; 15,6) | 16,4 (8,0; 18,7) | 7,4 (5,1; 9,7) | 9,8 (3,1; 17,0) | 0,936 | 0,448 |
TM7 | 1,3 (0; 1,8) | 4,2 (2,5; 6,5) | – | 1 (0; 2,1) | 7,879 | 0,013 |
Spirochaetes | 0,9 (0; 2,7) | 2,3 (0,1; 20,0) | 6,1 (5,3; 7,0) | 0,4 (0; 1,1) | 0,799 | 0,522 |
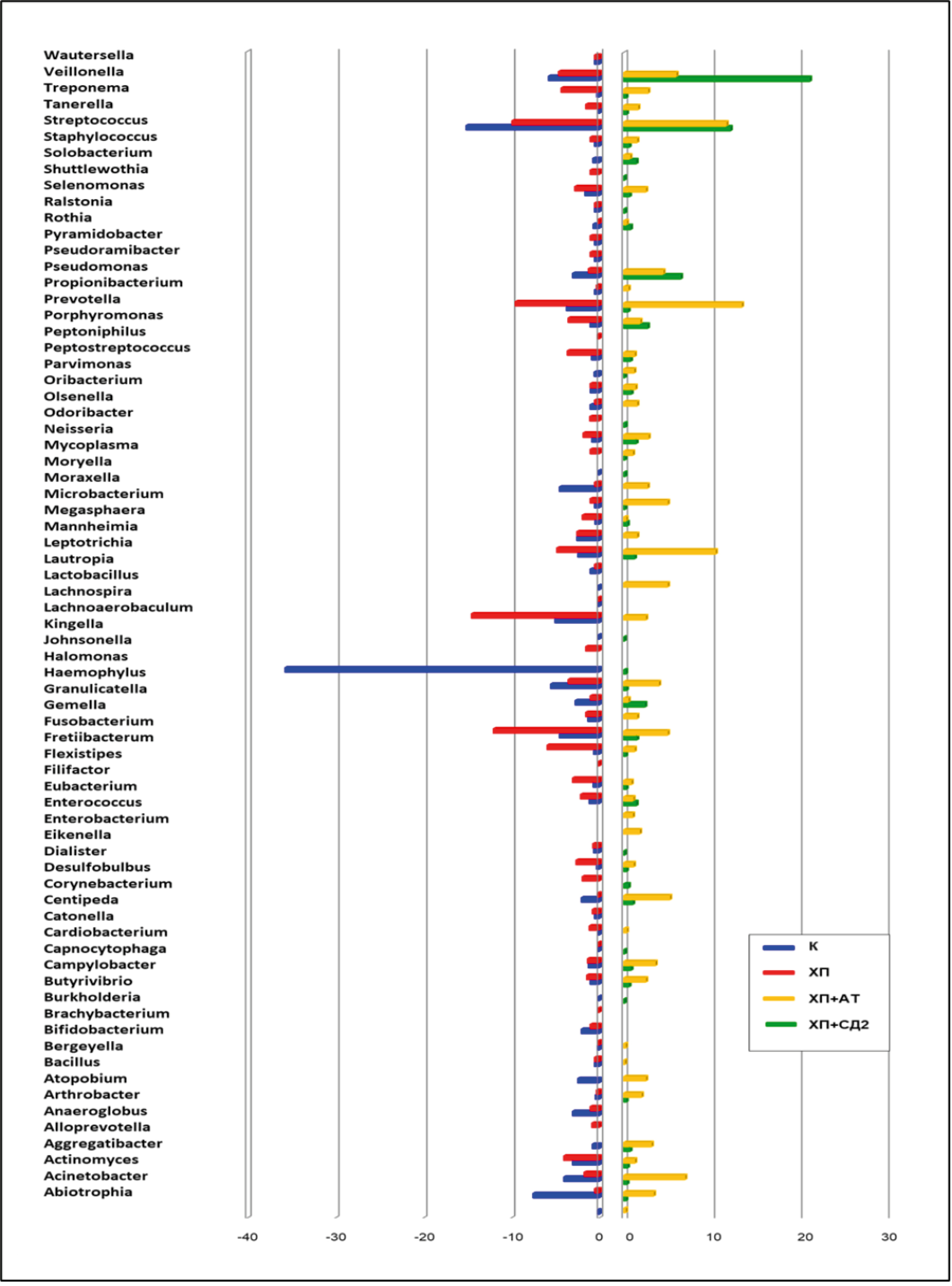
Полученные результаты в сравнительном аспекте касались процентного соотношения отдельных бактерий различной родовой принадлежности в различных группах исследования (рис. 1).
Рис. 1. Представительство наиболее часто встречающихся родов бактерий в субгингивальном микробиоме по группам исследования.
Fig. 1. Representation of the most frequent bacterial genera in the subgingival microbiome by study group.
Во всех образцах субгингивального микробиома, содержащих представителей рода Filifactor и вошедших в данное исследование, этому роду сопутствовали ещё 70 родов бактерий. В тех случаях, когда в источнике литературы имелись данные о видовом составе микробиома, в род Filifactor входил только один вид — F. alocis, причём в разных группах исследования содержание бактерий рода Filifactor было различным. Так, в группе здоровых людей доля этих бактерий (в %) среди представителей других видов с обозначением медианы (минимума; максимума) составляла только 0,66 (0,1; 5,8); в группе ХП без системной патологии — 3 (0,1; 13,0); при ассоциации ХП + AT показатель также был низким — 0,12 (0,05; 0,27); а при ассоциации ХП + СД2 — существенно выше: 1,6 (1,0; 2,3).
Таким образом, бактерии рода Filifactor показывали относительно низкое содержание в составе субгингивального микробиома у здоровых людей и при ассоциации ХП + АТ. При ассоциации ХП + СД2 медиана доли этих бактерий в микробиоме пародонта была в 2,4 раза выше, чем у здоровых людей, а у пациентов с ХП без системной патологии — в 4,5 раза выше. Тем не менее разброс данных по каждой выборке не позволил установить статистически значимые различия при сравнении методом One-way ANOVA: в группах «здоровые лица — ХП» F = 1,910 при р = 0,192; в группах «здоровые лица — ХП + АТ» F = 0,427 при р = 0,527; в группах «здоровые лица — ХП + СД2» F = 0,002 при р = 0,963.
В составе субгингивального микробиома здоровых людей преобладали представители следующих родов: Haemophilus, Streptococcus, Abiotrophia, Granulicatella, Moraxella, Veillonella; у пациентов с ХП без системной патологии — Fusobacterium, Fretibacterium, Lachnoaerobaculum, Prevotella, Streptococcus; при ХП + АТ — Streptococcus, Veillonella, Propionibacterium; при ХП + СД2 — Prevotella, Streptococcus, Leptotrix, Veillonella, Actinomyces.
Все эти данные послужили основой для выполнения корреляционного анализа, который позволил определить возможные связи бактерий рода Filifactor с другими бактериями той же локализации.
Метаанализ корреляционных связей бактерий рода Filifactor c другими родами бактерий в составе субгингивального микробиома в группах исследования
Корреляционный анализ данных в виде парных корреляций проводили путём определения коэффициента корреляции Спирмена (r) и его статистической значимости (р) между бактериями рода Filifactor (вид F. alocis) и остальными бактериями в составе субгингивального микробиома отдельно для каждой группы исследования.
Содержание бактерий рода Filifactor, представленного только одним видом F. alocis, в составе микробиома зубодесневой борозды людей со здоровым пародонтом было относительно небольшим и было корреляционно связано на статистически значимом уровне с бактериями 8 родов (рис. 2, а). С бактериями родов Actinomyces, Brachybacterium, Mogibacterium, которые, как и Filifactor, относятся к грамположительным микроорганизмам, корреляции были положительными, в то время как с остальными 5 родами (Acinetobacter, Alloprevotella, Campylobacter, Miсrobacterium, Moraxella) — отрицательными.
Рис. 2. Корреляционные связи F. alocis c другими бактериями субгингивального микробиома у здоровых людей (а), при ХП (б), ХП + АТ (в), ХП + СД2 (г). DOI: https://doi.org/10.36233/0372-9311-500-2
Диаметр круга, обозначающего родовую принадлежность бактерии, примерно отражает количественное представительство данного рода бактерий среди остальных обнаруженных родов. Сплошные стрелки — положительная корреляция, пунктирные — отрицательная. На рисунках представлены только статистически значимые корреляции при р < 0,05.
Fig. 2. Correlations of F. alocis with other bacteria of the subgingival microbiome in healthy subjects (a), in CP (b), CP + AT (c), CP + T2D (d). DOI: https://doi.org/10.36233/0372-9311-500-2
The diameter of the circle denoting the genus of the bacterium roughly reflects the quantitative representation of this genus of bacteria among the other detected genera. Solid arrows indicate positive correlation, dashed arrows indicate negative correlation. The presented figures show only statistically significant correlations at p < 0.05.
У больных ХП с неотмеченным сопутствующим системным эффектом отмечается явное количественное преобладание бактерий рода Fusobacterium (16,8%), отрицательно корреляционно связанных с бактериями рода Filifactor (рис. 2, б). Кроме того, значительно возрастает доля самого F. alocis в микробиоме и примерно в 2 раза увеличивается число корреляционных связей этого пародонтопатогена с другими бактериями, притом что примерно половина из них являются положительными, обеспечивающими синергизм колонизации биотопа. К числу последних принадлежат бактерии родов Desulfobulbus, Eubacterium, Lachnospira, Mycoplasma, Neisseria, Odoribacter, Propionibacterium, Pseudoramibacter, среди которых в равной степени представлены как грамположительные, так и грамотрицательные микроорганизмы.
Содержание бактерий рода Filifactor при осложнении ХП таким системным заболеванием, как АТ, было минимальным (рис. 2, в). Тем не менее обращает на себя внимание обилие корреляционных пар, рост спектра вовлечённых в них микроорганизмов при явном преобладании положительных корреляций. В составе корреляционных пар появляются такие роды бактерий, как Aggregatibacter, Atopobium, Cardiobacterium, Fretibacterium, Haemophylus, Halomonas, Kingella, Mеgasphera, Peptostreptococcus, Streptococcus, Treponema, Veillonella, среди которых только 3 рода (Atopobium, Peptostreptococcus, Streptococcus) принадлежат к грамположительным микроорганизмам.
На рис. 2, г представлен круг корреляций F. alocis, численность которого была примерно в 10 раз выше, чем при сопутствующем АТ, c родами бактерий, входящими в субгингивальный микробиом при ХП, ассоциированном с СД2. При корреляционном анализе в этом аспекте число парных корреляций падало по сравнению с другими анализируемыми патологическими состояниями до 11 при примерно одинаковом соотношении положительных и отрицательных корреляций. Среди положительных корреляций можно отметить участие грамотрицательных бактерий родов Aggregatibacter, Alloprevotella, Dialister, а среди грамположительных — Atopobium, Eubacterium, Lachnoanaerobaculum.
Обсуждение
Оценивая результаты корреляционного анализа, следует подчеркнуть несколько особенностей. Во всех группах содержание бактерий F. alocis было различным — от минимального при ассоциации ХП и АТ до максимального при ХП без системных эффектов. В составе корреляционных пар не было ни одного рода бактерий, который встречался бы во всех 4 группах. Представленные в нашей работе диаграммы свидетельствуют о существовании многочисленных корреляционных связей F. alocis с другими компонентами микробиома, что в сочетании с данными о наличии у него экзотоксина, как и у A. actinomycetemcomitans [12], позволяет поставить вопрос о его отнесении к пародонтопатогенным видам I порядка.
Один из зарубежных метаанализов, проведённый в рамках международного проекта Human Microbiome Project, показал, что над- и поддесневые зубные отложения (биоплёнка) представлены 14 таксонами микроорганизмов, которые являются самыми многочисленными как по частоте выявления, так и в количественном выражении в составе биоплёнки десны: Actinomyces, Aggregatibacter, Capnocytophaga, Corynebacterium, Haemophilus, Fusobacterium, Neisseria, Prevotella, Porphyromonas, Rothia, Selenomonas, Streptococcus, Veillonella, Wolinella [6].
Исходя из цели метаанализа, представленного в нашей статье, особого внимания заслуживали те бактерии, которые появлялись в составе корреляционных пар: либо только при ХП без системных эффектов; либо только при ассоциации ХП как с АТ, так и с СД2; либо только при АТ или только при СД2. Видовую принадлежность бактерий, связанных корреляционно с F. alocis, установить не удалось, поскольку только единичные из отобранных источников литературы содержали эти сведения, что оказалось недостаточным для выявления статистически значимых корреляций. Только в первую категорию корреляций (ХП без системных эффектов) с бактериями рода Filifactor входили представители 8 родов: Desulfobulbus, Gemella, Granulicatella, Lachnospira, Mycoplasma, Odoribacter, Oribacterium, Pseudoramibacter. Среди указанных родов не было родов бактерий, содержащих общепризнанных пародонтопатогенов. Вторая категория, связанная с проявлением системных эффектов болезней пародонта, включала только 3 рода: Aggregatibacter, Atopobium, Megasphera. При этом представители рода Aggregatibacter, вошедшего в состав статистически значимых корреляционных пар, регистрировались в 58% образцов, а остальные 2 рода — в 42%. В тех источниках, что содержали сведения о видовом составе бактерий субгингивального микробиома, бактерии рода Aggregatibacter включали только один вид — А. аctinomycetemcommitans, который относят к пародонтопатогенам I порядка.
Только при АТ регистрировались корреляционно значимые роды: Cardiobacterium, Fretibacterium, Haemophylus, Halomonas, Kingella, Leptotrichia, Peptostreptococcus, Streptococcus, Treponema, Veillonella. Только при СД2 в составе корреляционных пар отмечены 4 рода: Dialister, Lachnoanaero- baculum, Rothia, Sphingomonas. По видовому составу, где это было обозначено, принадлежности к пародонтопатогенам не было отмечено ни в одном случае.
Заключение
- Проведён сравнительный метаанализ поддесневого микробиома в норме, при ХП и ХП, ассоциированным с коморбидной патологией, охватывающий 1529 здоровых лиц и 2394 пациентов с ХП, который позволил получить принципиально новые данные, подтверждающие различия групп сравнения по составу микробиома. По таксономической характеристике в составе поддесневого микробиома здоровых людей преобладали представители следующих родов: Haemophilus, Streptococcus, Abiotrophia, Granulicatella, Moraxella, Veillonella; у пациентов с ХП без системной патологии — Fusobacterium, Fretibacterium, Lachnoaerobaculum, Prevotella, Streptococcus; при ХП в ассоциации с АТ — Streptococcus, Veillonella, Propionibacterium; при ХП в ассоциации с СД2 — Prevotella, Streptococcus, Leptotriсhia, Veillonella, Actinomyces.
- В результате метаанализа данных по родовой принадлежности бактерий, корреляционно связанных с бактериями рода Filifactor, которые в тех случаях, когда источники содержали сведения о видовом составе бактерий, принадлежали к виду F. alocis, установлено, что развитие ХП, условно не связанного с системной патологией, не позволяло определить значимых корреляций этих бактерий с другими пародонтопатогенами. Вместе с тем уточнено место F. alocis как пародонтопатогенного вида (возможно I порядка), т. к. он практически не встречается в составе нормальной микробиоты, что указывает на его относительно высокую вирулентность.
- В группах пациентов, у которых ХП сопровождался наличием системной патологии — АТ или СД2, помимо ассоциантов-комменсалов, было подтверждено наличие статистически значимых корреляций с родом бактерий Aggregatibacter, содержащим пародонтопатогены I порядка, — A. actinomycetemcomitans. Представители родов Porphyromonas (P. gingivalis) и Tannerella (T. forsythia), по-видимому, имеют самостоятельное значение как пародонтопатогенные виды I порядка, ввиду высоких вирулентных свойств, независимо от симбионтов из других таксонов поддесневого микробиома.
1 National Center for Biotechnology Information. Taxonomy Browser. URL: https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi
Об авторах
Татьяна Викторовна Царева
Российский университет медицины
Email: nikola777@rambler.ru
ORCID iD: 0000-0001-9571-0520
к.м.н., доцент кафедры микробиологии, вирусологии, иммунологии
Россия, МоскваИрина Петровна Балмасова
Российский университет медицины
Email: nikola777@rambler.ru
ORCID iD: 0000-0001-8194-2419
д.м.н., профессор, зав. лаб. патогенеза и методов лечения инфекционных заболеваний Научно-исследовательского медицинского стоматологического института
Россия, МоскваВиктор Николаевич Царев
Российский университет медицины
Автор, ответственный за переписку.
Email: nikola777@rambler.ru
ORCID iD: 0000-0002-3311-0367
д.м.н., профессор, зав. каф. микробиологии, вирусологии, иммунологии
Россия, МоскваСписок литературы
- Кузьмина Э.М., Янушевич О.О., Кузьмина И.Н. Стоматологическая заболеваемость населения России. Эпидемиологическое стоматологическое обследование. М.;2019. Kuz'mina E.M., Yanushevich O.O., Kuz'mina I.N. Dental Morbidity of the Russian Population. Epidemiological Dental Examination. Moscow;2019.
- Abusleme L., Dupuy A.K., Dutzan N., et al. The subgingival microbiome in health and periodontitis and its relationship with community biomass and inflammation. ISME J. 2013;7(5): 1016–25. DOI: https://doi.org/10.1038/ismej.2012.174
- Kassebaum N.J., Bernabé E., Dahiya M., et al. Global burden of severe periodontitis in 1990–2010: a systematic review and meta-regression. J. Dent. Res. 2014;93(11):1045–53. doi: https://doi.org/10.1177/0022034514552491
- Könönen E., Gursoy M., Gursoy U.K. Periodontitis: a multifaceted disease of tooth-supporting tissues. J. Clin. Med. 2019;8(8):1135. DOI: https://doi.org/10.3390/jcm8081135
- Tonetti M.S., Jepsen S., Jin L., Otomo-Corgel J. Impact of the global burden of periodontal diseases on health, nutrition and wellbeing of mankind: a call for global action. J. Clin. Periodontol. 2017;44(5):456–62. doi: https://doi.org/10.1111/jcpe.12732
- Балмасова И.П., Царев В.Н., Янушевич О.О. и др. Микроэкология пародонта. Взаимосвязь локальных и системных эффектов. М.;2021. Balmasova I.P., Tsarev V.N., Yanushevich O.O., et al. Microecology of Periodontal Disease. The Relationship of Local and Systemic Effects. Moscow;2021. EDN: https://elibrary.ru/myzmbu
- Bui F.Q., Almeida-da-Silva C.L.C., Huynh B., et al. Association between periodontal pathogens and systemic disease. J. Biomed. Sci. 2019;42(1):27–35. doi: https://doi.org/10.1016/j.bj.2018.12.001
- Царев В.Н., Николаева Е.Н., Ипполитов Е.В. Пародонтопатогенные бактерии – основной фактор возникновения и развития пародонтита. Журнал микробиологии, эпидемиологии и иммунобиологии. 2017;(5):101–12. Tsarev V.N., Nikolaeva E.N., Ippolitov E.V. Periodontophatogenic bacteria of the main factors of emergence and development of periodontitis. Journal of Microbiology, Epidemiology and Immunobiology. 2017;(5):101–12. doi: https://doi.org/10.36233/0372-9311-2017-5-101-112 EDN: https://elibrary.ru/ctbcar
- Nikolaeva E.N., Tsarev V.N., Tsareva T.V., et al. Interrelation of cardiovascular diseases with anaerobic bacteria of subgingival biofilm. Contemp. Clin. Dent. 2019;10(4):637–42. doi: https://doi.org/10.4103/ccd.ccd_84_19
- Socransky S.S., Haffajee A.D., Cugini M.A., et al. Microbial complexes in subgingival plaque. J. Clin. Periodontol. 1998;25(2):134–44. doi: https://doi.org/10.1111/j.1600-051x.1998.tb02419.x
- Ahmed H.J., Svensson J.A., Cope L.D., et al. Prevalence of cdtABC genes encoding cytolethaldistanding toxin among Haemophilus ducreyi and Actinobacillus actinomycetemcomitans strains. J. Med. Microbiol. 2001;50(10):860–4. doi: https://doi.org/10.1099/0022-1317-50-10-860
- Ozuna H., Snider I., Belibasakis G.N., et al. Aggregatibacter actinomycetemcomitans and Filifactor alocis: Two exotoxin-producing oral pathogens. Front. Oral Health. 2022;3:981343. DOI: https://doi.org/10.3389/froh.2022.981343
- Ушаков Р.В., Царев В.Н. Антимикробная терапия в стоматологии: принципы и алгоритмы. М.;2019. Ushakov R.V., Tsarev V.N. Antimicrobial Therapy in Dentistry: Principles and Algorithms. Moscow;2019.
- Rafiei M., Kiani F., Sayehmiri K., et al. Prevalence of anaerobic bacteria (P. gingivalis) as major microbial agent in the incidence periodontal diseases by meta-analysis. J. Dent. (Shiraz). 2018;19(3):232–42.
- Hiranmayi K.V., Sirisha K., Ramoji Rao M.V., Sudhakar P. Novel pathogens in periodontal microbiology. J. Pharm. Bioallied Sci. 2017;9(3):155–63. doi: https://doi.org/10.4103/jpbs.jpbs_288_16
- Moffatt C.E., Whitmore S.E., Griffen A.L., et al. Filifactor alocis interactions with gingival epithelial cells. J. Mol. Oral Microbiol. 2011;26(6):365–73. doi: https://doi.org/10.1111/j.2041-1014.2011.00624.x
- Wang Q., Wright C.J., Dingming H., et al. Oral community interactions of Filifactor alocis in vitro. PLoS One. 2013;8(10):e76271. DOI: https://doi.org/10.1371/journal.pone.0076271
- Aja Е., Mishra А., Dou Н., Fletcher H.M. Role of the Filifactor alocis hypothetical protein FA519 in oxidative stress resistance. Microbiol. Spectr. 2021;9(3):e0121221. doi: https://doi.org/10.1128/spectrum.01212-21
- Царева Т.В., Янушевич О.О., Царев В.Н., Балмасова И.П. Бактерии рода Filifactor у больных пародонтитом и сахарным диабетом по данным метагеномного анализа микробиома пародонта. Журнал микробиологии, эпидемиологии и иммунобиологии. 2023;100(6):485–94. Tsareva T.V., Yanushevich O.O., Tsarev V.N., Balmasova I.P. Bacteria of genus Filifactor in patients with periodontitis and type 2 diabetes in accordance with metagenomic analysis of the periodontal microbiome. Journal of Microbiology, Epidemiology and Immunobiology. 2023;100(6):485–94. doi: https://doi.org/10.36233/0372-9311-428 EDN: https://elibrary.ru/yqoxxk
- Янушевич О.О., Царёв В.Н., Балмасова И.П. и др. Первый опыт применения отечественного диагностического набора генетических праймеров для выявления нового пародонтопатогена Filifactor alocis и его ассоциации с Porphyromonas gingivalis. Клиническая лабораторная диагностика. 2022;67(12):744–8. Yanushevich O.O., Tsarev V.N., Balmasova I.P., et al. The first experience of using a domestic diagnostic set of genetic primers to identify a new periodontopathogen Filifactor alocis and its association with Porphyromonas gingivalis. Clinical Laboratory Diagnostics. 2022;67(12):744–8. DOI: https://doi.org/10.51620/0869-2084-2022-67-12-744-748 EDN: https://elibrary.ru/djlzfu
- Янушевич О.О., Царев В.Н., Николаева Е.Н. и др. Первый отечественный опыт выявления ассоциации анаэробных бактерий Filifactor alocis и Porphyromonas gingivalis молекулярно-биологическими методами при заболеваниях пародонта и коморбидной патологии (сравнительное исследование). Вестник Российской академии медицинских наук. 2022;77(6):437–46. Yanushevich O.O., Tsarev V.N., Nikolaeva E.N., et al. The first domestic experience of detecting the association of anaerobic bacteria Filifactor alocis and Porphyromonas gingivalis by molecular biological methods in periodontal diseases and comorbid pathology (comparative research). Annals of the Russian Academy of Medical Sciences. 2022;77(6):437–46. DOI: https://doi.org/10.15690/vramn2262 EDN: https://elibrary.ru/pzajok
- Kumar P.S., Griffen A.L., Moeschberger M.L., et al. Identification of candidate periodontal pathogens and beneficial species by quantitative 16S clonal analysis. J. Clin. Microbiol. 2005;43(8):3944–55. doi: https://doi.org/10.1128/jcm.43.8.3944-3955.2005
- Griffen A.L., Beall C.J., Campbell J.H., et al. Distinct and complex bacterial profiles in human periodontitis and health revealed by 16S pyrosequencing. ISME J. 2012;6(6):1176–85. DOI: https://doi.org/10.1038/ismej.2011.191
- Liu B., Faller L.L., Klitgord N., et al. Deep sequencing of the oral microbiome reveals signatures of periodontal disease. PLoS One. 2012;7(6):e37919. doi: https://doi.org/10.1371/journal.pone.0037919
- Costalonga M., Herzberg M.C. The oral microbiome and the immunobiology of periodontal disease and caries. Immunol. Lett. 2014;162(2 Pt. A):22–38. doi: https://doi.org/10.1016/j.imlet.2014.08.017
- Filho W.S.S., Casarin R.C.V., Junior E.L.N., et al. Microbial diversity similarities in periodontal pockets and atheromatous plaques of cardiovascular disease patients. PLoS One. 2014;9(10):e109761. doi: https://doi.org/10.1371/journal.pone.0109761
- Lourenço T.G.B., Heller D., da Silva-Boghossian C.M., et al. Microbial signature profiles of periodontally healthy and diseased patients. J. Clin. Periodontol. 2014;41(11):1027–36. doi: https://doi.org/10.1111/jcpe.12302
- Pérez-Chaparro P.J., Gonçalves C, Figueiredo L.C., et al. Newly identified pathogens associated with periodontitis. J. Dent. Res. 2014;93(9):846–58. doi: https://doi.org/10.1177/0022034514542468
- Park O.J., Yi H., Jeon J.H., et al. Pyrosequencing analysis of subgingival microbiota in distinct periodontal conditions. J. Dent. Res. 2015;94(7):921–7. doi: https://doi.org/10.1177/0022034515583531
- Ziganshina E.E., Sharifullina D.M., Lozhkin A.P., et al. Bacterial communities associated with atherosclerotic plaques from Russian individuals with atherosclerosis. PLoS One. 2016;11(10):e0164836. doi: https://doi.org/10.1371/journal.pone.0164836
- Demmer R.T., Breskin A., Rosenbaum M., et al. The subgingival microbiome, systemic inflammation and insulin resistance: The oral infections, glucose intolerance and insulin resistance study. J. Clin. Periodontol. 2017;44(3):255–65. doi: https://doi.org/10.1111/jcpe.12664
- Ganesan S.M., Joshi V., Fellows M., et al. A tale of two risks: smoking, diabetes and the subgingival microbiome. ISME J. 2017;11(9):2075–89. doi: https://doi.org/10.1038/ismej.2017.73.
- Long J., Cai Q., Steinwandel M., et al. Association of oral microbiome with type 2 diabetes risk. J Periodontal Res. 2017;52(3):636–43. https://doi.org/10.1111/jre.12432
- Graves D.T., Corrêa J.D., Silva T.A. The oral microbiota is modified by systemic diseases. J. Dent. Res. 2019;98(2): 148–56. DOI: https://doi.org/10.1177/0022034518805739
- Rodríguez-Hernández A.P., de Lourdes Márquez-Corona M., Pontigo-Loyola A.P., et al. Subgingival microbiota of Mexicans with type 2 diabetes with different periodontal and metabolic conditions. Int. J. Environ. Res. Public Health. 2019;16(17):3184. doi: https://doi.org/10.3390/ijerph16173184
- Saeb A.T.M., Al-Rubeaana K.A., Aldosaryb K., et al. Relative reduction of biological and phylogenetic diversity of the oral microbiota of diabetes and pre-diabetes patients. Microb. Pathog. 2019;128:215–29. doi: https://doi.org/10.1016/j.micpath.2019.01.009
- Matsha T.E., Prince Y., Davids S., et al. Oral microbiome signatures in diabetes mellitus and periodontal disease. J. Dent. Res. 2020;99(6):658–65. doi: https://doi.org/10.1177/0022034520913818
- Bouzid F., Gtif I., Alfadhli S., et al. A potential oral microbiome signature associated with coronary artery disease in Tunisia. Biosci. Rep. 2022;42(7):BSR20220583. doi: https://doi.org/10.1042/BSR20220583
- Curia M.C., Pignatelli P., D’Antonio D.L., et al. Oral Porphyromonas gingivalis and Fusobacterium nucleatum abundance in subjects in primary and secondary cardiovascular prevention, with or without heterozygous familial hypercholesterolemia. Biomedicines. 2022;10(9):2144. doi: https://doi.org/10.3390/biomedicines10092144
- Rao A., Lokesh J., D'Souza C., et al. Metagenomic analysis to uncover the subgingival and atherosclerotic plaque microbiota in patients with coronary artery disease. Indian J. Microbiol. 2023;63(3):281–90. doi: https://doi.org/10.1007/s12088-023-01082-9
- Kato-Kogoe N., Sakaguchi S., Kamiya K., et al. Characterization of salivary microbiota in patients with atherosclerotic cardiovascular disease: а case-control study. J. Atheroscler. Thromb. 2022;29(3):403–21. doi: https://doi.org/10.5551/jat.60608
- Solbiati J., Frias-Lopez J. Metatranscriptome of the oral microbiome in health and disease. J. Dent. Res. 2018;97(5):492–500. DOI: https://doi.org/10.1177/0022034518761644
- Figuero E., Sánchez-Beltrán M., Cuesta-Frechoso S., et al. Detection of periodontal bacteria in atheromatous plaques by nested polymerase chain reaction. J. Periodontol. 2011;82(10): 1469–77. DOI: https://doi.org/10.1902/jop.2011.100719
- Ohki T., Itabashi Y., Kohno T., et al. Detection of periodontal bacteria in thrombi of patients with acute myocardial infarction by polymerase chain reaction. Am. Heart J. 2012;163(2):164–7. DOI: https://doi.org/10.1016/j.ahj.2011.10.012
- Calandrini C.A., Ribeiro A.C., Gonnelli A.C., et al. Microbial composition of atherosclerotic plaques. Oral Dis. 2014; 20(3):128–34. DOI: https://doi.org/10.1111/odi.12205
Дополнительные файлы