Генотипический портрет SARS-CoV-2 на территории Приморского края в период пандемии COVID-19
- Авторы: Попова А.Ю.1, Щелканов М.Ю.2,3, Крылова Н.В.2,3, Белик А.А.2, Семейкина Л.М.2,4, Запорожец Т.С.2, Смоленский В.Ю.1, Персиянова Е.В.2, Просянникова М.Н.4, Белов Ю.А.2,3, Иунихина О.В.2,3, Потт А.Б.2, Хомичук Т.Ф.4, Симакова А.И.5, Абрамова С.А.2, Романова О.Б.4, Детковская Т.Н.6, Крыжановский С.П.7, Беседнова Н.Н.2,7
-
Учреждения:
- Федеральная служба по надзору в сфере защиты прав потребителей и благополучия человека
- НИИ эпидемиологии и микробиологии им. Г.П. Сомова
- Дальневосточный федеральный университет
- Центр гигиены и эпидемиологии в Приморском крае
- Тихоокеанский государственный медицинский университет
- Управление Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека в Приморском крае
- Медицинское объединение Дальневосточного отделения Российской академии наук
- Выпуск: Том 101, № 1 (2024)
- Страницы: 19-35
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/18530
- DOI: https://doi.org/10.36233/0372-9311-497
- EDN: https://elibrary.ru/pujffa
- ID: 18530
Цитировать
Аннотация
Введение. Пандемия COVID-19, этиологически связанная с SARS-CoV-2, стала самой продолжительной для острого респираторного заболевания и оказала заметное воздействие на демографию, экономику и политику в мировом масштабе. Уроки этой пандемии имеют важное значение для устойчивого развития человеческого общества. Подробный анализ приобретённого опыта по контролю эпидемического процесса должен включать детализацию эволюции возбудителя вплоть до регионального уровня.
Цель исследования — установление закономерностей эпидемического процесса COVID-19 в связи со сменой геновариантов возбудителя на территории Приморского края.
Материалы и методы. Проведён ретроспективный эпидемиологический анализ заболеваемости COVID-19 и динамики циркуляции геновариантов SARS-CoV-2 в период пандемии этого заболевания (11.03.2020–05.05.2023) на территории Приморского края. Использованы данные Управления Роспотребнадзора по Приморскому краю, Центра гигиены и эпидемиологии в Приморском крае, НИИ эпидемиологии и микробиологии им. Г.П. Сомова Роспотребнадзора и Российской платформы агрегации информации о геномах вирусов (VGARus), в частности 1055 нуклеотидных последовательностей полноразмерных геномов SARS-CoV-2 из Приморского края, среди которых 553 были секвенированы в НИИ эпидемиологии и микробиологии им. Г.П. Сомова.
Результаты. При анализе эпидемической динамики COVID-19 в Приморском крае (2020–2023 гг.) выделены 7 подъёмов заболеваемости с различными клинико-эпидемиологическими проявлениями в зависимости от геновариантов возбудителя. В начале пандемии в Приморском крае, как и по всей России, доминировали Ухань-подобные варианты SARS-CoV-2, в дальнейшем — геноварианты Delta и Omicron. К концу апреля – началу мая 2023 г. доля субвариантов Omicron (XBB.1.9.2 и XBB.1.16) в Приморском крае была выше, чем в среднем по России, и сопоставима с таковой в сопредельных странах (Республика Корея и Япония).
Заключение. В связи с продолжающейся эволюцией SARS-CoV-2, возможностью появления новых патогенов, особенностями географического расположения и политико-экономического значения Приморского края необходимо последовательно совершенствовать региональные возможности оперативного молекулярно-вирусологического мониторинга.
Ключевые слова
Полный текст
Введение
Коронавирусное заболевание 2019 г. (Coronavirus disease 2019 — COVID-19) этиологически связано с коронавирусом тяжёлого острого респираторного синдрома 2-го типа (Severe acute respiratory syndrome coronavirus 2 — SARS-CoV-2; Nidovirales: Coronaviridae, Betacoronavirus, подрод Sarbecovirus) [1, 2] и возникло в результате преодоления возбудителем межвидового барьера и проникновения из популяций летучих мышей (Chiroptera, Microchiroptera) в человеческую популяцию [3, 4]. COVID-19 вызвал наиболее продолжительную (11.03.2020–05.05.2023, или 1150 дней), наиболее масштабную (свыше 750 млн подтверждённых случаев) и одну из самых смертоносных (порядка 6 млн летальных исходов) пандемий острого респираторного заболевания в истории человечества [3, 5]. Пандемия COVID-19 стала первой, связанной с коронавирусом рукокрылых (Chiroptera) [1, 6] — второго по массовости после грызунов отряда млекопитающих, обладающих рядом уникальных эволюционных особенностей [7–9], эпидемиологическое значение которых стало понятно лишь в начале XXI в. после эпидемий, вызванных коронавирусами тяжёлого острого респираторного синдрома (Nidovirales: Coronaviridae, Betacoronavirus, подрод Sarbecovirus) [1, 10] и Ближневосточного респираторного синдрома (Nidovirales: Coronaviridae, Betacoronavirus, подрод Merbecovirus) [11, 12].
Пандемия COVID-19 стимулировала широкое внедрение молекулярно-генетических [13, 14] и информационных [15, 16] технологий в практику противоэпидемических мероприятий. В условиях деградации широких международных связей в начале 2020-х гг., усугубившейся пандемией, и падения авторитета международных организаций, включая Всемирную организацию здравоохранения (ВОЗ), Российская Федерация была вынуждена развернуть собственную национальную платформу молекулярно-генетических данных — Virus genome aggregator of Russia (VGARus), разработанную Федеральной службой по надзору в сфере защиты прав потребителей и благополучия человека (Роспотребнадзор). VGARus была введена в эксплуатацию на базе Центрального НИИ эпидемиологии Роспотребнадзора Распоряжением Правительства РФ № 448 от 23.03.2021. К концу 2023 г. в системе зарегистрировано более 150 организаций, осуществляющих её пополнение; в базе данных содержатся свыше 300 тыс. нуклеотидных последовательностей SARS-CoV-2, из которых более половины — полноразмерные вирусные геномы [14, 17]. Таким образом, в отличие даже от последней пандемии гриппа А (H1N1 pdm09) (Articulavirales: Orthomyxoviridae, Alphainfluenzavirus) (2009–2010 гг.) [18], когда молекулярно-генетические методы активно использовались для контроля инфекционного процесса, пандемия COVID-19 стала первым глобальным эпидемическим процессом, который детально характеризовался с помощью данных полногеномного секвенирования «в режиме реального времени» (по крайней мере — в развитых странах) [14, 17, 19].
Россия, имеющая самую большую в мире территорию, обладает колоссальным региональным разнообразием природно-климатических и социально-демографических условий, что необходимо учитывать в процессе эпидемиологического анализа динамики инфекционных заболеваний. Например, Приморский край (ПК), расположенный на крайнем юге российского Дальнего Востока (практически — на широте Крымского полуострова), характеризуется уникальным муссонным умеренным климатом, формируемым широтной климатической аномалией вследствие проникновения из Татарского пролива холодного Приморского течения. ПК обеспечивает удобный выход через незамерзающие порты (Владивосток, Восточный, Зарубино, Находка, Посьет) к динамично развивающемуся Азиатско-Тихоокеанскому региону и традиционно тесно связан с северо-восточными провинциями Китайской Народной Республики всеми видами транспорта. Административный центр ПК Владивосток, являющийся столицей Дальневосточного федерального округа, хорошо узнаваем как в России, так и за её пределами благодаря статусу свободного порта и наличию одного из крупнейших в азиатской части страны Дальневосточного федерального университета.
Цель работы — молекулярно-генетический анализ закономерностей развития эпидемического процесса COVID-19 на территории ПК в период пандемии этого заболевания (2020–2023 гг.).
Материалы и методы
Назофарингеальные смывы для проведения ПЦР-тестирования и получения вирусных штаммов отбирали у лиц, проживавших на территории ПК. Отбор биологического материала осуществлялся квалифицированным персоналом, прошедшим предварительное обучение и регулярную проверку навыков на риноларингологических симуляторах [20, 21]. Исследование проводилось при добровольном информированном согласии пациентов. Протокол исследования одобрен Этическим комитетом НИИ эпидемиологии и микробиологии им. Г.П. Сомова (протокол № 2 от 16.11.2021).
Индикация РНК SARS-CoV-2 осуществлялась с помощью метода обратной транскрипции с последующей полимеразной цепной реакцией в режиме реального времени (ОТ-ПЦР-РВ): выделение РНК проводили с использованием комплекса реагентов «М-Сорб-НК» (НПФ «Синтол») ручным способом или (при больших количествах образцов) на автоматической станции «AutoPure 96» («Hangzhou Allsheng Instruments Co.»), набора реагентов «ОТ-ПЦР-РВ-SARS-CoV-2» (НПФ «Синтол»). Положительные образцы, предназначенные для последующего использования в исследовательских целях, хранились при –20ºС без разрыва холодовой цепи.
Секвенирование полноразмерного генома SARS-CoV-2 проводили для положительных образцов с пороговым циклом при первичном ПЦР-тестировании Сt ≤ 25 на технологической платформе Nanopore [22, 23] согласно протоколу ARTIC SARS-CoV-2 v3: обратную транскрипцию осуществляли с помощью набора «Midnight RT-PCR Expansion»; ампликоны получали и кодировали с использованием 29 пар перекрывающихся праймеров и «Rapid Barcoding Kit 96» (SQK-RBK110.96); очистку кДНК проводили на магнитных частицах «AMPure XP beads»; полученные геномные библиотеки секвенировали на приборе «MinION» с использованием ячеек «FLO-MIN106 R9.4.1» («Oxford Nanopore Technologies»). Полученные данные в формате FAST5 (программный пакет «MinKNOW» переводили в формат FASTQс помощью программы «Guppy v. 6.3.8»). Сборку геномов SARS-CoV-2 в формате FASTA проводили путём выравнивания относительно референсных последовательностей из VGARus с использованием программного пакета «Epi2me v. 22» и модуля «ARTIC v. 1». Для оценки качества собранных последовательностей и распределения геномов по линиям использовали сервисы «Nextclade» и «Pangolin COVID-19 Lineage Assigner v. 4.3». Последовательности, в которых количество нераспознанных или неоднозначных нуклеотидов составляло более 10% полногеномной последовательности SARS-CoV-2, были исключены из выборки для анализа.
Филогенетический анализ нуклеотидных последовательностей проводили после множественного выравнивания с использованием программы «MAFFT v. 7.475» методом «ближайших соседей» [24] средствами программного пакета «MEGA v. 11.0.13» при уровне bootstrap-поддержки в 1000 повторов. Визуализацию полученных данных осуществляли с помощью сервиса «iTOL v. 6».
Изоляция штаммов SARS-CoV-2 проводилась из ОТ-ПЦР-РВ-положительных образцов назофарингеальных смывов, осветлённых низкоскоростным центрифугированием и фильтрованных через насадки «Millex» («Merck») с диаметром пор 0,22 мкм, на модели клеточной линии Vero E6 (почки африканской зелёной мартышки), которую культивировали в пластиковых пробирках («Nunc») со скошенным дном площадью 5,5 см2 в среде DMEM («БиолоТ») с добавлением 1% эмбриональной телячьей сыворотки («ПанЭко»), 100 ЕД/мл гентамицина при 37ºС в атмосфере 5% СО2 [25, 26]. Эффективность изоляции вируса оценивали по снижению порогового цикла ПЦР в 3 последовательных пассажах. На 5-е сутки после инфицирования культуральную жидкость отбирали для постановки ОТ-ПЦР-РВ (после 3-го пассажа дополнительно проводили определение lg 50% инфекционной дозы тканевой культуры (lg TCID50) с помощью МТТ-теста).
МТТ-тест использовали для оценки выживаемости клеток Vero E6 под действием различных разведений вируса при оценке инфекционного титра штаммов SARS-CoV-2 после 3-го пассажа. Сущность этого метода заключается в способности жизнеспособных клеток превращать хорошо растворимый жёлтый бромид 3-(4,5-диметилтиазол-2-ил)-2,5-дифенилтетразолия (МТТ) в нерастворимые внутриклеточные кристаллы (E,Z)-5(4,5-диметилтиазол-2-ил)-1,3-дифенилформазана (МТТ-формазана) под действием внутриклеточных дегидрогеназ [27, 28]:
На 5-е сутки после заражения в лунки с клетками 96-луночного планшета («Merck») добавляли по 20 мкл раствора 5 мг/мл МТТ («Sigma-Aldrich»); инкубировали при 37ºC в атмосфере с 5% CO2 в течение 2 ч; после удаления культуральной жидкости в лунки вносили по 150 мкл изопропилового спирта, подкисленного 0,4 М HCl; оптическую плотность в лунке определяли при 540 нм (вблизи максимума поглощения МТТ-формазана) с вычетом фонового значения при 620 нм с помощью планшетного ридера («Labsystems»). Уровень цитопатогенности (cytopathogenicity level — CPL) штамма в конкретном разведении вычисляли по формуле:
,
где Dv — оптическая плотность инфицированного образца; D0 — оптическая плотность неинфицированной клеточной культуры.
Инфекционный титр штамма определяли как предельное разведение исходной вирусосодержащей жидкости, зафиксированное в эксперименте, для которого CPL > 0 (неравенство нулю оценивалось в t-тесте Стьюдента при уровне значимости p = 95% и 8-кратных повторах каждого разведения вирусосодержащей жидкости).
Эпидемиологический анализ заболеваемости COVID-19 в ПК с 20.04.2020 по 30.06.2023 проводили на основе данных Управления Роспотребнадзора по Приморскому краю, Центра гигиены и эпидемиологии в Приморском крае1, в России — по данным официального сайта Роспотребнадзора2 и российского информационного портала «Стопкоронавирус»3. Использовали информацию о распространении генетических вариантов SARS-CoV-2 на территории России из обзорно-аналитической работы [17, 19] и российской базы данных VGARus, в странах Восточной Азии — из международной базы данных GISAID4.
Статистическую обработку данных осуществляли с помощью следующих функций из программного пакета «MS Excel 2010»: объём анализируемой выборки (n); среднее арифметическое (М); стандартная ошибка среднего (m); медиана (Me); нижний (25%) квартиль (LQ); верхний (75%) квартиль (UQ); интерквартильный размах (LQ–UQ). Нормальность распределения количественных признаков проверяли с использованием W-критерия Шапиро–Уилка; t-критерия Стьюдента (при уровне значимости альтернативной гипотезы р = 0,05); U-критерия Манна–Уитни [29, 30].
Метод главных компонент как способ поиска подпространства меньшей размерности, в проекции на которое исходные данные имеют наименьший разброс квадратичных отклонений [31, 32], применяли с помощью программных пакетов «Statistica v. 7.0» («StatSoft Inc.») и «Mathworks Matlab R2007b» («MathWorks Inc.»).
Результаты
Индикация РНК SARS-CoV-2 в назофарингеальных смывах в период пандемии COVID-19 на территории ПК проводилась в 24 лабораториях (в том числе 5 частных): было проведено 3 007 009 ОТ-ПЦР-РВ-тестирований, из которых 399 821 (13,3%) оказались положительными5. Получаемая информация интегрировалась для оперативного анализа в эпидемиологических подразделениях Центра гигиены и эпидемиологии в Приморском крае и Управления Роспотребнадзора по Приморскому краю.
Полногеномные нуклеотидные последовательности SARS-CoV-2, полученные из образцов биологического материала, собранного на территории ПК в период пандемии COVID-19, представлены в табл. 1. Помимо 553 геномов, секвенированных в НИИ эпидемиологии и микробиологии им. Г.П. Сомова в рамках реализации Федерального проекта «Санитарный щит страны — безопасность для здоровья», ещё 502 генома секвенированы в других научных учреждениях, в которые были направлены ОТ-ПЦ-РВ-положительные образцы из Центра гигиены и эпидемиологии в Приморском крае в соответствии с Приказом Роспотребнадзора6.
Таблица 1. Число полногеномных последовательностей SARS-CoV-2, обнаруженных на территории ПК в 2020–2023 гг., в базе данных VGARus
Table 1. Data on full-genome sequences of SARS-CoV-2 detected in the territory of Primorsky Krai (2020–2023) in the VGARus database
Год Year | НИИ эпидемиологии и микробиологии им. Г.П. Сомова (Владивосток) G.Р. Somov Institute of Epidemiology and Microbiology (Vladivostok) | Государственный научный центр вирусологии и биотехнологии «Вектор» (Новосибирск) State Research Center of Virology and Biotechnology “Vector" (Novosibirsk) | НИИ гриппа (Санкт-Петербург) Institute of Influenza (St.-Petersburg) | Всего Total |
2020 | 173 | 2 | 23 | 198 |
2021 | 90 | 11 | 1 | 102 |
2022 | 105 | 158 | 306 | 569 |
2023 | 185 | 0 | 1 | 186 |
Итого | Total | 553 | 171 | 331 | 1055 |
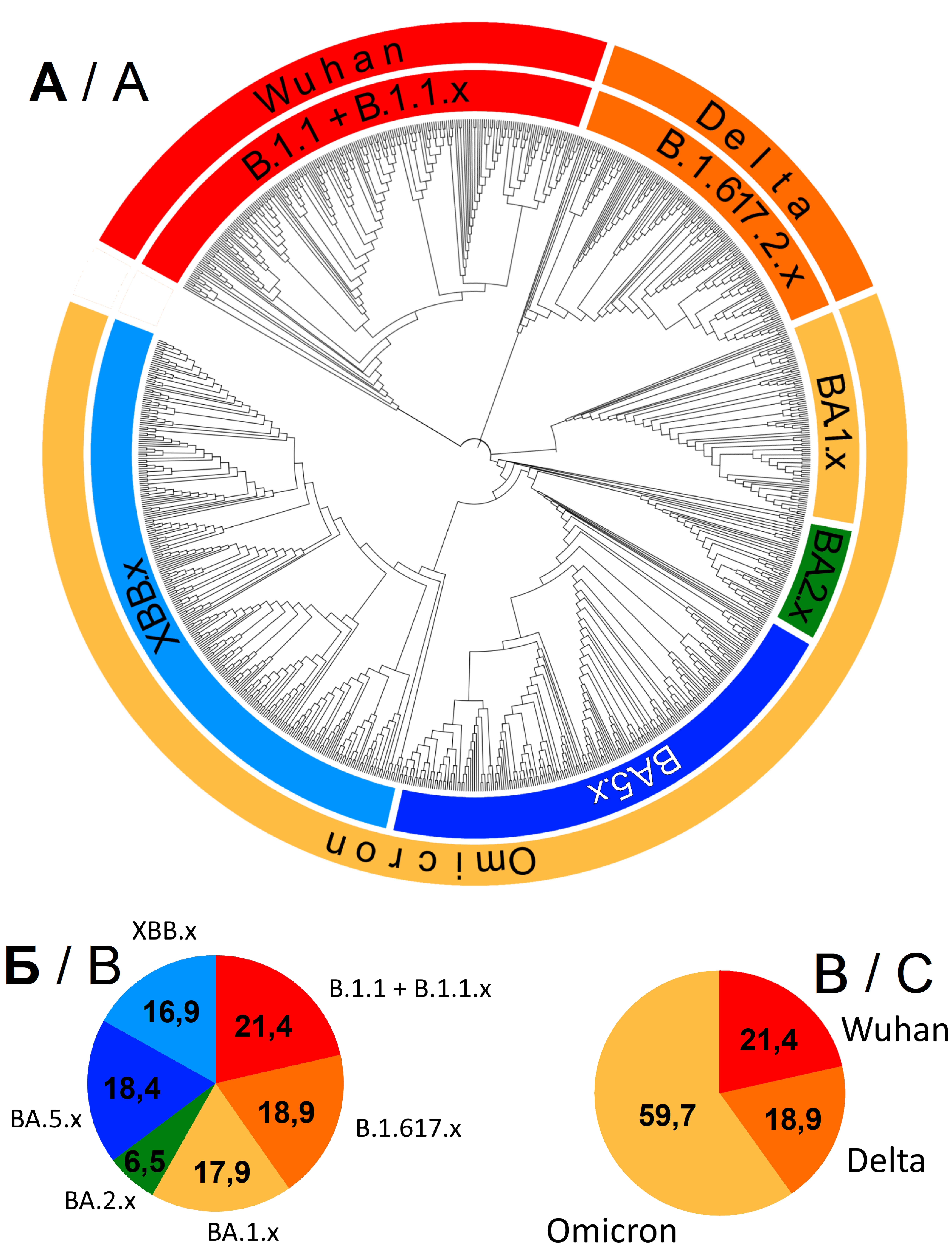
Результаты филогенетического анализа полноразмерных геномов SARS-CoV-2 с территории ПК (табл. 1), представленные на рис. 1, а, свидетельствуют о дифференцировке на геноварианты по классификации Pango (рис. 1, б) и ВОЗ (рис. 1, в).
Рис. 1. Генотипирование 804 полногеномных нуклеотидных последовательностей SARS-CoV-2, выявленных в ПК в 2020–2023 гг. а — филогенетическое древо; б — распределение по генетическим линиям Pango; в — распределение по геновариантам ВОЗ.
Fig. 1. Genotyping of 804 full-genome SARS-CoV-2 nucleotide sequences identified in Primorsky Krai (2020–2023). a — phylogenetic tree; b — distribution according to Pango genetic lines; c — distributions according to WHO genovariants.
Эпидемическая динамика COVID-19 в связи со сменой геновариантов SARS-CoV-2 на территории ПК (2020–2023 гг.) представлена в табл. 2 и на рис. 2: как и в целом по России, подъём заболеваемости COVID-19 за период пандемии этого заболевания (11.03.2020–05.05.2023), начавшийся с крупных мегаполисов [17, 19], имел выраженный волнообразный характер с 7 эпидемическими периодами.
Таблица 2. Динамика эпидемии COVID-19 в ПК в сравнении с Россией в целом в 2020–2023 гг.
Table 2. Characteristics of epidemic dynamics of COVID-19 in Primorsky Krai in comparison with a whole Russian Federation (2020–2023)
Период Period | Территория Territory | Сроки эпидемических периодов Dates of epidemic periods | Заболеваемость на 100 тыс. населения Incidence (per 100 000 population) | Клиническая форма Clinical form | ||||||||||||||
начало | beginning | конец | end | продолжительность, недели duration, weeks | заболеваемость в начале периода incidence in the beginning of the period | заболеваемость в конце периода incidence in the end of the period | максимум maximum | M | m | Me | LQ | UQ | общее число случаев total number of incidences | тяжёлая, % severe, % | средней тяжести, % average severity, % | лёгкая и бессимптомная, % mild and asymptomatic, % | ||||
дата date | номер недели number of a week | дата date | номер недели number of a week | |||||||||||||||
I | ПК | PK | 20.04.2020 | 17 | 20.09.2020 | 38 | 22 | 12,83 | 2,65 | 41,8 | 27,6 | 1,7 | 28,2 | 21,1 | 32,6 | 606,6 | 2,1 | 31,5 | 66,4 |
РФ | RF | 30.03.2020 | 13 | 30.08.2020 | 36 | 22 | 19,6 | 25,6 | 54,5 | 29,8 | 3,0 | 29,8 | 25,1 | 38,8 | 654,9 | 4,5 | 47,7 | 47,8 | |
II | ПК | PK | 21.09.2020 | 39 | 09.05.2021 | 18 | 33 | 28,7 | 9,5 | 91,6 | 54,0 | 5,4 | 54,9 | 24,2 | 87,2 | 1783,4 | 5,8 | 32,2 | 62 |
РФ | RF | 31.08.2020 | 36 | 09.05.2021 | 18 | 36 | 28,1 | 41,3 | 154,9 | 73,4 | 6,2 | 60,6 | 42,5 | 101,7 | 2641,9 | 3,1 | 41,6 | 55,3 | |
III | ПК | PK | 10.05.2021 | 19 | 03.10.2021 | 39 | 21 | 11,8 | 82,6 | 107,7 | 67,4 | 7,4 | 83,7 | 32,6 | 93,5 | 1415,9 | 3,1 | 35,6 | 61,3 |
РФ | RF | 10.05.2021 | 19 | 12.09.2021 | 37 | 18 | 39 | 101,6 | 119,2 | 83,6 | 7,0 | 93,5 | 47,6 | 110,3 | 1504,3 | 2,6 | 41,1 | 56,3 | |
IV | ПК | PK | 04.10.2021 | 40 | 09.01.2022 | 1 | 14 | 86,67 | 100,7 | 177,1 | 142,9 | 9,3 | 157,7 | 109,8 | 171,8 | 2001,1 | 3,0 | 36,5 | 60,5 |
РФ | RF | 13.09.2021 | 37 | 09.01.2022 | 1 | 17 | 115,7 | 95,5 | 191,8 | 141,8 | 8,3 | 145,5 | 115,7 | 172,8 | 2411,1 | 2,2 | 40,9 | 56,8 | |
V | ПК | PK | 10.01.2022 | 2 | 18.07.2022 | 29 | 27 | 104,8 | 8,7 | 581,5 | 144,2 | 35,8 | 56,9 | 11,1 | 149,6 | 3893,5 | 1,3 | 29,8 | 68,9 |
РФ | RF | 10.01.2022 | 2 | 18.07.2022 | 29 | 27 | 76,5 | 15,6 | 915,1 | 201,3 | 52,2 | 65,8 | 21,5 | 240,6 | 5435,3 | 0,4 | 33,0 | 66,6 | |
VI | ПК | PK | 19.07.2022 | 29 | 09.01.2023 | 2 | 25 | 10,8 | 20,4 | 232,2 | 69,9 | 14,0 | 30,7 | 18,3 | 107,3 | 1747,7 | 0 | 12,1 | 87,9 |
РФ | RF | 19.07.2022 | 26 | 09.01.2023 | 2 | 25 | 20,3 | 18,0 | 258 | 91,8 | 16,2 | 44,9 | 29,9 | 164,9 | 2296 | – | – | – | |
VII | ПК | PK | 10.01.2023 | 2 | 25.06.2023 | 25 | 23 | 14,3 | 3,04 | 44,4 | 20,1 | 2,5 | 16,1 | 12,7 | 28,7 | 482,2 | 0 | 25,1 | 74,9 |
РФ | RF | 10.01.2023 | 2 | 04.06.2023 | 22 | 20 | 15,2 | 8,8 | 64,9 | 36,2 | 3,9 | 34,4 | 21,3 | 54,7 | 767,1 | – | – | – | |
Примечание. РФ — Российская Федерация; «–» — нет данных.
Note. PK — Primorsky Krai; RF — Russian Federation; “–” — no data.
Рис. 2. Заболеваемость COVID-19 и генетическое разнообразие SARS-CoV-2 на территории ПК в 2020–2023 гг.
Fig. 2. Incidence of COVID-19 and genetic diversity of SARS-CoV-2 on the territory of Primorsky Krai (2020–2023).
В ПК первый случай COVID-19 был зарегистрирован 21.03.2020 (женщина 30 лет, гражданка России, проживающая во Владивостоке, вернувшаяся 15.03.2020 из служебной командировки в Мексику, г. Канкун), и на 05.05.2023 документировано 217 196 случаев этого заболевания.
В целом динамика эпидемического процесса в регионе соответствовала таковой в среднем по России (рис. 2, табл. 2), но с некоторым запаздыванием (примерно на 2 нед) по сравнению с центральными регионами страны (аналогично тому, как это имело место при распространении пандемического гриппа А (H1N1 pdm09) [18]). Первый подъём заболеваемости COVID-19 в ПК характеризовался медленным нарастанием в течение 13 нед с пиком на 29-й неделе. В России этот процесс развивался более быстрыми темпами — пик заболеваемости был зарегистрирован уже через 5 нед от начала подъёма с локальным максимумом через 19 нед. Более медленное развитие эпидемического роста в ПК было обусловлено в первую очередь введением строгих режимно-ограничительных мероприятий. В этот период в России от больных COVID-19 выделяли в основном Ухань-подобные (Wuhan) варианты возбудителя: B.1.1 (62,0%), B.1.1.317 (3,3%). В ПК большинство геновариантов вируса также относились к линиям B.1.1 (26%) и B.1.1.х (B.1.1.317 и B.1.1.277.2) — 74% (рис. 2, табл. 3).
Таблица 3. Распределение геновариантов вируса SARS-CoV-2 по периодам пандемии COVID-19 на территории ПК и России в целом в 2020–2023 гг.
Table 3. Distribution of SARS-CoV-2 genetic variants by COVID-19 pandemic periods on the territory of Primorsky Krai and a whole Russian Federation (2020–2023).
Период Period | Территория Territory | Число образцов Number of samples | Геноварианты, % | Genovariants, % | |||||||
Wuhan | Alpha | Delta | Omicron | не встречавшиеся в ПК not found in PK | ||||||
B.1.1 + B.1.1.х | B.1.1.7.x | B.1.617.2.x | BA.1.x | BA.2.x | BA.5.x | XBB.x | ||||
I | ПК | PK | 23 | 100,0 | 0,0 | 0,0 | 0,0 | 0,0 | 0,0 | 0,0 | 0,0 |
РФ | RF | 1701 | 65,3 | 0,0 | 0,0 | 0,0 | 0,0 | 0,0 | 0,0 | 34,7 | |
II | ПК | PK | 201 | 100,0 | 0,0 | 0,0 | 0,0 | 0,0 | 0,0 | 0,0 | 0,0 |
РФ | RF | 7417 | 55,0 | 7,2 | 1,6 | 0,0 | 0,0 | 0,0 | 0,0 | 36,2 | |
III | ПК | PK | 30 | 3,3 | 0,0 | 96,6 | 0,0 | 0,0 | 0,0 | 0,0 | 0,0 |
РФ | RF | 10 602 | 5,1 | 2,5 | 89,6 | 0,0 | 0,0 | 0,0 | 0,0 | 2,8 | |
IV | ПК | PK | 46 | 2,2 | 0,0 | 97,8 | 0,0 | 0,0 | 0,0 | 0,0 | 0,0 |
РФ | RF | 23 315 | 0,1 | 0,0 | 93,5 | 6,1 | 0,0 | 0,0 | 0,0 | 0,3 | |
V | ПК | PK | 368 | 0,0 | 0,0 | 33,4 | 48,9 | 17,7 | 0,0 | 0,0 | 0,0 |
РФ | RF | 24 416 | 0,1 | 0,0 | 11,4 | 38,9 | 42,5 | 6,2 | 0,0 | 0,9 | |
VI | ПК | PK | 201 | 0,0 | 0,0 | 1,0 | 0,0 | 1,0 | 98,0 | 0,0 | 0,0 |
РФ | RF | 28 300 | 0,0 | 0,0 | 0,0 | 2,1 | 11,4 | 85,7 | 0,7 | 0,1 | |
VII | ПК | PK | 130 | 0,0 | 0,0 | 0,9 | 0,0 | 2,7 | 1,1 | 96,2 | 0,0 |
РФ | RF | 4928 | 0,0 | 0,0 | 0,0 | 1,5 | 0,3 | 35,9 | 62,3 | 0,0 | |
Примечание. РФ — Российская Федерация.
Note. PK — Primorsky Krai; RF — Russian Federation.
Во время II волны рост заболеваемости как в ПК, так и в среднем по России был более медленным — 11 и 17 нед с пиками на 50-й и 52-й неделях соответственно. Доля B.1.1 в России снизилась до 33,7%, а доля B.1.1.317 и B.1.1.523 увеличилась до 21,3%; появились новые геноварианты: Alpha (B.1.1.7) — 7,2% и Delta (B.1.617.2) — 1,6 %. В ПК в этот период доминировала генетическая линия B.1.1 (68,4%); прочие геноварианты B.1.1.х (прежде всего B.1.1.317 и B.1.1.277.2) в совокупности составили 31,6%. В 2021 г. периоды подъёма заболеваемости были менее длительными, но характеризовались более интенсивным развитием эпидемического процесса. В это время в ПК были зарегистрированы III и IV подъёмы уровней заболеваемости с пиками на 27-й и 48-й неделях соответственно; в России III подъём продолжался в среднем 4 мес с максимальным уровнем заболеваемости на 29-й неделе, IV подъём — с сентября по январь 2021 г. В этот период активно шёл процесс изменения биологических свойств возбудителя: доля геноварианта Delta (B.1.617.2.x) увеличилась до 93–97%, практически вытеснив все ранее циркулировавшие линии.
Пятый подъём заболеваемости в целом по России и в ПК характеризовался резким скачком — пик заболеваемости был достигнут спустя 4 и 5 нед от начала подъёма. Показатели заболеваемости в этот период были значимо (р < 0,05) выше, чем в предыдущие периоды. Этот подъём был обусловлен совместной циркуляцией уходящего варианта Delta (B.1.617.2.х) и приходящих ему на смену первых двух линий варианта Omicron (BA.1.х и BA.2.х). К концу V периода в мире установилось доминирование варианта Omicron. Сублинии Omicron BA.5.х, доля которых увеличилась до 90%, вызвали VI подъём заболеваемости в различных регионах России. При этом и в ПК, и в России в целом течение эпидемического процесса отличалось от предыдущего периода меньшей интенсивностью и скоростью достижения пиковых показателей. Во время VII периода наблюдалось дальнейшее снижение интенсивности эпидемического процесса. Максимальное значение пика заболеваемости в среднем в России было зарегистрировано на 8-й неделе, в ПК — на 9-й неделе от начала подъёма. В этот период значительно увеличилось разнообразие субвариантов Omicron, один из них (XBB) быстро распространился по миру [33]. К концу января 2023 г. в России в целом и в ПК субварианты XBB.х преобладали, их доля в структуре субвариантов Omicron возросла до 62,3 и 92,8% соответственно.
При анализе клинических проявлений COVID-19 в ПК, как и в России в целом, преобладали заболевания в лёгкой (68,4 и 54,2% соответственно) и средней (29,3 и 42,7%) тяжести. Доля тяжёлых форм составляла 2,3 и 3,1% соответственно. Первая волна характеризовалась высокой долей пневмоний (20,9%) и бессимптомных форм (30,3%), доля тяжёлых форм составила 2,1% (табл. 2). По мере развития эпидемического процесса во II волне отмечался самый большой за весь период наблюдения удельный вес тяжёлых форм заболевания (5,8%), доля внебольничных пневмоний составляла 17,7%. На протяжении последующих периодов доля внебольничных пневмоний постепенно уменьшалась с 16,2% в III волне до 1,9% в VI волне. Удельный вес тяжёлых форм пневмоний также снизился с 3,1% до 0. Следует отметить, что удельный вес форм средней степени тяжести течения COVID-19 распределялся по периодам подъёма неравномерно: увеличивался от I к III периоду (с 31,5 до 36,5%), к VI периоду снижался до 12,1% и в VII периоде вновь увеличивался до 25,1%.
В VII эпидемическом периоде (май 2023 г.) в ПК преобладал вариант XBB.x, впервые зарегистрированный 31.01.2023 (линия XBB.1.14), к концу марта — линия XBB.1.5.24, подвид варианта XBB.1.5 (Kraken), к 05.05.2023 доминирующим стал вариант XBB.1.9.1 (Hyperion) (рис. 3). Особенно важно отметить высокую долю вариантов XBB.1.5.24 (9% в ПК и 6% по России в целом) и XBB.1.9.1 (45 и 51% соответственно), что резко контрастировало с генетическим пейзажем штаммов SARS-CoV-2 в сопредельных странах Юго-Восточной Азии (рис. 4). Такая картина, возможно, является следствием своевременного введения в России жёстких ограничительных мер, включающих прекращение транспортного сообщения со странами Азиатско-Тихоокеанского региона, в то время как относительная свобода сообщения России со странами Европы и ПК с центральной частью России сохранялась на всём протяжении пандемии.
Рис. 3. Генетическое разнообразие SARS-CoV-2 в ПК с января по май 2023 г.
Fig. 3. Genetic diversity of SARS-CoV-2 in Primorsky Krai from January to May 2023.
Рис. 4. Генетическое разнообразие SARS-CoV-2 в ПК, России в целом и сопредельных странах Восточной Азии накануне официального окончания пандемии 05.05.2023 (цифры при секторах круговых диаграмм соответствуют процентной доле данного геноварианта по данным GISAID).
Fig. 4. Genetic diversity of SARS-CoV-2 in Primorsky Krai, whole Russia, and neighboring countries of East Asia on the eve of the official end of the pandemic on May 05, 2023 (figures in the sectors of pie charts correspond to the percent portion of this genovariant according to GISAID).
Изоляция штаммов SARS-CoV-2 была успешно проведена из 151 образца ОТ-ПЦР-РВ-положительных назофарингеальных смывов (эффективность изоляции составила 151/217 ≈ 69,6%). Полученные вирусные штаммы были депонированы в коллекцию патогенных микроорганизмов НИИ эпидемиологии и микробиологии им. Г.П. Сомова. Признаки цитопатического действия вируса в культуре клеток Vero-E6 наблюдались начиная со 2–4-х суток после инфицирования (рис. 5, а, б). Распределение полученных штаммов SARS-CoV-2 по генетическим линиям представлено на рис. 5, в, а по значениям lg TCID50 — в табл. 4.
Рис. 5. Штаммы SARS-CoV-2, изолированные в чувствительной клеточной линии Vero-E6 в НИИ эпидемиологии и микробиологии им. Г.П. Сомова. а — интактная клеточная линия; б — клеточная линия с проявлениями цитопатогенного действия штамма SARS-CoV-2/Russia_Primorje/FE-R-6932 (Delta AY.122) на 5-е сутки после инфицирования (3 пассаж); в — распределение штаммов по геновариантам (цифры при секторах круговых диаграмм соответствуют процентной доле данного геноварианта).
Fig. 5. SARS-CoV-2 strains isolated in the sensitive Vero-E6 cell line at the G.P. Somov Institute of Epidemiology and Microbiology of Rospotrebnadzor a — intact cell line; b — cell line with manifestations of cytopathogenic action of the SARS-CoV-2 strain/Russia_Primorje/FE-R-6932 (Delta AY.122) for 5 days after infection (3 passage); с — the distribution of strains by genovariants (the numbers in the sectors of the pie charts correspond to the percentage of this genovariant).
Таблица 4. Инфекционные титры штаммов SARS-CoV-2, изолированных в НИИ эпидемиологии и микробиологии им. Г.П. Сомова (2020–2023 гг.)
Table 4. Infectious titers of SARS-CoV-2 strains isolated at the G.P. Somov Institute of Epidemiology and Microbiology (2020–2023)
Геновариант штамма Genovariant of the strain | Количество штаммов Number of strains | Распределение штаммов по значениям десятичного логарифма инфекционного титра, lg TCID50 Distribution of strains according to the values of the decimal logarithm of the infectious titer, lg IT | M ± m | |||||
3 | 4 | 5 | 6 | |||||
Wuhan | B.1.1 | 49 | 2 | 8 | 11 | 28 | 5,3 ± 0,1 | 5,3 ± 0,1 |
B.1.1.1 | 2 | 0 | 0 | 1 | 1 | 5,5 ± 0,5 | ||
B.1.1.317 | 4 | 0 | 1 | 2 | 1 | 5,0 ± 0,4 | ||
B.1.1.397 | 5 | 0 | 0 | 2 | 3 | 5,6 ± 0,2 | ||
B.1.1.485 | 1 | 0 | 0 | 1 | 0 | 5,0 ± 0,0 | ||
B.1.143 | 1 | 0 | 1 | 0 | 0 | 4,0 ± 0,0 | ||
Delta | AY.121 | 1 | 0 | 0 | 1 | 0 | 5,0 ± 0,0 | 4,3 ± 0,2 |
AY.122 | 36 | 8 | 14 | 9 | 5 | 4,3 ± 0,2 | ||
Omicron | BA.1.x | 20 | 10 | 4 | 6 | 0 | 3,8 ± 0,2 | 3,7 ± 0,1 |
BA.2 | 5 | 2 | 3 | 0 | 0 | 3,6 ± 0,2 | ||
BA.5.2 | 5 | 1 | 3 | 1 | 0 | 4,0 ± 0,3 | ||
XBB.x | 22 | 13 | 8 | 1 | 0 | 3,5 ± 0,1 | ||
Плоскость главных компонент для различных периодов пандемии COVID-19 в ПК и России, рассматриваемых как точки в восьмимерном пространстве признаков — компонент распределения SARS-CoV-2 по геновариантам (табл. 3) — представлена на рис. 6. Две первые главные компоненты на рис. 6, соответствующие двум наибольшим собственным значениям ковариационной матрицы признаков, включают 63,6% информации о взаимном расположении объектов в многомерном признаковом пространстве. Известно, что вращение главных компонент на один и тот же угол не изменяет значения собственных чисел ковариационной матрицы [32]: на рис. 6 вращение компонент осуществлено таким образом, чтобы максимизировать дисперсию факторных нагрузок.
Рис. 6. Плоскость двух первых главных компонент для различных эпидемических периодов COVID-19, обозначаемых латинскими цифрами (так же, как на рис. 2 и в табл. 2, 3): серые точки соответствуют ПК, белые — России.
Fig. 6. The plane of the first two main components for various epidemic periods of COVID-19 indicated by Latin numerals (as in Figure 2 and Tables 2–3): the gray dots correspond to the Primorsky Krai, the white ones — to the Russian Federation.
Обсуждение
В России пандемия COVID-19 началась позже, чем во многих странах, вследствие применения строгих режимно-ограничительных и противоэпидемических мероприятий в отношении COVID-19, которые были регламентированы Указами Президента РФ7, Распоряжениями Правительства РФ8 и Постановлениями Главного государственного санитарного врача РФ9. Мероприятия по предупреждению завоза и снижению рисков распространения COVID-19 на территории ПК вводились на основании как вышеперечисленных указов и распоряжений, так и Постановления губернатора ПК10 и главного государственного санитарного врача по ПК11. Все проведённые на начальном этапе мероприятия позволили замедлить темпы распространения COVID-19, увеличить объём исследований на COVID-19 и мощность лабораторных баз, подготовить медицинские организации для оказания помощи больным, что предотвратило увеличение числа случаев тяжёлых форм заболевания и летальности.
Анализ проявлений эпидемического процесса COVID-19 в период с апреля 2020 г. по июнь 2023 г. на территории ПК и России в целом выявил 7 подъёмов заболеваемости, различавшихся по клинико-эпидемиологическим проявлениям течения COVID-19 в зависимости от геноварианта возбудителя (рис. 1, 2, табл. 2, 3). Сравнение заболеваемости COVID19 в каждом периоде показало, что динамика развития эпидемического процесса в ПК аналогична общероссийской, что особенно наглядно демонстрирует рис. 6. При этом в структуре клинических форм заболевания в течение всей пандемии преобладали лёгкие и средней степени тяжести клинические формы.
Введение ограничительных мер в марте–апреле 2020 г. практически прекратило перемещение людей между ПК и сопредельными государствами (Китаем, Республикой Кореей, Японией), в результате чего динамика циркуляции геновариантов вируса SARS-CoV-2 в регионе объяснялась исключительно внутрироссийской миграцией, поэтому во время I и II периодов пандемии в ПК (как и в России в целом) доминирующим был геновариант Wuhan, в III и IV периодах — Delta, в V и в последующих подъёмах — различные геноварианты Omicron (рис. 2). Смена доминирующих геновариантов проиллюстрирована на рис. 3.
С середины 2022 г. началось постепенное смягчение режима перемещения с сопредельными странами, что не могло не сказаться на разнообразии геновариантов Omicron, циркулирующих в ПК. Так, в ПК к концу апреля – началу мая 2023 г. доля вариантов XBB.1.9.2 и XBB.1.16 (Arcturus) была несколько выше, чем в России в целом. В то же время доля варианта XBB.1.9.2 была наиболее велика в Республике Корея, а варианта XBB.1.16 — в Японии (рис. 4). Эти данные позволяют предполагать определённое участие сопредельных стран Азиатско-Тихоокеанского региона в формировании генетического ландшафта SARS-CoV-2 в ПК на заключительном этапе пандемии. Данные филогенетического анализа образцов редкого в России субварианта BN (рис. 5) также могут служить косвенным подтверждением множественности путей проникновения вируса в регион. При этом в ПК доля зарегистрированных случаев COVID-19, этиологическим фактором которых являлись XBB.1.9.2 (5%) и XBB.1.16 (3%), была сопоставима с таковыми в Республике Корея (XBB.1.9.2 — 7%), и в Японии (XBB.1.16 — 12%; рис. 4), в отличие от среднероссийских значений (XBB.1.9.2 — 2%, XBB.1.16 — 1%) [34–36]. Эти данные позволяют предполагать возможное участие геновариантов возбудителя, циркулирующих в сопредельных странах Азиатско-Тихоокеанского региона, в формировании генетического ландшафта SARS-CoV-2 в ПК на заключительном этапе пандемии.
Филогенетический анализ изолятов, относящихся к линии BN, слабо представленной в России (около 150 образцов в GISAID), но характерной для Республики Корея [37], подтверждает возможность завоза новых вариантов вируса в ПК из этой страны. По данным сайта Pango, 37% изолятов линии BN.1.2, 41% линии BN.1.2.3 и 25% линии BN.1.3 происходят из РК. Филогенетический анализ наиболее близких по структуре образцов SARS-CoV-2 из России и Республики Корея показал, что образец prim000233 (BN.1.2, дата забора 14.04.2023) относится, скорее, к группе южнокорейских изолятов, в то время как образец prim000314 (BN.1.2.3, дата забора 10.04.2023) — к группе изолятов, выделенных из центральной части России (рис. 7; низкие значения bootstrap-поддержки для некоторых кластеров на этом рисунке объясняются высокой степенью гомологии анализируемых последовательностей, что характерно для большинства региональных исследований SARS-CoV-2 [38–42]). Данные результаты могут также служить косвенным подтверждением множественности путей проникновения вируса в регион.
Рис. 7. Филогенетическое древо наиболее гомологичных друг другу вирусов SARS-CoV-2 линии BN. Для образцов из России указаны номера в VGARus и номера в GISAID (в скобках). Для образцов из Республики Корея указаны номера в GISAID. Номера образцов из ПК выделены жирным шрифтом.
Fig. 7. Phylogenetic tree of the most homologous viruses of the BN line of SARS-CoV-2. Numbers in VGARus and GISAID (in parentheses) databases are given for the samples from Russia. Numbers in GISAID database are given for the samples from Republic of Korea. Numbers of samples from Primorsky Krai are given in bold.
В процессе изоляции штаммов SARS-CoV-2 отмечено значимое снижение титров геновариантов Delta и Omicron, что свидетельствует об уменьшении их вирулентности (скорости репликации и накопления вируса) по сравнению с Wuhan-подобными вариантами (табл. 4). Полученные результаты согласуются с эпидемиологическими проявлениями инфекционного процесса — повышением контагиозности вариантов SARS-CoV-2 и снижением тяжести клинических проявлений инфекции.
Распределение генотипов SARS-CoV-2, выявленных в клиническом материале (рис. 1, б, в), свидетельствует о преобладании варианта Omicron (59,7%), в то время как среди изолированных штаммов доминируют Wuhan-подобные варианты (41,0%), что объясняется их более высокой вирулентностью. Это подтверждает правильность выбора стратегии оперативного эпидемиологического мониторинга, основанного на секвенировании полноразмерного вирусного генома именно из первичного клинического материала (хотя это не исключает необходимость изоляции различных вариантов возбудителя для проведения вирусологических исследований).
Таким образом, уникальность формирования генотипического портрета SARS-CoV-2 на территории ПК в период пандемии COVID-19 заключается в возможности завоза вируса как из европейской части России, так и из стран Восточной Азии. Последнее направление представляется наиболее актуальным в связи со стратегическим «восточным вектором» внешней политики, который последовательно реализует Правительство РФ. Сохраняющаяся возможность возникновения в сопредельных государствах новых, особо опасных вариантов вируса делает молекулярно-генетический мониторинг SARS-CoV-2 на территории ПК особенно актуальным. Ретроспективный анализ эпидемического процесса COVID19 на конкретной территории является необходимым условием для формирования моделей и прогнозов развития ситуации, позволяет идентифицировать факторы риска, связанные с распространением и тяжестью заболевания, оценить эффективность принятых мер, повысить надёжность обеспечения биологической безопасности.
1 Отчётная форма Роспотребнадзора № 970 «Информация о случаях заболевания лиц с подозрением на коронавирусную инфекцию», отчёт № 1076 «Сводный отчет о случаях COVID-19 в регионах».
2 URL: https://www.rospotrebnadzor.ru/region/korono_virus/epid.php
3 URL: https://стопкоронавирус.рф/
4 URL: https://gisaid.org
5 Количество ОТ-ПЦР-РВ-положительных проб не следует ассоциировать с количеством пациентов с COVID-19: в 2020 г. больные обследовались в среднем 3,6 раза, т.к. в то время из лечебного учреждения выписывали после получения двукратного отрицательного результата, который достигался не с первого раза; в 2021 г. выписка стала производиться при получении однократного отрицательного результата, а позже — по факту выздоровления.
6 Приказ Роспотребнадзора от 19.02.2021 № 56 «О совершенствовании молекулярно-генетического мониторинга штаммов возбудителя новой коронавирусной инфекции».
7 Указы Президента: от 25.09.2020 № 206; от 02.04.2020 № 239; от 02.04.2020 № 294.
8 Распоряжения Правительства РФ: от 30.01.2020 № 140-р; от 31.01.2020 № 154-р; от 03.02.2020 № 194-р; от 18.02.2020 № 338-р; от 27.02.2020 № 447-р; от 27.02.2020 № 446-р; от 27.02.2020 № 448-р; от 16.03.2020 № 635-р; от 06.03.2020 № 550-р; от 12.03.2020 № 597-р; от 14.03.2020 № 622-р; от 16 марта 2020 № 730-р; от 27.03.2020 № 763-р.
9 Постановления Главного государственного санитарного врача РФ: от 24.01.2020 № 2; от 31.01.2020 № 3; от 02.03.2020 № 5; от 13.03.2020 № 6; от 18.03.2020 № 7; от 30.03.2020 № 9.
10 Постановление губернатора ПК от 18.03.2020 № 21-пг.
11 Постановление Главного государственного санитарного врача ПК от 20.04.2020 № 17.
Об авторах
Анна Юрьевна Попова
Федеральная служба по надзору в сфере защиты прав потребителей и благополучия человека
Email: uglevas@bk.ru
ORCID iD: 0000-0003-2567-9032
д.м.н., профессор, руководитель Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека
Россия, МоскваМихаил Юрьевич Щелканов
НИИ эпидемиологии и микробиологии им. Г.П. Сомова; Дальневосточный федеральный университет
Автор, ответственный за переписку.
Email: adorob@mail.ru
ORCID iD: 0000-0001-8610-7623
д.б.н., директор НИИ эпидемиологии и микробиологии им. Г.П. Сомова; зав. каф. эпидемиологии, микробиологии и паразитологии с Международным научно-образовательным Центром биологической безопасности Роспотребнадзора Школы наук о жизни и биомедицины Дальневосточного федерального университета
Россия, Владивосток; ВладивостокНаталья Владимировна Крылова
НИИ эпидемиологии и микробиологии им. Г.П. Сомова; Дальневосточный федеральный университет
Email: krylovanatalya@gmail.com
ORCID iD: 0000-0002-9048-6803
д.б.н., в.н.с., зав. лаб. респираторных инфекций НИИ эпидемиологии и микробиологии им. Г.П. Сомова; доцент каф. эпидемиологии, микробиологии и паразитологии с Международным научно-образовательным Центром биологической безопасности Роспотребнадзора Школы наук о жизни и биомедицины Дальневосточного федерального университет
Россия, Владивосток; ВладивостокАлексей Анатольевич Белик
НИИ эпидемиологии и микробиологии им. Г.П. Сомова
Email: belik_a_a@mail.ru
ORCID iD: 0000-0002-0303-3188
к.б.н., н.с. лаб. респираторных инфекций НИИ эпидемиологии и микробиологии им. Г.П. Сомова
Россия, ВладивостокЛюбовь Михайловна Семейкина
НИИ эпидемиологии и микробиологии им. Г.П. Сомова; Центр гигиены и эпидемиологии в Приморском крае
Email: lms78@mail.ru
ORCID iD: 0009-0002-1095-7968
аспирант НИИ эпидемиологии и микробиологии им. Г.П. Сомова; зам. зав. отделом эпидемиологии Центра гигиены и эпидемиологии в Приморском крае
Россия, Владивосток; ВладивостокТатьяна Станиславовна Запорожец
НИИ эпидемиологии и микробиологии им. Г.П. Сомова
Email: niiem_vl@mail.ru
ORCID iD: 0000-0002-8879-8496
д.м.н., г.н.с. лаб. респираторных вирусных инфекций, зам. директора по научной работе НИИ эпидемиологии и микробиологии им. Г.П. Сомова
Россия, ВладивостокВячеслав Юрьевич Смоленский
Федеральная служба по надзору в сфере защиты прав потребителей и благополучия человека
Email: aidscouncil@gsen.ru
ORCID iD: 0000-0003-1773-4097
к.м.н., зам. руководителя Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека
Россия, МоскваЕлена Викторовна Персиянова
НИИ эпидемиологии и микробиологии им. Г.П. Сомова
Email: helen-pers@yandex.ru
ORCID iD: 0000-0002-5686-8672
к.б.н., с.н.с. лаб. респираторных вирусных инфекций НИИ эпидемиологии и микробиологии им. Г.П. Сомова
Россия, ВладивостокМарина Николаевна Просянникова
Центр гигиены и эпидемиологии в Приморском крае
Email: marpros67@mail.ru
ORCID iD: 0009-0002-5265-8106
зав. лаб. вирусных и особо опасных бактериальных инфекций Центра гигиены и эпидемиологии в Приморском крае
Россия, ВладивостокЮрий Александрович Белов
НИИ эпидемиологии и микробиологии им. Г.П. Сомова; Дальневосточный федеральный университет
Email: bornley@yandex.ru
ORCID iD: 0000-0001-8313-5610
м.н.с., зав. Центром молекулярной диагностики НИИ эпидемиологии и микробиологии им. Г.П. Сомова; ассистент каф. эпидемиологии, микробиологии и паразитологии с Международным научно-образовательным Центром биологической безопасности Роспотребнадзора Школы наук о жизни и биомедицины Дальневосточного федерального университета
Россия, Владивосток; ВладивостокОльга Викторовна Иунихина
НИИ эпидемиологии и микробиологии им. Г.П. Сомова; Дальневосточный федеральный университет
Email: olga-iun@inbox.ru
ORCID iD: 0000-0002-6723-582X
к.м.н., зав. лаб. зоонозных инфекций НИИ эпидемиологии и микробиологии им. Г.П. Сомова; доцент каф. эпидемиологии, микробиологии и паразитологии с Международным научно-образовательным Центром биологической безопасности Роспотребнадзора Школы наук о жизни и биомедицины Дальневосточного федерального университета
Россия, Владивосток; ВладивостокАнастасия Борисовна Потт
НИИ эпидемиологии и микробиологии им. Г.П. Сомова
Email: adorob@mail.ru
ORCID iD: 0000-0002-1235-4960
н.с. лаб. респираторных вирусных инфекций НИИ эпидемиологии и микробиологии им. Г.П. Сомова
Россия, ВладивостокТатьяна Фатеевна Хомичук
Центр гигиены и эпидемиологии в Приморском крае
Email: adorob@mail.ru
ORCID iD: 0009-0007-3941-228X
зав. отделом эпидемиологии Центра гигиены и эпидемиологии в Приморском крае
Россия, ВладивостокАнна Ивановна Симакова
Тихоокеанский государственный медицинский университет
Email: adorob@mail.ru
ORCID iD: 0000-0002-3334-4673
д.м.н., зав. каф. инфекционных болезней Тихоокеанского государственного медицинского университета
Россия, ВладивостокСветлана Алексеевна Абрамова
НИИ эпидемиологии и микробиологии им. Г.П. Сомова
Email: adorob@mail.ru
ORCID iD: 0000-0002-2428-3186
м.н.с. лаб. патоморфологии НИИ эпидемиологии и микробиологии им. Г.П. Сомова
Россия, ВладивостокОльга Борисовна Романова
Центр гигиены и эпидемиологии в Приморском крае
Email: adorob@mail.ru
главный врач Центра гигиены и эпидемиологии в Приморском крае
Россия, ВладивостокТатьяна Николаевна Детковская
Управление Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека в Приморском крае
Email: adorob@mail.ru
ORCID iD: 0000-0002-7543-0633
руководитель Управления Роспотребнадзора по Приморскому краю
Россия, ВладивостокСергей Петрович Крыжановский
Медицинское объединение Дальневосточного отделения Российской академии наук
Email: adorob@mail.ru
ORCID iD: 0000-0002-1981-1079
д.м.н., профессор, член-корреспондент РАН, научный руководитель Медицинского объединения Дальневосточного отделения Российской академии наук
Россия, ВладивостокНаталия Николаевна Беседнова
НИИ эпидемиологии и микробиологии им. Г.П. Сомова; Медицинское объединение Дальневосточного отделения Российской академии наук
Email: adorob@mail.ru
ORCID iD: 0000-0002-2760-9778
D. Sci. (Med.), Professor, Full Member of the Russian Academy of Sciences, main researcher, Laboratory of immunobiological preparations, G.Р. Somov Institute of Epidemiology and Microbiology; Chief Researcher, Research Department, Medical Association of the Far Eastern Branch of the Russian Academy of Sciences
Россия, Владивосток; ВладивостокСписок литературы
- Щелканов М.Ю., Попова А.Ю., Дедков В.Г. и др. История изучения и современная классификация коронавирусов (Nidovirales: Coronaviridae). Инфекция и иммунитет. 2020;10(2):221–46. Shchelkanov M.Yu., Popova A.Yu., Dedkov V.G., et al. History of investigation and current classification of coronaviruses (Nidovirales: Coronaviridae). Russian Journal of Infection and Immunity. 2020;10(2):221–46. DOI: https://doi.org/10.15789/2220-7619-HOI-1412 EDN: https://elibrary.ru/kziwrq
- Никифоров В.В., Колобухина Л.В., Сметанина С.В. и др. Новая коронавирусная инфекция (COVID-19): этиология, эпидемиология, клиника, диагностика, лечение и профилактика. М.;2020. Nikiforov V.V., Kolobukhina L.V., Smetanina S.V., et al. Novel Coronavirus Infection (COVID-19): Etiology, Epidemiology, Clinics, Diagnostics, Treatment, and Prophylaxis. Moscow;2020. EDN: https://elibrary.ru/hgqiyk
- Щелканов М.Ю. Этиология COVID-19. В кн.: COVID-19: от этиологии до вакцинопрофилактики. Руководство для врачей. М.;2023:11–53. Shchelkanov M.Yu. Etiology of COVID-19. In: COVID-19: from Etiology to Vaccine Prevention. Guide for Doctors. Moscow;2023:11–53. DOI: https://doi.org/10.33029/9704-7967-4-COV-2023-1-288 EDN: https://elibrary.ru/gegddl
- Шестопалов А.М., Кононова Ю.В., Гаджиев А.А. и др. Биоразнообразие и эпидемический потенциал коронавирусов (Nidovirales: Coronaviridae) рукокрылых. Юг России: экология, развитие. 2020;15(2):17–34. Shestopalov A.M., Kononova Yu.V., Gadzhiev A.A., et al. Biodiversity and epidemic potential of chiropteran coronaviruses (Nidovirales: Coronaviridae). South of Russia: Ecology, Development. 2020;15(2):17–34 DOI: https://doi.org/10.18470/1992-1098-2020-2-17-34 EDN: https://elibrary.ru/csbxlk
- Щелканов М.Ю., Колобухина Л.В., Бургасова О.А. и др. COVID-19: этиология, клиника, лечение. Инфекция и иммунитет. 2020;10(3):421–45. Shchelkanov M.Yu., Kolobukhina L.V., Burgasova O.A., et al. COVID-19: etiology, clinic, treatment. Russian Journal of Infection and Immunity. 2020;10(3):421–45. DOI: https://doi.org/10.15789/2220-7619-CEC-1473 EDN: https://elibrary.ru/imaadb
- Щелканов М.Ю., Дунаева М.Н., Москвина Т.В. и др. Каталог вирусов рукокрылых (2020). Юг России: экология, развитие. 2020;15(3):6–30. Shchelkanov M.Yu., Dunaeva M.N., Moskvina N.V., et al. Catalog of bat viruses (2020). South of Russia: Ecology, Development. 2020;15(3):6–30. DOI: https://doi.org/10.18470/1992-1098-2020-3-6-30 EDN: https://elibrary.ru/tzqsap
- Щелканов Е.М., Уколов С.С., Дунаева М.Н. и др. Эхолокация рукокрылых (Chiroptera Blumenbach, 1779) как элемент их экологической пластичности. Юг России: экология, развитие. 2020;15(4):6–20. Shchelkanov E.M., Ukolov S.S., Dunaeva M.N., et al. Echolocation of bats (Chiroptera Blumenbach, 1779) as the element of their ecological plasticity. South of Russia: Ecology, Development. 2020;15(4):6–20. DOI: https://doi.org/10.18470/1992-1098-2020-4-6-20 EDN: https://elibrary.ru/mubjcm
- Щелканов М.Ю., Табакаева Т.В., Щелканов Е.М. и др. Паукообразные-эктопаразиты рукокрылых. Владивосток;2022. Shchelkanov M.Yu., Tabakaeva T.V., Shchelkanov E.M., et al. Arachnids-Ectoparasites of Chiropterans. Vladivostok;2022. DOI: https://doi.org/10.24866/7444-5377-0 EDN: https://elibrary.ru/ulhbco
- Щелканов М.Ю., Табакаева Т.В., Щелканов Е.М. и др. Насекомые-эктопаразиты рукокрылых. Владивосток;2022. Shchelkanov M.Yu., Tabakaeva T.V., Shchelkanov E.M., et al. Insects-Ectoparasites of Chiropterans. Vladivostok;2022. DOI: https://doi.org/10.24866/7444-5404-3 EDN: https://elibrary.ru/wfkcuc
- Щелканов М.Ю., Колобухина Л.В., Львов Д.К. Коронавирусы человека (Nidovirales, Coronaviridae): возросший уровень эпидемической опасности. Лечащий врач. 2013;(10):49–54. Shchelkanov M.Yu., Kolobukhina L.V., Lvov D.K. Human coronaviruses (Nidovirales, Coronaviridae): increased level of epidemic threat. The Practitioner. 2013;(10):49–54. EDN: https://elibrary.ru/takhvr
- Щелканов М.Ю., Ананьев В.Ю., Кузнецов В.В., Шуматов В.Б. Ближневосточный респираторный синдром: когда вспыхнет тлеющий очаг? Тихоокеанский медицинский журнал. 2015;(2):94–8. Shchelkanov M.Yu., Ananiev V.Yu., Kuznetsov V.V., Shumatov V.B. Middle East respiratory syndrome: when will smouldering focus outbreak? Pacific Medical Journal. 2015;(2):94–8. EDN: https://elibrary.ru/ulfnff
- Щелканов М.Ю., Ананьев В.Ю., Кузнецов В.В., Шуматов В.Б. Эпидемическая вспышка Ближневосточного респираторного синдрома в Республике Корея (май–июль 2015 г.): причины, динамика, выводы. Тихоокеанский медицинский журнал. 2015;(3):89–93. Shchelkanov M.Yu., Ananiev V.Yu., Kuznetsov V.V., Shumatov V.B. Epidemic outbreak of MERS in the Republic of Korea (May–July, 20015): reasons, dynamics, conclusions. Pacific Medical Journal. 2015;(3):89–93. EDN: https://elibrary.ru/ulhaer
- Kiselev D., Matsvay A., Abramov I., et al. Current trends in diagnostics of viral infections of unknown etiology. Viruses. 2020;12(2):211. DOI: https://doi.org/10.3390/v12020211 EDN: https://elibrary.ru/tvffro
- Акимкин В.Г., Семененко Т.А., Углева С.В. и др. COVID-19 в России: эпидемиология и молекулярно-генетический мониторинг. Вестник Российской академии медицинских наук. 2022;77(4):254–60. Akimkin V.G., Semeneko T.A., Ugleva S.V., et al. COVID-19 in Russia: epidemiology and molecular genetic monitoring. Annals of the Russian Academy of Medical Sciences. 2022;77(4):254–60. DOI: https://doi.org/10.15690/vramn2121 EDN: https://elibrary.ru/dozijs
- Грибова В.В., Окунь Д.Б., Шалфеева Е.А. и др. Облачный сервис для дифференциальной клинической диагностики острых респираторных вирусных заболеваний (в том числе – связанных с особо опасными коронавирусами) методами искусственного интеллекта. Якутский медицинский журнал. 2020;(2):44–7. Gribova V.V., Okun D.B., Shalfeeva E.A., et al. Cloud service for the differential clinical diagnostics of acute respiratory viral diseases (including those associated with highly contagious coronaviruses) with an application of methods of artificial intelligence. Yakut Medical Journal. 2020;(2):44–7. DOI: https://doi.org/10.25789/YMJ.2020.70.13 EDN: https://elibrary.ru/etwrev
- Латыпова М.Ф., Цибин А.Н., Комаров А.Г. и др. Организация геномного надзора за SARS-CoV-2 в структуре департамента здравоохранения города Москвы. Проблемы социальной гигиены, здравоохранения и истории медицины. 2022;30(S):1061–6. Latypova M.F., Tsibin A.N., Komarov A.G., et al. Organization of genomic surveillance for SARS-CoV-2 within the Moscow city health department. Problems of Social Hygiene, Public Health and History of Medicine, Russian Journal. 2022;30(S):1061–6. DOI: https://doi.org/10.32687/0869-866X-2022-30-s1-1061-1066 EDN: https://elibrary.ru/mshhnn
- Акимкин В.Г., Попова А.Ю., Хафизов К.Ф. и др. COVID-19: эволюция пандемии в России. Сообщение II: Динамика циркуляции геновариантов вируса SARS-CoV-2. Журнал микробиологии, эпидемиологии и иммунобиологии. 2022;99(4):381–96. Akimkin V.G., Popova A.Yu., Khafizov K.F., et al. COVID-19: Evolution of the pandemic in Russia. Report II: Dynamics of the circulation of SARS-CoV-2 genetic variants. Journal of Microbiology, Epidemiology and Immunobiology. 2022;99(4):381–96. DOI: https://doi.org/10.36233/0372-9311-295 EDN: https://elibrary.ru/kvulas
- Щелканов М.Ю., Львов Д.Н., Федякина И.Т. и др. Динамика распространения пандемического гриппа А/H1N1 swl на Дальнем Востоке в 2009 г. Вопросы вирусологии. 2010;55(3):10–5. Shchelkanov M.Yu., Lvov D.N., Fedyakina I.T., et al. Trends in the spread of pandemic influenza A(H1N1) swl in the Far East in 2009. Problems in Virology. 2010;55(3):10–5. EDN: https://elibrary.ru/muekiz
- Акимкин В.Г., Попова А.Ю., Плоскирева А.А. и др. COVID-19: эволюция пандемии в России. Сообщение I: Проявления эпидемического процесса COVID-19. Журнал микробиологии, эпидемиологии и иммунобиологии. 2022;99(4):381–96. Akimkin V.G., Popova A.Yu., Ploskireva A.A., et al. COVID-19: Evolution of the pandemic in Russia. Report I: Manifestation of the COVID-19 epidemic process. Journal of Microbiology, Epidemiology and Immunobiology. 2022;99(4):381–96. DOI: https://doi.org/10.36233/0372-9311-276 EDN: https://elibrary.ru/zxgtfd
- Shcheglov B.O., Galkina I.V., Lembikov A.O., et al. Rhinolaryngological simulators based on 3D printing open up new opportunities for professional training. Yakut Medical Journal. 2020;(3):58–60. DOI: https://doi.org/10.25789/YMJ.2020.71.16 EDN: https://elibrary.ru/sxnomh
- Щеглов Б.О., Дунаева М.Н., Баранчугов И.А. и др. Макет дыхательных путей человека. Патент RF 2740727 C1; 2021. Shcheglov B.O., Dunaeva M.N., Baranchugov I.A., et al. Model of human respiratory tract. Patent RF 2740727 C1; 2021. EDN: https://elibrary.ru/hmlszk
- Gonzalez-Recio O., Gutierrez-Rivas M., Peiro-Pastor R., et al. Sequencing of SARS-CoV-2 genome using different nanopore chemistries. Appl. Microbiol. Biotechnol. 2021;105(8):3225–34. DOI: https://doi.org/10.1007/s00253-021-11250-w
- Brejova B., Borsova K., Hodorova V., et al. Nanopore sequencing of SARS-CoV-2: Comparison of short and long PCR-tiling amplicon protocols. PLoS One. 2021;16(10):e0259277. DOI: https://doi.org/10.1371/journal.pone.0259277
- Felsenstein J. Inferring Phylogenies. Sunderland, Massachusetts;2003.
- Крылова Н.В., Иунихина О.В., Федореев С.А. и др. Анти-SARS-CoV-2 активность полифенольного комплекса из Maackia amurensis. Бюллетень экспериментальной биологии и медицины. 2023;176(8):216–9. Krylova N.V., Iunikhina O.V., Fedoreev S.A., et al. Anti-SARS-CoV-2 activity of the polyphenol complex from Maackia amurensis. Bulletin of Experimental Biology and Medicine. 2023;176(8):216–9. DOI: https://doi.org/10.47056/0365-9615-2023-176-8-216-219 EDN: https://elibrary.ru/ivgjae
- Krylova N.V., Kravchenko A.O., Iunikhina O.V., et al. Influence of the structural features of carrageenans from red algae of the Far Eastern seas on their antiviral properties. Marine Drugs. 2022;20(1):60. DOI: https://doi.org/10.3390/md20010060 EDN: https://elibrary.ru/yuukig
- Щелканов М.Ю., Сахурия И.Б., Полякова Е.Б. и др. Повышение качества МТТ-метода с помощью микродозаторных наконечников специальной конструкции. Иммунология. 1998;(4):57–9. Shchelkanov M.Yu., Sakhuria I.B., Polyakova E.B., et al. Improvement of the MTT-based assay by modification of pipette tips. Immunology (Moscow). 1998;(4):57–9. EDN: https://elibrary.ru/mpawbj
- Щелканов М.Ю., Ерёмин В.Ф., Сахурия И.Б. и др. Дегидрогеназная активность инфицированных клеток и биологические свойства различных вариантов ВИЧ-1. Биохимия. 1999;64(4):513–9. Shchelkanov M.Yu., Eremin V.F., Sakhuria I.B., et al. Dehydrogenase activity of infected cells and biological properties of HIV-1 variants. Biochemistry (Moscow). 1999;64(4):431–6. EDN: https://elibrary.ru/lfiulj
- Бейли Н. Математика в биологии и медицине. Пер. с англ. М.;1970. Bailey N. The Mathematical Approach to Biology and Medicine. London-NY-Sydney;1967.
- Урбах В.Ю. Статистический анализ в биологических и медицинских исследованиях. М.;1975. Urbach V.Yu. Statistical Analysis in Biological and Medical Research. Moscow;1975.
- Щелканов М.Ю., Пашкова Т.А., Сахурия И.Б. и др. Анализ биологических характеристик первичных изолятов ВИЧ-1 с помощью метода главных компонент. Вопросы вирусологии. 1998;43(3):117–21. Shchelkanov M.Yu., Pashkova T.A., Sakhuria I.B., et al. Analysis of biological characteristics of primary HIV-1 isolates using the main components method. Problems in Virology. 1998;43(3):117–21. EDN: https://elibrary.ru/mqdzqr
- Щелканов М.Ю., Юдин А.Н., Бурунова В.В. и др. Применение метода главных компонент для анализа эффективности панелей эпитоп-имитирующих пептидов при серотипировании ВИЧ. Иммунология. 1999;20(3):13–8. Shchelka- nov M.Yu., Yudin A.N., Burunova V.V., et al. Application of the basic components method for analysis of effectiveness of epitope-imitating peptides panels in HIV serotyping. Immunology (Moscow). 1999;20(3):13–8. EDN: https://elibrary.ru/yrqiqn
- Tamura T., Ito J., Uriu K., et al. Virological characteristics of the SARS-CoV-2 XBB variant derived from recombination of two Omicron subvariants. Nat. Commun. 2023;(14):2800. DOI: https://doi.org/10.1038/s41467-023-38435-3
- Yamasoba D., Uriu K., Plianchaisuk A., et al. Virological characteristics of the SARS-CoV-2 omicron XBB.1.16 variant. Lancet Infect. Dis. 2023;23(6):655–6. DOI: https://doi.org/10.1016/s1473-3099(23)00278-5
- WHO. XBB.1.16 Updated Risk Assessment; 05 June 2023. Available at: https://www.who.int/docs/default-source/coronaviruse/05062023xbb.1.16.pdf?sfvrsn=f1845468_3
- Lee D.W., Kim J.M., Park A.K., et al. Genomic epidemiology of SARS-CoV-2 Omicron variants in the Republic of Korea. Sci. Rep. 2022;12(1):22414. DOI: https://doi.org/10.1038/s41598-022-26803-w
- Kim I.H., No J.S., Kim J.A., et al. Genomic epidemiology of SARS-CoV-2 variants in South Korea between January 2020 and February 2023. Virology. 2023;587:109869. DOI: https://doi.org/10.1016/j.virol.2023.109869
- Браверман Э.М., Мучник И.Б. Структурные методы обработки эмпирических данных. М.;1983. Braverman E.M., Muchnik I.B. Structural Methods of Empirical Data Processing. Moscow;1983.
- Basheer A., Zahoor I. Genomic epidemiology of SARS-CoV-2 divulge B.1, B.1.36, and B.1.1.7 as the most dominant lineages in first, second, and third wave of SARS-CoV-2 infections in Pakistan. Microorganisms. 2021;9(12):2609. DOI: https://doi.org/10.3390/microorganisms9122609
- Goletic T., Konjhodzic R., Fejzic N., et al. Phylogenetic pattern of SARS-CoV-2 from COVID-19 patients from Bosnia and Herzegovina: lessons learned to optimize future molecular and epidemiological approaches. Bosn. J. Basic Med. Sci. 2021;21(4):484–7. DOI: https://doi.org/10.17305/bjbms.2020.5381
- Menasria T., Aguilera M. Genomic diversity of SARS-CoV-2 in Algeria and North African Countries: What we know so far and what we expect? Microorganisms. 2022;10(2):467. DOI: https://doi.org/10.3390/microorganisms10020467
- Градобоева Е.А., Тюлько Ж.С., Фадеев А.В. и др. Сравнительный анализ разнообразия линий SARS-CoV-2, циркулирующих в Омской области в 2020–2022 годах. Эпидемиология и вакцинопрофилактика. 2022;21(6):24–33. Gradoboeva E.A., Tyulko J.S., Fadeev A.V., et al. Comparative analysis of the diversity of SARS-CoV-2 lines circulating in the Omsk region in 2020-2022. Epidemiology and Vaccinal Prevention. 2022;21(6):24–33. DOI: https://doi.org/10.31631/2073-3046-2022-6-24-33
Дополнительные файлы