Генетическая характеристика вирусов гриппа A и B, циркулировавших в России в 2019–2023 годах
- Авторы: Яцышина С.Б.1, Артамонова А.А.1, Елькина М.А.1, Валдохина А.В.1, Буланенко В.П.1, Берсенева А.А.1, Акимкин В.Г.1
-
Учреждения:
- Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
- Выпуск: Том 101, № 6 (2024)
- Страницы: 719-734
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://microbiol.crie.ru/jour/article/view/18510
- DOI: https://doi.org/10.36233/0372-9311-480
- EDN: https://elibrary.ru/knbqnk
- ID: 18510
Цитировать
Аннотация
Актуальность. Вирусы гриппа обладают высоким потенциалом генетических изменений. Ежегодно по всему миру, в том числе в России, проводится мониторинг вирусов гриппа, чтобы определить доминирующие генетические группы и отобрать среди них штаммы, которые войдут в состав противогриппозных вакцин.
Цели исследования: анализ циркуляции вирусов гриппа в России в 2019–2023 гг., проведение филогенетического и молекулярного анализа последовательностей гемагглютинина (HA) вирусов гриппа, выявление мутаций резистентности к ингибиторам нейраминидазы (NA) и ингибиторам ионного канала М2-белка (M2).
Материалы и методы. Исследованы биологические образцы, содержащие РНК вирусов гриппа: 410 А(H1N1)pdm09, 147 А(H3N2) и 167 В(Виктория). Осуществляли секвенирование фрагментов сегментов HA, NA, M, проводили обработку и анализ данных.
Результаты. Исследованы нуклеотидные последовательности HA, NA, M вирусов гриппа А(H1N1)pdm09, А(H3N2) и B(Виктория), циркулировавших в 2019–2023 гг. Наибольшая вариабельность HA наблюдалась у вирусов А(H3N2). Все вирусы гриппа А(H1N1)pdm09 сезона 2022–2023 гг. имели не встречавшуюся ранее мутацию Е224А в HA, которая увеличивает его сродство к α-2,3-сиаловым кислотам — рецепторам, локализованным в лёгких человека, с которыми связывается вирус посредством HA. У 2 и 3% вирусов А(H1N1)pdm09 в сезонах 2019–2020 и 2022–2023 гг. соответственно в рецептор-связывающем сайте HA обнаружена мутация D222N, которая ассоциирована с более тяжёлым заболеванием. Мутация устойчивости к осельтамивиру и занамивиру H275Y в NA выявлена у 2,3% вирусов гриппа A(H1N1)pdm09 в 2022–2023 гг. Во всех исследованных вирусах гриппа A(H3N2) и B мутации устойчивости к осельтамивиру и занамивиру в NA не обнаружены. По данным секвенирования, во всех исследованных вирусах гриппа A(H1N1)pdm09 и A(H3N2) имелась мутация устойчивости к адамантанам S31N в M2.
Выводы. Обнаружение мутаций, затрагивающих антигенные и рецептор-связывающие сайты HA, а также мутаций резистентности в NA и M2 подтверждает необходимость постоянного генетического надзора за вирусами гриппа. Подавляющее большинство циркулирующих в настоящее время вирусов сохраняют чувствительность к ингибиторам NA.
Ключевые слова
Полный текст
Введение
Вирусы гриппа (Influenza virus) — это оболочечные вирусы семейства Orthomyxoviridae, которые классифицируются на 4 рода: вирусы гриппа A, B, C и D. Наибольшую опасность для здоровья человека, представляют вирусы гриппа A и B [1].
Вирусы гриппа A являются причиной большинства ежегодных эпидемий и всех периодических пандемических заболеваний человека. Они подразделяются на подтипы в соответствии с комбинацией 2 поверхностных гликопротеинов, расположенных в липидной мембране вирионов: гемагглютинина (HA) и нейраминидазы (NA). У птиц, основных естественных животных — резервуаров вирусов гриппа A, описаны 16 HA и 9 NA; ещё 2 HA и NA обнаружены у летучих мышей [2]. В настоящее время вирусы гриппа A подтипов H1N1pdm09 и H3N2 вызывают наибольшее число эпидемических заболеваний у людей1.
Вирусы гриппа B специфичны для людей (описаны также случаи у тюленей [3]) и подразделяются на 2 эволюционные линии, значительно различающиеся по антигенным свойствам (B/Виктория и B/Ямагата).
Вирусы гриппа находятся в состоянии постоянной эволюции в своих резервуарах, чему способствует высокая скорость мутаций из-за отсутствия у РНК-зависимой РНК-полимеразы механизмов коррекции ошибок в процессе репликации РНК вируса [4]. Основным резервуаром для вирусов гриппа А служат дикие перелётные птицы и другие млекопитающие, включая человека, в то время как вирусы гриппа В и С резервуара в дикой природе не имеют. Кумулятивные изменения в последовательностях, кодирующих HA и NA, приводят к антигенному дрейфу вирусов гриппа A и B: изменяется структура антигенных поверхностей, распознаваемых специфическими антителами, что способствует ежегодной эпидемии [5]. Для вирусов гриппа А также возможен антигенный сдвиг: сегменты, кодирующие HA (и в меньшей степени NA), эволюционировавшие в составе вирусов гриппа животных, могут комбинироваться с сегментами вируса гриппа человека с образованием новых штаммов — реассортантов, способных вызывать пандемии [4, 5]. С 1889 г. известны 5 пандемий вирусов гриппа A, самая серьёзная из которых была вызвана в 1918 г. подтипом H1N1, а последняя — в 2009 г. подтипом A(H1N1)pdm09 [4, 6]. Подтип H3N2 начал циркулировать c пандемии 1968 г. [7]. C 1971 г. ежегодно с большей или меньшей интенсивностью циркулируют оба подтипа вируса гриппа А — H3N2 и H1N1 (с 2009 г. — H1N1pdm09) и обе эволюционные линии вируса гриппа В.
Вирус гриппа B был идентифицирован в 1940 г. Две антигенные линии вирусов — B/Victoria/2/87-подобные (Виктория) и B/Yamagata/16/88-подобные (Ямагата) — коциркулируют с 1983 г. [8].
Вирусы гриппа В/Виктория быстрее эволюционируют с бóльшим давлением позитивной селекции, чем вирусы линии В/Ямагата [9], при этом последние не циркулировали с марта 2020 г.: с тех пор появлялись единичные сообщения об обнаружении вирусов гриппа В/Ямагата, однако без подтверждения секвенированием НА вируса2.
Под эгидой ВОЗ во всех странах мира проводится мониторинг вирусов гриппа, включая их типирование, исследование генетических характеристик и антигенных свойств, чтобы отобрать среди доминирующих антигенных групп штаммы вирусов, которые войдут в состав противогриппозных вакцин в следующем эпидемическом сезоне. Выбор вакцинных штаммов с определёнными иммуногенными свойствами необходим, чтобы обеспечивать иммунный ответ против вирусов гриппа [10]. Широкомасштабный генетический мониторинг позволяет проводить глубокую дифференцировку до клайдов и субклайдов и определять тренд эволюции вирусов гриппа: появление, распространение и исчезновение из циркуляции определённых генетических вариантов.
В ходе эволюции наиболее значимые изменения затрагивают НА вирусов гриппа. Гликопротеин HA вирусов гриппа синтезируется как единая полипептидная цепь, которая в дальнейшем подвергается протеолитическому расщеплению на 2 субъединицы: HA1 и HA2. HA1 отвечает за связывание вируса с сиаловыми кислотами (СК) на поверхности мембраны клетки, HA2 обеспечивает слияние мембран вируса и эндосомы.
Антигенные сайты, расположенные на вершине HA1 рядом с рецептор-связывающим сайтом, являются главными мишенями для нейтрализующих антител человека. Гликозилирование HA ассоциировано со многими свойствами, включая иммуногенность и специфичность к рецепторам, играет важную роль в защите антигенных сайтов от нейтрализующих антител [11]. Паттерн гликозилирования более вариабелен в HA1, чем в HA2, которая более консервативна [12].
Аминокислотные (АК) замены в рецептор-связывающем домене HA влияют на способность связываться с поверхностью клетки хозяина, что изменяет вирулентность вируса [13]. На поверхности клеток верхних дыхательных путей человека расположены СК преимущественно α-2,6-типа, тогда как в нижних дыхательных путях — α-2,3. Усиление сродства вируса к α-2,3-СК может увеличивать тяжесть заболевания [14].
Эволюции вирусов гриппа B/Виктория, помимо точечных мутаций, способствуют инсерции и делеции АК НА в области связи с рецептором, которые приводят к ускользанию от иммунного ответа [15].
АК-замены в NA (вирусов гриппа А и В) и M2 (вирусов гриппа А) снижают эффективность лекарственных препаратов: ингибиторов НА (осельтамивира, занамивира) и ингибиторов ионного канала М2 — адамантанов (амантадина и римантадина). По информации центров по контролю и профилактике заболеваний США, в настоящий момент практически 100% вирусов гриппа А устойчивы к действию амантадина и римантадина [16].
Цели работы — выполнить молекулярно-генетический анализ сегментов НА, NA и М вирусов гриппа A(H1N1)pdm09, A(H3N2) и B/Виктория, циркулировавших в России в эпидемические сезоны 2019–2023 гг., определить мутации АК в HA по сравнению с вакцинными штаммами, их возможное влияние на антигенные свойства и силу связи со специфическими рецепторами; провести филогенетический анализ по гену НА, а также анализ NA и M2 на наличие молекулярных маркеров резистентности к противовирусным препаратам.
Материалы и методы
Обнаружение РНК вирусов гриппа A и B в биологическом материале (мазках из носоглотки и ротоглотки, мокроте, аспиратах из трахеи, бронхоальвеолярном лаваже), полученных в результате рутинного мониторинга за вирусами гриппа, проводилось в лабораториях Центров гигиены и эпидемиологии Роспотребнадзора 50 субъектов Российской Федерации (Центрального, Северо-Западного, Южного, Северо-Кавказского, Приволжского, Уральского и Дальневосточного федеральных округов) методом полимеразной цепной реакции (ПЦР) с гибридизационно-флюоресцентной детекцией продуктов амплификации. Исследование проводилось при добровольном информированном согласии пациентов. Протокол исследования одобрен Этическим комитетом ФБУН ЦНИИ Эпидемиологии Роспотребнадзора (протокол № 3 от 27.03.2020). В случае неблагоприятного исхода заболевания исследовался посмертный материал (аутоптаты лёгких).
Для выполнения молекулярно-генетического анализа методом секвенирования нуклеиновых кислот биологический материал поступал в Референс-центр по мониторингу за инфекциями верхних и нижних дыхательных путей на базе Лаборатории молекулярной диагностики и эпидемиологии инфекций дыхательных путей ЦНИИ Эпидемиологии.
Экстракцию РНК вирусов гриппа из биологического материала и последующую реакцию обратной транскрипции проводили с использованием комплектов реагентов «РИБО-преп» и «РЕВЕРТА-L» («АмплиСенс», ЦНИИ Эпидемиологии). ПЦР для подтверждения обнаружения РНК вирусов гриппа осуществляли с использованием наборов реагентов «АмплиСенс Influenza virus A/B», «АмплиСенс Influenza virus A-тип-FL», «АмплиСенс Influenza virus A/H1-swine-FL», «АмплиСенс Influenza virus B-тип-FL» (ЦНИИ Эпидемиологии).
Для амплификации фрагментов генов HA, NA, M вирусов гриппа A/H1N1pdm09 и A/H3N2, HA и NA вирусов гриппа B проводили ПЦР с детекцией методом электрофореза на амплификаторах «Терцик» («ДНК-технология») с использованием реагентов «АмплиСенс» (ЦНИИ Эпидемиологии).
Фрагменты амплификации отдельных сегментов вирусов гриппа (НА, NA, M) секвенировали по методу Сэнгера в Научной группе генной инженерии и биотехнологии ЦНИИ Эпидемиологии методом «cycle sequence» с помощью набора «BigDye Terminator v1.1 Cycle Sequencing Kit» («Applied Biosystems by Thermo Fisher Scientific») на секвенаторе «3500xL Genetic Analyzer» («Applied Biosystems»).
Результаты секвенирования анализировали в Референс-центре по мониторингу за инфекциями верхних и нижних дыхательных путей с использованием блока программ DNASTAR (SeqMan, EditSeq, MegAlign). Полученные нуклеотидные последовательности были загружены в международную базу данных GISAID, могут быть отфильтрованы по Search patterns «CRIE». Филогенетический анализ проводили с помощью программы «BioNumerics v. 6.6» методом UPGMA. Для отслеживания АК-мутаций использовали онлайн-платформы Nextclade, FluSurver. Нумерация аминокислот приведена по соответствующему подтипу вирусов гриппа.
Результаты
Исследованы биологические образцы, содержащие РНК вирусов гриппа: 410 А(H1N1)pdm09, 147 А(H3N2) и 167 В(Виктория), циркулировавшие в эпидемических сезонах 2019–2020, 2020–2021, 2021–2022, 2022–2023 гг. В 1 образце (мазки из верхних дыхательных путей) сезона 2022–2023 гг. были одновременно обнаружены вирус гриппа A(H1N1)pdm09 и вирус гриппа В. Из рис. 1 видно, что количество исследованных вирусов гриппа в разные годы менялось, что обусловлено различной интенсивностью эпидемического процесса гриппа в разные сезоны.
Рис. 1. Количество вирусов гриппа, исследованных в Референс-центре по мониторингу за инфекциями верхних и нижних дыхательных путей в эпидемических сезонах 2019–2023 гг.
Fig. 1. Number of influenza viruses tested at the Reference center for monitoring upper and lower respiratory tract infections in 2019–2023.
Методом секвенирования были получены и проанализированы нуклеотидные последовательности генов NA, HA, M вирусов гриппа A(H1N1)pdm09 и A(H3N2) и генов NA и HA вируса гриппа B (табл. 1).
Таблица 1. Количество секвенированных сегментов
Table 1. Number of sequenced segments
Вирус гриппа Influenza virus | Сегмент Segment | Количество (от выздоровевших/от умерших) Number (from the recovered/from the deceased) | |||
2019–2020 | 2020–2021 | 2021–2022 | 2022–2023 | ||
(H1N1)pdm09 | HA | 59 (55/4) | – | – | 351 (296/55) |
NA | 59 (55/4) | – | – | 87 (69/18) | |
M | 59 (55/4) | – | – | 28 (16/2) | |
A(H3N2) | HA | 11 (11/0) | 2 (1/0) | 124 (124/0) | 10 (10/0) |
NA | 2 (2/0) | 1 (1/0) | 44 (44/0) | 1 (1/0) | |
M | 1 (1/0) | 2 (1/0) | 34 (34/0) | 1 (1/0) | |
В | HA | 56 (53/3) | – | 7 (7/0) | 104 (102/2) |
NA | 35 (34/1) | – | 1 (1/0) | 36 (36/0) | |
Проведён филогенетический и молекулярно-генетический анализ вирусов гриппа. Кластеризацию вирусов гриппа по клайдам и субклайдам проводили на основании нуклеотидных последовательностей HA, для сравнения использовали последовательности рекомендованных в каждом конкретном сезоне для Северного полушария вакцинных штаммов, культивированных на куриных эмбрионах, поскольку именно такие вакцины широко используются в России.
Вирусы гриппа A(H1N1)pdm09
Распределение секвенированных вирусов по генетическим кластерам (клайдам, субклайдам и подгруппам) и результаты сравнения с вакцинным штаммом каждого сезона представлены в табл. 2. Гомология нуклеотидных последовательностей гена HA исследованных вирусов и вакцинных штаммов варьировалась в диапазоне 99,2–97,4% в зависимости от принадлежности к генетическому кластеру. Наибольшие различия (до 2,6%) наблюдали в сезоне 2019–2020 гг.
Таблица 2. Результаты анализа нуклеотидных последовательностей НА вирусов гриппа A(H1N1)pdm09, поступивших в Референс-центр в 2019–2023 гг.
Table 2. Results of nucleotide sequence analysis of НА influenza A(H1N1)pdm09 viruses received by the Reference center in 2019–2023
Эпидемический сезон, годы Epidemic season | Вакцинный штамм (генетический кластер) Vaccine strain (genetic cluster) | Количество исследованных образцов Number of samples studied | Генетический кластер Genetic cluster | Гомология гена НА с вакцинным, % HA gene homology with the vaccine strain, % |
2019–2020 | A/Brisbane/02/2018 (6В.1A.1) | 39 | 6В.1A.5a | 98,4–99,2 |
18 | 6В.1A.5a.1 | 98,2–98,6 | ||
1 | 6В.1A.5a.2 | 99,2 | ||
1 | 6В.1A.7 | 98,2 | ||
2022–2023 | A/Victoria/2570/2019 (6В.1A.5a.2) | 351 | 6В.1A.5a.2a | 97,4–98,9 |
По результатам секвенирования, вирусы A(H1N1)pdm09 сезона 2019–2020 гг. относились к субклайду 6В.1A.5, подгруппам 5a, 5a.1, 5a.2 и субклайду 6В.1A.7. В этом сезоне превалировали вирусы подгруппы 5a субклайда 6В.1A.5 (66%), для которой характерны АК-замены N129D и T185A в HA1. К подгруппе 6В.1A.5a.1, для которой характерны АК-замены D187A, Q189E, относились 31% исследованных вирусов. Один вирус принадлежал к подгруппе 6В.1A.5a.2 (АК-замены N156K, L161I, V250A), 1 вирус — к субклайду 6В.1A.7 (АК-замены K302T в HA1 и I77M, N169S, E179D в HA2).
Штамм A/Brisbane/02/2018, рекомендованный для включения в состав вакцины на эпидемический сезон 2019–2020 гг. для Северного полушария, принадлежал к субклайду 6В.1A.1.
В сезонах 2020–2021 и 2021–2022 гг. вирусы гриппа A(H1N1)pdm09 в референс-центр не поступали, поскольку не циркулировали.
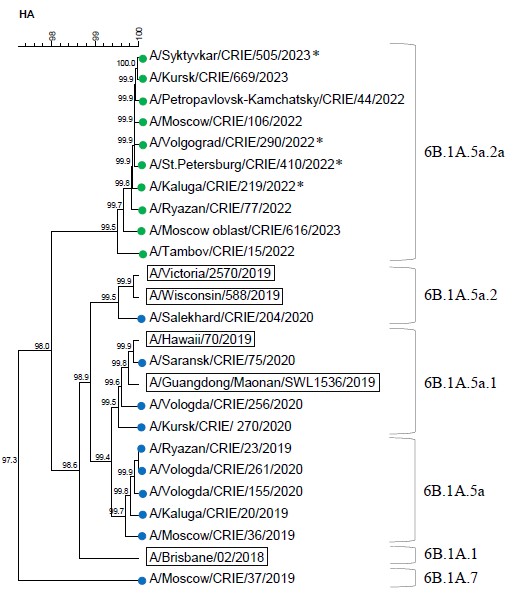
Все исследованные вирусы A(H1N1)pdm09 сезона 2022–2023 гг. относились к клайду 6В.1A.5a.2a, для которого характерны АК-замены K54Q, A186T, Q189E, E224A, R259K, K308R. Вакцинный штамм этого сезона A/Victoria/2570/2019 причислялся к подгруппе 6В.1A.5a.2.
Результаты филогенетического анализа по гену НА вирусов гриппа A подтипа H1N1pdm09 в сезонах 2019–2023 гг. отражены на рис. 2. На дендрограмме представлена выборка секвенированных последовательностей НА вирусов гриппа, максимально отражающая их генетическое разнообразие.
Рис. 2. Дендрограмма по гену НА вирусов гриппа A(H1N1)pdm09 (данные Референс-центра ЦНИИ Эпидемиологии за 2019–2020, 2022–2023 гг.).
Вакцинные штаммы выделены прямоугольниками. Синими и зелёными кругами обозначены последовательности вирусов, циркулировавших в 2019–2020 и 2022–2023 гг. соответственно. *Летальный случай.
Fig. 2. Dendrogram of the HA gene of influenza A(H1N1)pdm09 viruses (data of the CRIE Reference center, 2019–2020, 2022–2023).
Vaccine strains are indicated by rectangles. Blue and green circles indicate the sequences of viruses circulating in 2019–2020 and 2022–2023 influenza seasons, respectively.
Fatal cases are marked with an asterisk.
В молекуле НА вируса гриппа A(H1N1)pdm09 выделяют 5 антигенных сайтов, к которым относят следующие АК-позиции: Sa (121–122 и 150–162), Sb (184–195), Ca1 (163–167, 200–202 и 232–235), Ca2 (133–139 и 218–219), Cb (67–72) [17].
В АК-последовательности НА вирусов 2019–2020 гг. были обнаружены 7–12 мутаций в сравнении с вакцинным штаммом A/Brisbane/02/2018. Из них 7 находились в антигенных сайтах: Sa — S121I/N (n = 2), N156K (n = 1), L161I (n = 1), Sb — T185I (n = 57), D187A (n = 18), Q189E (n = 16), S190N (n = 1). Рядом с антигенным сайтом Ca1 обнаружена замена R205K (n = 2). Мутации D187A (n = 18), R221K (n = 2) и D222N (n = 1) находились в рецептор-связывающем сайте. Один из образцов относился к субклайду 6B.1A7 и нёс АК-замены E68D (в Cb), T120A, S121N (в Sa1), R223Q, K302T (в HA1) и I77M, N169S, E179D (в HA2).
Вирусы гриппа A(H1N1)pdm09 2022–2023 гг. относительно вакцинного штамма содержали 6–12 АК-замен. Чаще обнаруживались замены R259K, K54Q, A186T, Q189E, E224A, K308R/G в HA1. Часть мутаций затрагивала антигенные сайты: Sa — S121N (n = 1), S122L (n = 1), K154R (n = 1), G155E (n = 1), N162S (n = 1, потеря сайта гликозилирования), Sb — A186T (n = 351), D187V (n = 1), Q189E (n = 351), N194S (n = 1), Ca1 — V234I (n = 1), Cb — S69P (n = 1), L70F (n = 1). Рядом с антигенными сайтами обнаружены АК-замены N125H (n = 1), R205K (n = 3). Мутации D187V (n = 1) и D222N (n = 12) находились в рецептор-связывающем сайте. У 2 образцов вирусов обнаружена мутация D145N в HA2, которая приводит к образованию дополнительного сайта гликозилирования.
В 2019–2020 гг. у всех вирусов гриппа A(H1N1)pdm09 отсутствовали мутации, обусловливающие устойчивость к осельтамивиру и занамивиру, тогда как в 2022–2023 гг. имевшие мутацию H275Y в гене NA вирусы составили 2,3% и были обнаружены в респираторных мазках непривитых от гриппа пациентов из Архангельской области в декабре 2022 г. Все вирусы гриппа A(H1N1)pdm09, для которых была получена последовательность гена M, были устойчивы к адамантанам (мутация S31N в M2).
Вирусы гриппа A(H3N2)
Распределение исследованных вирусов гриппа A(H3N2) по генетическим кластерам и результаты сравнения с вакцинным штаммом каждого сезона представлены в табл. 3. Гомология нуклеотидных последовательностей гена HA исследованных вирусов и вакцинных штаммов варьировалась в диапазоне 99,0–95,8% в зависимости от принадлежности к генетическому кластеру. Максимальные различия (3,6–4,2%) наблюдались в сезоне 2019–2020 гг.
Таблица 3. Результаты анализа нуклеотидных последовательностей НА вирусов гриппа A(H3N2), поступивших в Референс-центр в 2019–2023 гг.
Table 3. Results of НА nucleotide sequence analysis of influenza A(H3N2) viruses received by the Reference center in 2019–2023
Эпидемический сезон, годы Epidemic season, years | Вакцинный штамм (генетический кластер) Vaccine strain (genetic cluster) | Количество исследованных образцов Number of samples studied | Генетический кластер Genetic cluster | Гомология гена НА с вакцинным штаммом, % HA gene homology with the vaccine strain, % |
2019–2020 | A/Kansas/14/2017 (3C.3a.1) | 7 | 3C.2a1b.1b | 96,2–96,4 |
4 | 3C.2a1b.2a | 95,8–96,0 | ||
2020–2021 | A/Hong Kong/2671/2019 (3C.2a1b.1b, ранее | previously 3C.2a1b + T135K-B) | 2 | 3C.2a1b.2a.2a.2 | 97,3–97,4 |
2021–2022 | A/Cambodia/e0826360/2020 (3C.2a1b.2a.1a) | 3 | 3C.2a1b.2a.2 | 98,6–98,7 |
3 | 3C.2a1b.2a.2a.1 | 98,6–98,7 | ||
105 | 3C.2a1b.2a.2a.2 | 98,1–98,9 | ||
13 | 3C.2a1b.2a.2c | 98,6–98,9 | ||
2022–2023 | A/Darwin/9/2021 (2a, ранее | previously 3C.2a1b.2a.2a) | 2 | 3C.2a1b.2a.2a | 99,4 |
2 | 3C.2a1b.2a.2a.1b | 98,9–99,0 | ||
3 | 3C.2a1b.2a.2a.3a.1 | 98,5–98,6 | ||
3 | 3C.2a1b.2a.2b | 98,5 |
Вирусы гриппа A(H3N2) сезона 2019–2020 гг. были отнесены к группе 3C.2a1b, кластерам 3C.2a1b.1b и 3C.2a1b.2a. Вакцинный штамм A/Kansas/14/2017 относился к группе 3C.3a.1 (характерны АК-замены в позициях S91N, N144K, последняя приводит к потере потенциального сайта гликозилирования, F193S в HA1 и D160N в HA2). В кластер 3C.2a1b.1b, для которого характерны АК-замены в позициях S137F, A138S, F193S в HA1, вошли 64% образцов. К кластеру 3C.2a1b.2a с характерными АК-заменами в позициях K83E, Y94N в HA1 и I193M в HA2 были отнесены 36% образцов.
В молекуле HA вируса гриппа A(H3N2) к антигенным сайтам относят АК-позиции:
- A (122, 124, 126, 130–133, 135, 137, 138, 140, 142–146, 150, 152, 168);
- B (128, 129, 155–160, 163–165, 186–190, 192–194, 196–198);
- C (44–48, 50, 51, 53, 54, 273, 275, 276, 278–280, 294, 297, 299, 300, 304, 305, 307–312);
- D (96, 102, 103, 117, 121, 167, 170–177, 179, 182, 201, 203, 207–209, 212–219, 226–230, 238, 240, 242, 244, 246–248);
- E (57, 59, 62, 63, 67, 75, 78, 80–83, 86–88, 91, 92, 94, 109, 260–262, 265) [18].
Три антигенных сайта перекрываются с рецептор-связывающим сайтом: сайт A — с петлей 130 (135, 136, 137, 138, 153); сайт B — со спиралью 190 (186, 190, 194, 195); сайт D — с петлей 220 (226 и 228) [19].
Оба вируса гриппа A(H3N2) сезона 2020–2021 гг. относились к субклайду 2a.2 подгруппы 3C.2a1b.2a.2a.2, для которой характерны АК-замены Y159N, T160I — потеря сайта гликозилирования, L164Q, G186D, D190N в HA1, тогда как вакцинный штамм A/Hong Kong/2671/2019 принадлежал к кластеру 3C.2a1b.1b.
В сезоне 2021–2022 гг., как и в предыдущем, все вирусы гриппа A(H3N2) относились к подгруппе 3C.2a1b.2a.2. По классификации 2022–2023 гг. вирусы были отнесены к 4 субклайдам: превалировали вирусы 3C.2a1b.2a.2a.2 (АК-замены D53G, R201L, S219Y), реже обнаруживались 3C.2a1b.2a.2c (АК-замены S205F, A212T), ещё реже — 3C.2a1b.2a.2 и 3C.2a1b.2a.2a.1 (АК-замены D53G, D104G, K276R). Вакцинный штамм A/Cambodia/e0826360/2020 относился к группе 3C.2a1b.2a, но другой генетической подгруппе 3C.2a1b.2a.1a, для которой характерны АК-замены L157I, K220R.
Вирусы гриппа А(H3N2) сезона 2022–2023 гг. принадлежали к клайду 3C.2a1b.2a.2, как и вакцинный штамм A/Darwin/9/2021, входивший в состав вакцин, применяемых в Северном полушарии в 2022–2023 гг. Внутри клайда вирусы дифференцировались на субклайды: 3 относились к 3C.2a1b.2a.2a.3a.1 (АК-замена I140K), 3 — к 3C.2a1b.2a.2b (характерны АК-замены E50K, F79V, I140K), 2 принадлежали к 3C.2a1b.2a.2a.1b (АК-замены I140K, R299K). Только 2 НА были близки вакцинному штамму A/Darwin/9/2021 (субклайд 3C.2a1b.2a.2a, для которого характерна АК-замена H156S).
Результаты филогенетического анализа по гену НА вирусов гриппа A подтипа H3N2 представлены на рис. 3. В сезоне 2019–2020 гг. в АК-последовательности НА вирусов были обнаружены 21–23 мутации по сравнению с вакцинным штаммом A/Kansas/14/2017, причём 19 из них находились в антигенных сайтах:
Рис. 3. Дендрограмма по гену НА вирусов гриппа A(H3N2) (данные Референс-центра ЦНИИЭ за 2019–2023 гг.).
Вакцинные штаммы выделены прямоугольниками. Синими, фиолетовыми, красными и зелёными кругами обозначены последовательности вирусов, циркулировавших в 2019–2020, 2020–2021, 2021–2022 и 2022–2023 гг. соответственно. *Летальный случай.
Fig. 3. Dendrogram of the HA gene of influenza A(H3N2) viruses (data from the CRIE Reference center, 2019–2023).
Vaccine strains are indicated by rectangles. Blue, violet, red and green circles indicate the sequences of viruses circulating in 2019–2020, 2020–2021, 2021–2022 and 2022–2023 influenza seasons, respectively. Fatal cases are marked with an asterisk.
- A — T131K (n = 3), T135K (n = 7), S137F (n = 7), I140K (n = 7), K144S (n = 10);
- B — A128T (n = 3), S159Y (n = 10), K160T (n = 10), N190D (n = 10), S193F (n = 3);
- D — N121K (n = 10), N171K (n = 10), V230I (n = 1), T246N (n = 10);
- E — E62G (n = 10), K83E (n = 3), N91S (n = 10), K92R (n = 10), Y94N/S (n = 3).
Три замены локализованы в рецептор-связывающем сайте: в петле 130 — T135K, S137F, в спирали 190 — N190D. Мутации I77V, M149I и G155E в HA2 содержали 11 вирусов.
Обнаружены мутации, которые приводят к появлению новых потенциальных сайтов N-гликозилирования: A128T (у 3 вирусов подгруппы 3C.2a1b.2a), K160T (у всех 10 вирусов), T246N (у всех 10 вирусов). У 7 вирусов подгруппы 3C.2a1b.1b имелась мутация T135K, которая приводит к потере сайта N-гликозилирования.
У вирусов A(H3N2), исследованных в сезоне 2020–2021 гг., в АК-последовательностях НА имелись 22–24 замены по сравнению с вакцинным штаммом A/Hong Kong/2671/2019, 16 из них находились в антигенных сайтах обоих вирусов: A — T131K, K135T, F137S, S138A; B — A128T, H156S, Y159N, L164Q, V186D, D190N; С — D53G; D — R201K, S219Y; E — K83E, Y94N. У 1 вируса в сайте D была мутация I214V; 5 замен были локализованы в рецептор-связывающем сайте: в петле 130 — K135T, F137S, S138A, в спирали 190 — V186D, D190N. Дополнительно вне антигенных сайтов у обоих вирусов обнаружены мутации N225D, A128T и K135T, последние из которых приводят к образованию новых потенциальных сайтов N-гликозилирования.
В сезоне 2021–2022 гг. АК-последовательности НА содержали 9–12 мутаций по сравнению с вакцинным штаммом A/Cambodia/e0826360/2020.
У 124 вирусов всех субклайдов выявлены следующие замены в антигенных сайтах: B — Y159N (у 1 — Y159S), K160I, L164Q, R186D, D190N, P198S; D — N171K. Замены R186D, D190N находились в рецептор-связывающем сайте. Три вируса субклайда 3C.2a1b.2a.2 имели мутации: A — I140K, D — R201I. У 1 вируса в антигенном сайте D обнаружена мутация S219Y.
Три вируса субклайда 3C.2a1b.2a.2a.1 содержали мутации: С — D53G, K276R; B — H156S; вне антигенных сайтов — D104G; 105 вирусов субклайда 3C.2a1b.2a.2a.2 имели мутации в антигенном сайте D — R201K, S219Y. У 104 вирусов вне антигенных сайтов была замена I25V. У 4 вирусов были выявлены мутации, которые приводят к потере сайта гликозилирования: у 3 вирусов — N122D, у 1 — N165K. Тринадцать вирусов субклайда 3C.2a1b.2a.2с имели мутацию A212T в антигенном сайте D. У 12 вирусов вне антигенных сайтов была замена S205F. У 2 вирусов обнаружена мутация S124N, которая приводит к потере сайта гликозилирования.
В сезоне 2022–2023 гг. исследованные вирусы A(H3N2) в НА имели 2–9 АК-замен по сравнению с вакцинным штаммом A/Darwin/9/2021 и 3–9 замен по сравнению с A/Darwin/6/2021 (последовательность штамма A/Darwin/9/2021 отличается от A/Darwin/6/2021 мутацией G53D). 2 вируса субклайда 3C.2a1b.2a.2a имели мутацию в сайте D — I217V. У 2 вирусов субклайда 3C.2a1b.2a.2a.1b были локализованы мутации в сайте A — I140K, С — K276R и R299K. Три вируса субклайда 3C.2a1b.2a.2a.3a.1 содержали мутации в сайте С — E50K, D53N, A — I140K, B — I192F; 3 вируса субклайда 3C.2a1b.2a.2b имели мутации E50K в сайте C и I140K в сайте A. У 2 вирусов имелась AK-замена N96S в HA1, приводящая к появлению сайта N-гликозилирования, у 1 мутация N122D в HA1 приводила к потере сайта N-гликозилирования.
В NA исследованных вирусов гриппа A(H3N2) сезонов 2019–2023 гг. мутаций резистентности к осельтамивиру и занамивиру не обнаружено, в то время как все имели мутацию устойчивости к адамантанам S31N в M2.
Вирусы гриппа B
Все поступившие на исследование в 2019–2023 гг. вирусы гриппа B принадлежали к линии В/Виктория (по результатам ПЦР и секвенирования).
Распределение исследованных вирусов гриппа A(H3N2) по генетическим кластерам и результаты сравнения с вакцинным штаммом каждого сезона представлены в табл. 4. Гомология нуклеотидных последовательностей гена HA исследованных вирусов и вакцинных штаммов варьировала в диапазоне 99,7–98,2% в зависимости от принадлежности к генетическому кластеру. Максимальное сходство (99,0–99,7%) наблюдалось в сезоне 2022–2023 гг.
Таблица 4. Результаты анализа нуклеотидных последовательностей НА вирусов гриппа B/Виктория, поступивших в Референс-центр в 2019–2023 гг.
Table 4. Results of НА nucleotide sequence analysis of influenza B viruses of the Victoria lineage received by the Reference center in 2019–2023
Эпидемический сезон, годы Epidemic season, years | Вакцинный штамм (генетический кластер) Vaccine strain (genetic cluster) | Количество исследованных образцов Number of samples studied | Генетический кластер Genetic cluster | Гомология гена НА с вакцинным штаммом, % HA gene homology with the vaccine strain, % |
2019–2020 | B/Colorado/06/2017 (V1A.1, ранее | previously 1A(∆2)B) | 55 | V1A.3 | 98,2–98,8 |
1 | V1A.3a.1 | 98,6 | ||
2021–2022 | B/Washington/02/2019 (V1A.3, ранее | previously 1A(∆3)B) | 7 | V1A.3a.2 | 98,2–98,7 |
2022–2023 | B/Austria/1359417/2021 (V1A.3a.2) | 104 | V1A.3a.2 | 99,0–99,7 |
В сезоне 2019–2020 гг. 98% вирусов гриппа B относились к линии Виктория субклайду V1A.3 (1A(Δ3)B), для которого характерны тройная делеция АК-остатков 162–164 и АК-замены K136E, G133R в НА, остальные принадлежали к подгруппе V1A.3a.1, для которой характерны АК-замены в HA1 V117I, V220M. Вакцинный штамм на эпидемический сезон 2019–2020 гг. для Северного полушария B/Colorado/06/2017 относился к субклайду V1A.1 (1A(Δ2)B) линии Виктория, для которого характерны двойная делеция АК-остатков 162–163 и АК-замен в позициях D129G, I180V в HA1, R151K в HA2.
В сезоне 2021–2022 гг. 7 вирусов гриппа B принадлежали к линии Виктория субклайду V1A.3, подгруппе V1A.3a.2 (характерны АК-замены в позициях HA1 A127T, P144L, K203R). Вакцинный штамм B/Washington/02/2019, входивший в вакцины для Северного полушария в 2021–2022 гг., относился к линии Виктория субклайду V1A.3.
В сезоне 2022–2023 гг. все вирусы гриппа B относились к линии Виктория подгруппе V.1A.3a.2. Вакцинный штамм B/Austria/1359417/2021, входивший в вакцины в России в 2022–2023 гг., также принадлежал к линии Виктория подгруппе V.1A.3a.2. Результаты филогенетического анализа по гену HA вирусов гриппа B линии Виктория представлены на рис. 4.
Рис. 4. Дендрограмма по гену НА вирусов гриппа B/Виктория.
Данные Референс-центра ЦНИИ Эпидемиологии за 2019–2020, 2020–2021 и 2022–2023 гг. Вакцинные штаммы выделены прямоугольниками. Синими, красными и зелёными кругами обозначены последовательности вирусов, циркулировавших в 2019–2020, 2021–2022 и 2022–2023 гг. соответственно. *Летальный случай.
Fig. 4. Dendrogram of the HA gene of influenza B viruses of the Victoria lineage.
Data from the CRIE Reference center, 2019–2020, 2020–2021, 2022–2023. Vaccine strains are indicated by rectangles. Blue, red and green circles indicate the sequences of viruses circulating in 2019–2020, 2021–2022 and 2022–2023 influenza seasons, respectively. Fatal cases are marked with an asterisk.
В молекуле НА вируса гриппа B выделяют 4 антигенных сайта: петля 120 и прилегающие регионы (116–137), петля 150 (141–150), петля 160 (162–167), спираль 190 и окружающие её области (194–202) [20]. Рецептор-связывающий сайт сформирован спиралью 190 (193–202), петлёй 240 (237–242), петлёй 140 (136–143) [21].
В сезоне 2019–2020 гг. АК-последовательности НА вирусов имели 8–12 мутаций по сравнению с вакцинным штаммом B/Colorado/06/2017, в том числе все — дополнительную делецию АК в позиции 164. В антигенных сайтах находились 11 замен: в петле 120 — I117V (n = 2), R118K (n = 2), N126K (n = 5), A127T (n = 1), E128K (n = 1), D129N (n = 55), G133R (n = 50), Y135D (n = 1), K136E (n = 56); в петле 160 — N166D (n = 1); в спирали 190 — N197D (n = 1). Три замены находились в рецептор-связывающем сайте: в спирали 190 — N197D; в петле 240 — P241Q (n = 1); в петле 140 — K136E. По 1 вирусу имели замены N166D, N197D и N233S, которые приводят к потере сайта гликозилирования.
В сезоне 2021–2022 гг. вирусы гриппа B имели 8–9 АК-замен в НА по сравнению с вакцинным штаммом B/Washington/02/2019. АК-замены находились в 5 позициях антигенных сайтов: в петле 120 — A127T (n = 7), R133G (n = 7); в петле 150 — P144L (n = 7), N150K (n = 7); в спирали 190 — N197D/E (n = 7). Одна замена находилась в рецептор-связывающем сайте, в спирали 190 — N197D/E. Мутации N197D у 6 вирусов и N197E у 1 вируса приводят к потере сайта гликозилирования.
В НА вирусов гриппа B в сезоне 2022–2023 гг. имелись 1–6 мутаций по сравнению с вакцинным штаммом B/Austria/1359417/2021. Семь замен находились в антигенных сайтах: в петле 120 — T121N (n = 25), H122N (n = 3), E128K (n = 67), в петле 150 — G149E (n = 1), в спирали 190 — D197E (n = 40), T199A/I (n = 100). Две замены находились в рецептор-связывающем сайте, в спирали 190 — D197E, T199A/I. У 1 вируса в HA2 обнаружена замена T196I, которая привела к потере сайта гликозилирования.
В исследованных вирусах гриппа B сезонов 2019–2023 гг. мутации в гене NA, снижающие чувствительность к осельтамивиру и занамивиру, не обнаружены.
Обсуждение
Этиологическая структура гриппа в разные эпидемические сезоны в России и в мире в целом варьировалась. Поступавшие на исследование в Референс-центр образцы, содержащие вирусы гриппа, были отобраны в регионах России при скрининге больных случайным образом, поэтому можно считать, что их спектр и генетические характеристики отражают общие закономерности по стране и позволяют судить о структуре гриппа и генетическом разнообразии циркулировавших вирусов в России.
В сезоне 2019–2020 гг. в Референс-центр поступали в основном вирусы гриппа A(H1N1)pdm09 и B: 46,8% вирусов гриппа A(H1N1)pdm09, 8,7% вирусов гриппа A(H3N2), 44,5% вирусов гриппа В/Виктория. По данным Национального центра по гриппу ВОЗ, в России в этом сезоне присутствовали в популяции все 3 подтипа гриппа, при этом преобладал вирус гриппа В/Виктория (43,7%) [22]. По данным Европейского центра по профилактике и контролю заболеваний (ECDC), в европейском регионе среди вирусов, которые были типированы, обнаружены 51% A(H1N1)pdm09, 40,1% A(H3N2), 8,7% B/Виктория, 0,2% B/Ямагата3. Второе место по численности занимали вирусы A(H3N2), а не вирусы гриппа В/Виктория, как в России, что может быть связано с различным характером распространения данных типов вирусов в разных странах.
В сезоне 2020–2021 гг. в Референс-центре было исследовано только 2 вируса гриппа (A(H3N2)). По данным Национального центра по гриппу ВОЗ, в России при скрининге методом ПЦР-диагностики гриппа за весь сезон обнаружено всего 37 положительных материалов, а выделить в культуре и антигенно охарактеризовать удалось только 2 вируса гриппа В/Виктория [22]. В европейском регионе в этом сезоне также было отмечено снижение числа детектированных случаев гриппа на 99,4% по сравнению с сезоном 2019–2020 гг. Вирусы гриппа были распределены следующим образом: 14,2% A(H1N1)pdm09, 80,6% A(H3N2), 4,9% B/Виктория, 0,3% B/Ямагата [9], т. е. так же, как в России, преобладал вирус A(H3N2).
Низкая активность вирусов гриппа по всему миру была вызвана появлением нового бетакоронавируса SARS-CoV-2 в декабре 2019 г. в Китае и последующими ответными ограничительными мерами [23]. К ним относились ограничения передвижения людей: закрытие границ стран, приостановление полётов международных рейсов, карантин для пребывающих в страну и изоляция заболевших. Кроме того проводился контроль выполнения правил личной гигиены (частая обработка рук, использование дезинфицирующих средств, средств индивидуальной защиты), что способствовало снижению передачи вирусов гриппа. Сроки принятия этих мер напрямую коррелируют с резким падением заболеваемости гриппом в 2020–2021 гг.
В сезоне 2021–2022 гг. количество исследованных в Референс-центре вирусов значительно увеличилось, превалировал вирус гриппа A(H3N2) (94,7%), вирусы гриппа В составили 5,3%. Образцы, содержащие вирус гриппа A(H1N1)pdm09, не поступали. По данным Национального центра по гриппу ВОЗ, в России обнаруживался преимущественно подтип A(H3N2) с незначительным участием гриппа В/Виктория при полном отсутствии вируса A(H1N1)pdm09 [21]. Таким образом, этиологическая структура гриппа в этом сезоне в России была представлена вирусом гриппа A(H3N2) с незначительным вкладом вируса гриппа В.
По данным ECDC, в европейском регионе также преобладал вирус гриппа A(H3N2), гораздо реже обнаруживались A(H1N1)pdm09 (8,7%) и В: A(H3N2) — 90,9%, B/Виктория — 0,4%, B-Ямагата — < 0,1%4. Следует отметить, что несмотря на сообщения о единичных находках вирусов гриппа В/Ямагата, в доступных базах данных генетической информации не представлены нуклеотидные последовательности НА вирусов гриппа В/Ямагата, обнаруженные после марта 2020 г., а значит, их принадлежность к линии Ямагата не была подтверждена секвенированием.
Спектр исследованных в сезоне 2022–2023 гг. в Референс-центре вирусов гриппа выглядел следующим образом: 75,5% вирусов гриппа A(H1N1)pdm09, 2,1% вирусов гриппа A(H3N2), 22,4% вирусов гриппа В/Виктория. Среди поступивших и исследованных вирусов 12%, преимущественно гриппа A(H1N1)pdm09 и в меньшей степени гриппа В/Виктория составили вирусы, обнаруженные в аутопсийном материале, что свидетельствовало об увеличении тяжести заболевания гриппом в сравнении с сезоном 2021–2022 гг., когда превалировал вирус гриппа A(H3N2) и аутопсийный материал не поступал (табл. 1). Увеличение числа летальных исходов при гриппе в сезоны активной циркуляции вируса гриппа A(H1N1)pdm09 можно объяснить большим сродством этого подтипа вируса гриппа А к α-2,3-СК, что облегчает его распространение в лёгких и чаще приводит к развитию пневмонии. Кроме того, более тяжёлой эпидемии мог способствовать низкий уровень иммунитета населения из-за отсутствия контакта с вирусом гриппа A(H1N1)pdm09 в течение сезонов 2020–2021 и 2021–2022 гг.
По данным Национального центра по гриппу ВОЗ, в целом по России на пике эпидемического подъёма гриппа и острых респираторных вирусных заболеваний вирусы гриппа обнаруживались в 30% образцов больных, исследованных при скрининге методом ПЦР, при этом превалировал подтип A(H1N1)pdm095. В сезоне 2022–2023 гг., по данным ECDC, в европейском регионе структура гриппа отличалась от таковой в России: 47,3% A(H1N1)pdm09, 47,9% A(H3N2), 4,8% B/Виктория, 0% В/Ямагата6, что могло быть связано с различным характером распространения данных типов вирусов в разных странах.
Полученные нами данные распределения вирусов A(H1N1)pdm09 по генетическим группам совпали с данными НИИ гриппа им. А.А. Смородинцева: в сезоне 2019–2020 гг. преобладала генетическая подгруппа 6B.1A5 (референс-штамм A/Норвегия/3433/2018)7, в сезоне 2022–2023 гг. — генетическая подгруппа 6В.1А.5а.2 (референс-вирус A/Сидней/5/2021)8.
Значение обнаруженных мутаций, приводящих к АК-заменам, оценивали по данным литературы, подтверждённым экспериментально. При этом нужно учитывать, что разные аминокислоты отличаются по физико-химическим свойствам (неполярные, полярные заряженные и незаряженные), они могут по-разному изменять пространственную конфигурацию белка, даже если замена произошла в том же положении АК-цепи. Функциональное значение каждой мутации должно быть доказано экспериментально. В ряде случаев допустимо проводить аналогию, например, между НА подтипов H1 и Н5, поскольку HA вирусов гриппа А H1 и H5 относятся к одной группе (H1/H2/H5/H6/H11/H13), которые, по предсказанной АК-последовательности НА, могут иметь схожие пространственные конформационные структуры НА [25].
В антигенном сайте Sa вируса гриппа A(H1N1)pdm09 были обнаружены мутации N156K и L161I, обе у 1 вируса. Мутация S159N в HA вируса гриппа А(H5N1) (положение эквивалентно N156K в исследованных A(H1N1)pdm09) приводит к усилению связывания с α-2,6-СК слизистой респираторного тракта хорьков [26]. Для мутации N156K в A(H1N1)pdm09 было предсказано изменение антигенных свойств в эксперименте на хорьках [27].
В антигенном сайте Sb вируса гриппа A(H1N1)pdm09 обнаружены мутации T185I, D187A, Q189E, S190N. В HA вируса гриппа A(H1N1) замена D187A (190 по нумерации H3) приводит к снижению связывания с α-2,6- и увеличению сродства к α-2,3-СК [28]. В HA эпидемического вируса гриппа A(H1N1) замена S190N (193 по нумерации H3) изменяет конформацию антигенного сайта Sb [29]. Рядом с антигенным сайтом Ca1 вируса A(H1N1)pdm09 обнаружена мутация R205K. В HA вируса H5N1 замена N224K (по нумерации H3) в эквивалентном положении приводит к усилению связывания с α-2,6-СК [30].
Рядом с антигенным сайтом Ca2 вируса A(H1N1)pdm09 обнаружена мутация R221K, входящая в состав рецептор-связывающего сайта HA A(H1N1)pdm09. Показано, что изменения этой аминокислоты могут влиять на антигенные свойства вирусов [31].
Аминокислота аргинин (R) в положении 223 усиливает сродство A(H1N1)pdm09 к рецепторам птичьего типа (α-2,3-СК). Учитывая тот факт, что α-2,3-СК входят в состав гликокаликса клеток эпителия, выстилающего альвеолы лёгких человека, такие мутантные штаммы могут вызывать пневмонию и далее генерализованный воспалительный процесс. К 2020 г. большинство циркулирующих вирусов гриппа A(H1N1)pdm09 (99,80%) имели мутацию R223Q, в том числе все секвенированные нами вирусы, тогда как на ранней стадии пандемии 2009 г. в составе второстепенной популяции ещё циркулировали штаммы, имеющие аргинин (R) в этом положении [32]. По-видимому, элиминация мутаций, которые могут снижать распространение вирусов в связи с высокой опасностью таких штаммов для жизни хозяина, является одним из механизмов эволюции вирусов гриппа.
Вне антигенных сайтов у 2 вирусов подтипа A(H1N1)pdm09 была обнаружена мутация D94N, у 1 — D94E. В HA вируса гриппа A(H5N1) замена D94N приводит к снижению связывания с α-2,6-СК и увеличению сродства к α-2,3-СК, а также усиливает HA-опосредованное слияние с мембраной клеток млекопитающих [33].
Мутация Е224А в рецептор-связывающем сайте A(H1N1)pdm09 увеличивает сродство к α-2,3-СК (рецепторам «птичьего» типа), локализованным в лёгких человека [34].
Мутация D222N в рецептор-связывающем сайте также усиливает связь с α-2,3-СК. С мутациями D222N и D222G связывают тяжёлое течение гриппа, включая пневмонию и острый респираторный дистресс-синдром [35]. В результате молекулярно-генетического анализа вирусов гриппа A(H1N1)pdm09, циркулировавших в России с 2009 по 2014 г., показано, что АК-замена D на G или N в положении 222 статистически значимо чаще выявлялась в лёгких умерших пациентов, нежели в респираторных мазках выздоровевших (p < 0,0001 и p = 0,007) [36]. В 2019–2020 гг. мутация D222N была обнаружена у 1 (2%) вируса из бронхоальвеолярного лаважа пациента с внебольничной пневмонией тяжёлой степени. В 2022–2023 гг. мутация D222N имелась у вирусов гриппа A(H1N1)pdm09, обнаруженных в образцах аутопсийного материала 12 пациентов (22% от количества вирусов из аутопсийного материала; 3% от общего количества).
Распределение вирусов A(H3N2) по генетическим группам соответствовало данным НИИ гриппа им. А.А. Смородинцева: в 2019–2020 гг. циркулировали в соотношении 2 : 1 вирусы генетической подгруппы 3С.2a1b + Т131К (референс-штамм A/Южная Австралия/34/2019) и 3С.2а1b + T135K-B (референс-штамм A/Гонконг/2675/2019)9; в 2021–2022 гг. превалировала генетическая группа 3C.2a1b.2а.210, а в 2022–2023 гг. — подгруппа 3C.2a1b.2a.2 (референс-вирус Bangladesh/ 4005/202011.
В сезоне 2020–2021 гг. в НИИ гриппа им. А.А. Смородинцева вирусы гриппа A(H3N2) не были выделены12, при этом исследованные нами вирусы относились к группе 3C.2a1b.2a.2, что соответствовало данным ECDC: в популяции вирусов гриппа А(H3N2) циркулировали вирусы группы 3C.2a1b, большинство из которых относились к Камбоджа- (3C.2a1b.2a.1) и Бангладеш-подобным (3C.2a1b.2a.2) вирусам13.
С 1968 по 2003 г. антигенный дрейф вируса гриппа A(H3N2) был вызван в основном одиночными мутациями в 7 АК-позициях в HA (145 — в сайте A, 155, 156, 158, 159, 189, 193 — в сайте B) рядом с рецептор-связывающим сайтом [37].
В HA вируса гриппа A(H3N2) замена A131D (в наших образцах — T131K) увеличивает заряд молекулы HA и приводит к снижению нейтрализации моноклональным антителом [38]. Показано, что мутации S193R и S193K (в наших образцах — S193F) влияют на предпочтительное связывание вируса с α-2,6- и α-2,3-СК соответственно [39].
В HA вируса гриппа свиней A(H3N2) замена S138A (аналогична обнаруженной в исследованных нами образцах) приводит к снижению репликации вирусов в эпителиальных клетках респираторного тракта свиней, в которых экспрессируются рецепторы α-2,6- и α-2,3-СК [40].
В HA вируса гриппа A(H3N2) замена K156Q (в наших образцах — H156S) приводит к снижению действия нейтрализующих антител. Это обусловлено тем, что остаток этой АК формирует глобулярную головку HA, где образует новый эпитоп, примыкающий к рецептор-связывающему домену [30].
Полученные нами данные распределения вирусов гриппа B по генетическим группам также соответствовали данным НИИ гриппа им. А.А. Смородинцева: в 2019–2020 гг. абсолютное большинство секвенированных вирусов гриппа типа В относилось к кладу V1А (del162–164) Викторианской линии (референс-вирус В/Вашингтон/02/2019)14; в сезоне 2021–2022 г. выявлены вирусы гриппа типа В/Виктория генетической подгруппы V1A.3a.215, в сезоне 2022–2023 гг. вирусы гриппа типа В были отнесены к генетической подгруппе V1A.3a.2 и подобны референс-вирусу B/Austria/1359417/2021.
У 40 вирусов выявлена мутация D197E в рецептор-связывающем сайте HA. Значимость АК-замен в этом положении НА была экспериментально доказана группой исследователей путём пассирования штамма B/Brisbane/60/2008 в линии эпителиальных клеток лёгких человека Calu-3: после 10 последовательных пассажей в НА появилась мутация D197T и было показано, что штаммы с этой заменой обладали значительно более низкой аффинностью к α-2,3-СК («птичьего» типа). Это можно объяснить тем, что α2,3-связанный гликан образует 2 водородные связи с аминокислотой в положении 197, и любая замена в этом положении может повлиять на связывание HA с рецепторами [41].
Заключение
В статье представлены результаты генетического мониторинга вирусов гриппа A(H1N1)pdm09, A(H3N2) и В, обнаруженных в 50 регионах России с 2019 по 2023 г. Полученные результаты отражают общие закономерности и позволяют судить об этиологической структуре гриппа, интенсивности эпидемического процесса и генетическом разнообразии циркулировавших в России вирусов.
В системе Роспотребнадзора мониторинг за вирусами гриппа проводится ежегодно, даже в сезоне 2020–2021 гг. на фоне пандемии COVID-19 скрининг больных на грипп методом ПЦР с гибридизационно-флуоресцентной детекцией продуктов амплификации, типирование и секвенирование обнаруженных вирусов гриппа продолжались в прежнем объёме. Именно поэтому можно констатировать, что в России на фоне пандемии COVID-19 в сезоне 2020–2021 гг. вирусы гриппа практически исчезли из циркуляции и появились снова в сезоне 2021–2022 года.
Выполненный анализ продемонстрировал явления непрерывной эволюции с появлением в каждом сезоне генетических вариантов вирусов гриппа A(H1N1)pdm09, A(H3N2) и В, которые имели изменения в гене HA по сравнению с вакцинным штаммом. Мутации, приводящие к АК-заменам, были зафиксированы в антигенных сайтах, в области связывания с рецепторами, некоторые из них приводили к образованию новых потенциальных сайтов гликозилирования или к их потере.
При сравнении вирусов гриппа A(H1N1)pdm09, циркулировавших в 2022–2023 гг. в России, с вакцинным штаммом 2019–2020 гг. A/Brisbane/02/2018 степень различий нуклеотидных последовательностей гена HА составила 2,7–3,1%, а с первым вакцинным штаммом вируса гриппа A(H1N1)pdm09 A/California/07/2009 — 5,0–5,3%.
Степень различий нуклеотидных последовательностей гена HА вирусов гриппа А(H3N2), циркулировавших в 2022–2023 гг. в России, с вакцинным штаммом 2019–2020 гг. A/Kansas/14/2017 составила 5,3–6,0%.
Степень различий нуклеотидных последовательностей гена HА вирусов гриппа B/Виктория, циркулировавших в 2022–2023 гг. в России, с вакцинным штаммом 2019–2020 гг. B/Colorado/06/2017 составила 2,2–2,8%. Вирусы гриппа B/Ямагата за время исследования не были идентифицированы.
Наибольшая вариабельность HA наблюдалась у вирусов А(H3N2), что вызвало необходимость замены вакцинного штамма 3 раза за 4 сезона.
Следует особо отметить, что все вирусы гриппа А(H1N1)pdm09 сезона 2022–2023 гг. имели не встречавшуюся ранее мутацию Е224А в HA, которая увеличивает сродство к α-2,3-СК, локализованным в лёгких человека, что может способствовать развитию осложнений. Мутация D222N, которая ассоциирована с более тяжёлым заболеванием, была обнаружена в HA 2% и 3% вирусов гриппа А(H1N1)pdm09 в 2019–2020 и 2022–2023 гг. соответственно.
В основном все вирусы гриппа были чувствительны к осельтамивиру и занамивиру, только у 2,3% вирусов гриппа A(H1N1)pdm09 в 2022–2023 гг. выявлена мутация устойчивости H275Y в NA. Во всех исследованных вирусах гриппа A(H1N1)pdm09 и A(H3N2) обнаружена мутация устойчивости к адамантанам S31N в M2.
Полученные нами результаты могут помочь понять направление эволюции вирусов гриппа. Непрерывное появление мутаций вирусов гриппа представляет глобальную проблему для здравоохранения из-за того, что некоторые мутации обеспечивают селективное преимущество для репликации вирусов в верхних дыхательных путях и передачи от человека к человеку, а также снижают чувствительность к противовирусным препаратам. Часть мутаций способствует более тяжёлому течению гриппа и развитию осложнений. Мутации в антигенных сайтах позволяют вирусам гриппа ускользать от анамнестических и поствакцинальных антител.
Следовательно, необходимо продолжать отслеживать вирусы гриппа с помощью молекулярно-генетического анализа, который позволяет проводить их глубокую дифференцировку и определять тренд эволюции вирусов гриппа: появление, распространение и исчезновение из циркуляции определённых генетических вариантов.
1 ВОЗ. Грипп (сезонный). URL: https://www.who.int/ru/news-room/fact-sheets/detail/influenza-(seasonal)
2 ECDC. Influenza virus characterisation, Summary Europe, July 2021. Stockholm;2021. URL: https://www.ecdc.europa.eu/en/publications-data/influenza-virus-characterisation-summary-europe-july-2021; ECDC. Influenza virus characterization: summary report, Europe, July 2022. Copenhagen;2022. URL: https://www.ecdc.europa.eu/en/publications-data/influenza-virus-characterization-summary-europe-july-2022
3 ECDC. Influenza virus characterisation, summary Europe, July 2020. Stockholm; 2020. URL: https://www.ecdc.europa.eu/en/publications-data/influenza-virus-characterisation-summary-europe-july-2020
4 ECDC. Influenza virus characterisation, Summary Europe, July 2021. Stockholm; 2021. URL: https://www.ecdc.europa.eu/en/publications-data/influenza-virus-characterisation-summary-europe-july-2021
5 НИИ гриппа имени А.А. Смородинцева. Еженедельный национальный бюллетень по гриппу и ОРВИ за 39-ю неделю 2023 года. URL: https://influenza.spb.ru/system/epidemic_situation/laboratory_diagnostics/?year=2023&week=39
6 ECDC. Influenza virus characterization: summary report, Europe, February 2023. Copenhagen–Stockholm;2023. URL: https://www.ecdc.europa.eu/en/publications-data/influenza-virus-characterization-summary-europe-february-2023
7 НИИ гриппа имени А.А. Смородинцева. Еженедельный национальный бюллетень по гриппу и ОРВИ за 39-ю неделю 2020 года. URL: https://www.influenza.spb.ru/system/epidemic_situation/laboratory_diagnostics/?year=2020&week=39
8 НИИ гриппа имени А.А. Смородинцева. Еженедельный национальный бюллетень по гриппу и ОРВИ за 39-ю неделю 2023 года. URL: https://influenza.spb.ru/system/epidemic_situation/laboratory_diagnostics/?year=2023&week=39
9 НИИ гриппа имени А.А. Смородинцева. Еженедельный национальный бюллетень по гриппу и ОРВИ за 39-ю неделю 2020 года. URL: https://www.influenza.spb.ru/system/epidemic_situation/laboratory_diagnostics/?year=2020&week=39
10 НИИ гриппа имени А.А. Смородинцева. Еженедельный национальный бюллетень по гриппу и ОРВИ за 24-ю неделю 2022 года. URL: https://www.influenza.spb.ru/system/epidemic_situation/laboratory_diagnostics/?year=2022&week=24
11 НИИ гриппа имени А.А. Смородинцева. Еженедельный национальный бюллетень по гриппу и ОРВИ за 39-ю неделю 2023 года. URL: https://influenza.spb.ru/system/epidemic_situation/laboratory_diagnostics/?year=2023&week=39
12 НИИ гриппа имени А.А. Смородинцева. Еженедельный национальный бюллетень по гриппу и ОРВИ за 39-ю неделю 2021 года. URL: https://www.influenza.spb.ru/system/epidemic_situation/laboratory_diagnostics/?year=2021&week=39
13 ECDC. Influenza virus characterisation, Summary Europe, July 2021. Stockholm; 2021. URL: https://www.ecdc.europa.eu/en/publications-data/influenza-virus-characterisation-summary-europe-july-2021
14 НИИ гриппа имени А.А. Смородинцева. Еженедельный национальный бюллетень по гриппу и ОРВИ за 39-ю неделю 2020 года. URL: https://www.influenza.spb.ru/system/epidemic_situation/laboratory_diagnostics/?year=2020&week=39
15 НИИ гриппа имени А.А. Смородинцева. Еженедельный национальный бюллетень по гриппу и ОРВИ за 24-ю неделю 2022 года. URL: https://www.influenza.spb.ru/system/epidemic_situation/laboratory_diagnostics/?year=2022&week=24
Об авторах
Светлана Борисовна Яцышина
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Автор, ответственный за переписку.
Email: svetlana.yatsyshina@pcr.ms
ORCID iD: 0000-0003-4737-941X
к. б. н., зав. лаб. молекулярной диагностики и эпидемиологии инфекций дыхательных путей
Россия, МоскваАнна Александровна Артамонова
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: svetlana.yatsyshina@pcr.ms
ORCID iD: 0009-0004-6845-9982
лаборант-исследователь лаб. молекулярной диагностики и эпидемиологии инфекций дыхательных путей
Россия, МоскваМария Александровна Елькина
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: svetlana.yatsyshina@pcr.ms
ORCID iD: 0000-0003-4769-6781
н. с. лаб. молекулярной диагностики и эпидемиологии инфекций дыхательных путей
Россия, МоскваАнна Владимировна Валдохина
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: svetlana.yatsyshina@pcr.ms
ORCID iD: 0000-0002-4592-4755
н. с. научной группы генной инженерии и биотехнологии
Россия, МоскваВиктория Петровна Буланенко
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: svetlana.yatsyshina@pcr.ms
ORCID iD: 0000-0001-7055-1762
н. с. научной группы генной инженерии и биотехнологии
Россия, МоскваАлександра Андреевна Берсенева
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: svetlana.yatsyshina@pcr.ms
ORCID iD: 0000-0002-1503-7629
лаборант-исследователь лаб. молекулярной диагностики и эпидемиологии инфекций дыхательных путей
Россия, МоскваВасилий Геннадьевич Акимкин
Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
Email: svetlana.yatsyshina@pcr.ms
ORCID iD: 0000-0003-4228-9044
д. м. н., профессор, академик РАН, директор
Россия, МоскваСписок литературы
- Hause B.M., Collin E.A., Runxia L., et al. Characterization of a novel influenza virus in cattle and swine: proposal for a new genus in the Orthomyxoviridae family. mBio. 2014;5(2):e00031–14. DOI: https://doi.org/10.1128/mBio.00031-14
- Houser K., Subbarao K. Influenza vaccines: challenges and solutions. Cell Host Microbe. 2015;17(3):295–300. DOI: https://doi.org/10.1016/j.chom.2015.02.012
- Osterhaus A.D., Rimmelzwaan G.F., Martina B.E., et al. Influenza B virus in seals. Science. 2000;288(5468):1051–3. DOI: https://doi.org/10.1126/science.288.5468.1051
- Webster R.G., Govorkova E.A. Continuing challenges in influenza. Ann. NY Acad. Sci. 2014;1323(1):115–39. DOI: https://doi.org/10.1111/nyas.12462
- Treanor J. Influenza vaccine — outmaneuvering antigenic shift and drift. N. Engl. J. Med. 2004;350(3):218–20. DOI: https://doi.org/10.1056/NEJMp038238
- Saunders-Hastings P.R., Krewski D. Reviewing the history of pandemic influenza: understanding patterns of emergence and transmission. Pathogens. 2016;5(4):66. DOI: https://doi.org/10.3390/pathogens5040066
- Chang W.K. National influenza experience in Hong Kong, 1968. Bull. World Health Organ. 1969;41(3):349–51.
- Rota P.A., Wallis T.R., Harmon M.W., et al. Cocirculation of two distinct evolutionary lineages of influenza type B virus since 1983. Virology. 1990;175(1):59–68. DOI: https://doi.org/10.1016/0042-6822(90)90186-u
- Vijaykrishna D., Holmes E.C., Joseph U., et al. The contrasting phylodynamics of human influenza B viruses. Elife. 2015;4:e05055. DOI: https://doi.org/10.7554/eLife.05055
- Thyagarajan B., Bloom J.D. The inherent mutational tolerance and antigenic evolvability of influenza hemagglutinin. Elife. 2014;3:e03300. DOI: https://doi.org/10.7554/eLife.03300
- Wei C.J., Boyington J.C., Dai K., et al. Cross-neutralization of 1918 and 2009 influenza viruses: role of glycans in viral evolution and vaccine design. Sci. Transl. Med. 2010;2(24):24ra21. DOI: https://doi.org/10.1126/scitranslmed.3000799
- Job E.R., Deng Y.M., Barfod K.K., et al. Addition of glycosylation to influenza A virus hemagglutinin modulates antibody-mediated recognition of H1N1 2009 pandemic viruses. J. Immunol. 2013;190(5):2169–77. DOI: https://doi.org/10.4049/jimmunol.1202433
- Sriwilaijaroen N., Suzuki Y. Molecular basis of the structure and function of H1 hemagglutinin of influenza virus. Proc. Jpn Acad. Ser. B Phys. Biol. Sci. 2012;88(6):226–49. DOI: https://doi.org/10.2183/pjab.88.226
- Glaser L., Stevens J., Zamarin D., et al. A single amino acid substitution in 1918 influenza virus hemagglutinin changes receptor binding specificity. J. Virol. 2005;79(17):11533–6. DOI: https://doi.org/10.1128/JVI.79.17.11533-11536.2005
- Virk R.K., Jayakumar J., Mendenhall I.H., et al. Divergent evolutionary trajectories of influenza B viruses underlie their contemporaneous epidemic activity. Proc. Natl Acad. Sci. USA. 2020;117(1):619–28. DOI: https://doi.org/10.1073/pnas.1916585116
- Cheng P.K., Leung T.W., Ho E.C., et al. Oseltamivir- and amantadine-resistant influenza viruses A (H1N1). Emerg. Infect. Dis. 2009;15(6):966–8. DOI: https://doi.org/10.3201/eid1506.081357
- Nobusawa E., Aoyama T., Kato H., et al. Comparison of complete amino acid sequences and receptor-binding properties among 13 serotypes of hemagglutinins of influenza A viruses. Virology. 1991;182(2):475–85. DOI: https://doi.org/10.1016/0042-6822(91)90588-3
- Соболев И.А., Курская О.Г., Шаршов К.А. и др. Изменчивость вируса гриппа типа А. Юг России: экология, развитие. 2016;11(1):170–7. Sobolev I.A., Kurskaya O.G., Sharshov K.A., et al. Variability of the influenza A virus. South of Russia: ecology, development. 2016;11(1):170–7. DOI: https://doi.org/10.18470/1992-1098-2016-1-170-177 EDN: https://elibrary.ru/vsasur
- Ndifon W., Wingreen N.S., Levin S.A. Differential neutralization efficiency of hemagglutinin epitopes, antibody interference, and the design of influenza vaccines. Proc. Natl Acad. Sci. USA. 2009;106(21):8701–6. DOI: https://doi.org/10.1073/pnas.0903427106
- Wang Q., Cheng F., Lu M., et al. Crystal structure of unliganded influenza B virus hemagglutinin. J. Virol. 2008;82(6):3011–20. DOI: https://doi.org/10.1128/JVI.02477-07
- Wang Q., Tian X., Chen X., et al. Structural basis for receptor specificity of influenza B virus hemagglutinin. Proc. Natl Acad. Sci. USA. 2007;104(43):16874–9. DOI: https://doi.org/10.1073/pnas.0708363104
- Еропкин М.Ю., Коновалова Н.И., Комиссаров А.Б. и др. Особенности эволюции вирусов гриппа, циркулировавших в России в течение 2-х сезонов, предшествовавших пандемии COVID-19, и в два пандемических сезона. В кн.: Дни вирусологии 2022: Сборник тезисов III Международного форума, посвященного 55-летию со дня основания ФГБУ «НИИ гриппа им. А.А. Смородинцева» Минздрава России. СПб.;2022. Eropkin M.Yu., Konovalova N.I., Komissarov A.B., et al. Features of the evolution of influenza viruses that circulated in Russia during the 2 seasons preceding the COVID-19 pandemic and in two pandemic seasons. In: Days of Virology 2022: Collection of abstracts of the III International Forum dedicated to the 55th anniversary of the founding of the Federal State Budgetary Institution "A.A. Smorodintsev Influenza Research Institute" of the Ministry of Health of Russia. St. Petersburg;2022. EDN: https://elibrary.ru/chdpfr
- Karpova L.S., Stolyarov K.A., Popovtseva N.M., et al. Comparison of the first three waves of the COVID-19 pandemic in Russia in 2020–21. Epidemiol. Vaccine Prev. 2022;21(2):4–16. DOI: https://doi.org/10.31631/2073-3046-2022-21-2-4-16
- Zhu N., Zhang D., Wanget W., et al. A novel coronavirus from patients with pneumonia in China, 2019. N. Engl. J. Med. 2020;382(8):727–33. DOI: https://doi.org/10.1056/NEJMoa2001017
- Ha Y., Stevens D.J., Skehel J.J., et al. H5 avian and H9 swine influenza virus haemagglutinin structures: possible origin of influenza subtypes. EMBO J. 2002;21(5):865–75. DOI: https://doi.org/10.1093/emboj/21.5.865
- Wang W., Lu B., Zhou H., et al. Glycosylation at 158N of the hemagglutinin protein and receptor binding specificity synergistically affect the antigenicity and immunogenicity of a live attenuated H5N1 A/Vietnam/1203/2004 vaccine virus in ferrets. J. Virol. 2010;84(13):6570–7. DOI: https://doi.org/10.1128/JVI.00221-10
- Guarnaccia T., Carolan L.A., Maurer-Stroh S., et al. Antigenic drift of the pandemic 2009 A(H1N1) influenza virus in a ferret model. PLoS Pathog. 2013;9(5):e1003354. DOI: https://doi.org/10.1371/journal.ppat.1003354
- Liang Y. Pathogenicity and virulence of influenza. Virulence. 2023;14(1):2223057. DOI: https://doi.org/10.1080/21505594.2023.2223057
- Yoshida R., Igarashi M., Ozaki H., et al. Cross-protective potential of a novel monoclonal antibody directed against antigenic site B of the hemagglutinin of influenza A viruses. PLoS Pathog. 2009;5(3):e1000350. DOI: https://doi.org/10.1371/journal.ppat.1000350
- Imai M., Watanabe T., Hatta M., et al. Experimental adaptation of an influenza H5 HA confers respiratory droplet transmission to a reassortant H5 HA/H1N1 virus in ferrets. Nature. 2012;486(7403):420–8. DOI: https://doi.org/10.1038/nature10831
- Zhao X.N., Zhang H.J., Li D., et al. Whole-genome sequencing reveals origin and evolution of influenza A(H1N1)pdm09 viruses in Lincang, China, from 2014 to 2018. PLoS One. 2020;15(6):e0234869. DOI: https://doi.org/10.1371/journal.pone.0234869
- Yasugi M., Nakamura S., Daidoji T., et al. Frequency of D222G and Q223R hemagglutinin mutants of pandemic (H1N1) 2009 influenza virus in Japan between 2009 and 2010. PLoS One. 2012;7(2):e30946. DOI: https://doi.org/10.1371/journal.pone.0030946
- Su Y., Yang H.Y., Zhang B.J., et al. Analysis of a point mutation in H5N1 avian influenza virus hemagglutinin in relation to virus entry into live mammalian cells. Arch. Virol. 2008;153(12):2253–61. DOI: https://doi.org/10.1007/s00705-008-0255-y
- Lakdawala S.S., Jayaraman A., Halpin R.A., et al. The soft palate is an important site of adaptation for transmissible influenza viruses. Nature. 2015;526(7571):122–5. DOI: https://doi.org/10.1038/nature15379
- Kilander A., Rykkvin R., Dudman S.G., et al. Observed association between the HA1 mutation D222G in the 2009 pandemic influenza A(H1N1) virus and severe clinical outcome, Norway 2009‐2010. Euro Surveill. 2010;15(9):19498. DOI: https://doi.org/10.2807/ese.15.09.19498-en
- Yatsyshina S., Renteeva A., Deviatkin A., et al. Molecular genetic analysis of the Influenza A(H1N1)pdm09 virus from lethal and recovered cases in Russia from 2009 to 2014: Deletions in the nucleoprotein. Infect. Genet. Evol. 2015;34:160–72. DOI: https://doi.org/10.1016/j.meegid.2015.07.019
- Koel B.F., Burke D.F., Bestebroer T.M., et al. Substitutions near the receptor binding site determine major antigenic change during influenza virus evolution. Science. 2013;342(6161):976–9. DOI: https://doi.org/10.1126/science.1244730
- Nakajima S., Nakajima K., Nobusawa E., et al. Comparison of epitope structures of H3HAs through protein modeling of influenza A virus hemagglutinin: mechanism for selection of antigenic variants in the presence of a monoclonal antibody. Microbiol. Immunol. 2007;51(12):1179–87. DOI: https://doi.org/10.1111/j.1348-0421.2007.tb04013.x
- Medeiros R., Naffakh N., Manuguerra J.C., et al. Binding of the hemagglutinin from human or equine influenza H3 viruses to the receptor is altered by substitutions at residue 193. Arch. Virol. 2004;149(8):1663–71. DOI: https://doi.org/10.1007/s00705-003-0287-2
- Busch M.G., Bateman A.C., Landolt G.A., et al. Identification of amino acids in the HA of H3 influenza viruses that determine infectivity levels in primary swine respiratory epithelial cells. Virus Res. 2008;133(2):269–79. DOI: https://doi.org/10.1016/j.virusres.2008.01.014
- Ilyushina N.A., Lee N., Lugovtsev V.Y., et al. Adaptation of influenza B virus by serial passage in human airway epithelial cells. Virology. 2020;549:68–76. DOI: https://doi.org/10.1016/j.virol.2020.08.004
Дополнительные файлы